文摘
使用可编程的核酸酶使同源重组基因工程(人力资源)介导的基因敲入。然而,工党用来构造目标向量含有同源臂和困难在一些细胞类型和生物诱导人力资源代表应用程序的技术障碍HR-mediated敲入技术。在这里,我们介绍一种策略使用转录基因敲入activator-like效应核酸酶(取得)和定期聚集空间短回文的重复序列(CRISPR) / CRISPR-associated 9 (Cas9)由microhomology-mediated end-joining,称为球场(精确融入目标染色体)系统。TALEN-mediated音高,称为TAL-PITCh,使有效整合外源供体DNA在人类细胞和动物,包括蚕和青蛙。我们进一步证明CRISPR / Cas9-mediated音高,称为CRIS-PITCh,可以应用在人类细胞中没有携带质粒骨架序列。因此,我们PITCh-ing策略将被用于各种各样的应用程序,不仅在培养细胞,而且在各种生物,包括无脊椎动物和脊椎动物。
介绍
可编程的核酸酶,如转录activator-like效应核酸酶(取得)和RNA-guided内切酶,也就是说,经常聚集空间短回文的重复序列(CRISPR) / CRISPR-associated 9 (Cas9),已被广泛用于基因工程,包括基因敲除,敲入和各种染色体重组1,2。基因敲入co-introduction普遍实现的可编程的单链寡核苷酸和核酸酶3,4或一个目标向量窝藏左右同源性武器5,6诱导同源重组(人力资源)端依赖基因之外。虽然HR-mediated基因敲入允许精确的大的DNA片段,插入目标向量的建设往往是费力和定位效率取决于人力资源的实质性的变化频率感应细胞类型和生物物种。然而,悬臂的互补供体DNA片段或简单的线性化供体DNA质粒可以促进目标集成由异源end-joining在培养细胞7,8在斑马鱼9。据说专ligation-gated重组也能质粒DNA整合到一个目标基因位点10。这些方法利用可编程的核酸酶DNA双链断裂(双边带),5′悬臂(锌指核酸酶(ZFNs)和取得)或冲(CRISPR / Cas9)结束,然后依靠结扎相似的染色体目标站点和插入结束。因此,可以认为这些目标集成代表简单结扎。
相反,microhomology-mediated end-joining (MMEJ)端依赖突变经常被发现在可编程nuclease-mediated基因中断没有外生捐助者11。MMEJ是一个双边带修复机制,使用microhomologous序列(bp)做些容易出错的end-joining12。在细胞周期中,MMEJ修复活跃在G1 / S早期阶段,而人力资源是活跃在S / G2后期阶段13。因此,我们设计了一个新颖的MMEJ-mediated基因敲入策略,称为沥青(精确融入目标染色体)系统,使有效的有针对性的一体化的大型DNA片段在一个广泛的细胞和生物,即使是那些较低的人力资源活动。我们证明外源性报告基因的插入到人体细胞和动物使用沥青系统取得和CRISPR / Cas9。我们方法提供新的见解目标插入的外源供体DNA和另一种方式使把细胞和生物体。
结果
在人类细胞TAL-PITCh设计和应用
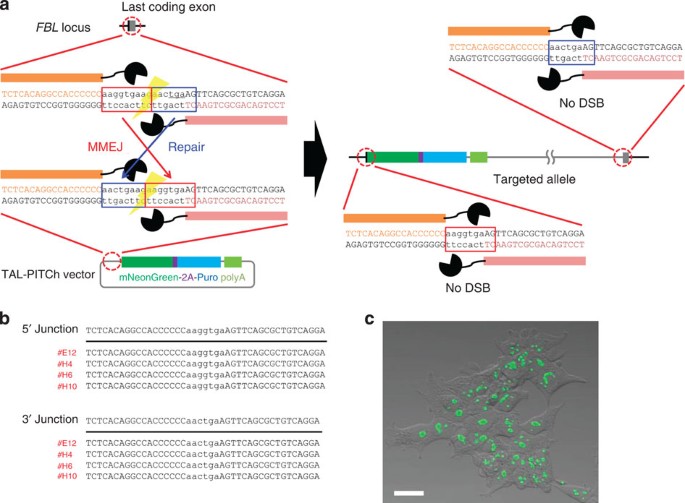
我们首先证明了音高体系TALEN-mediated敲入(TAL-PITCh)。在TAL-PITCh,一双单取得和TAL-PITCh向量包含取得目标站点构造和co-introduced (图1一个左面板)。生成microhomologous序列,取得目标站点TAL-PITCh向量应该包含一个不同的间隔序列与原始基因组序列相比,前,后一半一半的交换。基因组序列和TAL-PITCh向量可以减少相同的取得,和线性化TAL-PITCh向量包含microhomologous DNA基因组裂解位点对应结束。MMEJ-dependent集成后,整个矢量正是整合到基因组有两个取得目标网站(图1一个右面板)。然而,这些并不减少取得取得目标网站,因为它们含有缩短间隔区域,这是出于对双边带的最优范围介绍取得14。
作为一个概念验证实验中,我们首先证明了在培养细胞TAL-PITCh系统。我们有针对性的最后编码外显子的人类fibrillarin(FBL使用白金取得)基因15,把TAL-PITCh向量HEK293T细胞,导致c端融合mNeonGreen最近报道说,作为一个超亮的荧光蛋白基因16,紧随其后的是2 a-puromycin。TAL-PITCh向量不含哺乳动物细胞表达的启动子;因此,FBL-mNeonGreen-2A-puromycin应该由内生基因表达FBL启动子。嘌呤霉素选择后,单个细胞被限制稀释和培养独立隔离。六个潜在撞倒克隆被DNA测序和激光扫描荧光显微镜分析。基因组区域的5′和3′连接可以通过PCR和测序被放大的四个六个克隆(补充图1 a, b;补充表1)。所有的克隆测序正确目标等位基因由MMEJ (图1 b),显示核仁的荧光,与前一个报告是一致的16(图1 c)。
测试的适用性TAL-PITCh用于另一个基因位点,另一个细胞系,我们有针对性的人类β肌动蛋白(ACTB)基因在海拉细胞(补充图2)。六可能撞倒细胞克隆显示荧光和他们建立了连接被PCR分析。四个六被选为正确定位候选克隆(补充图3 a, b;补充表1)。在这种情况下,一个包含四个克隆3-bp插入5′结和三个克隆中含有5-27-bp插入和删除的3′结;一个克隆在双方正确的连接(补充图2 b)。应力纤维的荧光观察这个克隆(补充图2摄氏度)。我们进一步证实了正确集成通过印迹分析,表明没有随机整合发生(补充图4)。我们也证实了克隆形成高于TALEN-assisted基因敲入由人力资源,建议MMEJ-mediated集成与传统方法相比的优越性(补充图5)。
TAL-PITCh动物
检查TAL-PITCh系统的适用性在活的有机体内,我们下一个检查TAL-PITCh蚕(家蚕和青蛙非洲爪蟾蜍光滑的)。在蚕,取得可以诱导高效目标基因的突变和变异率G0配子可以超过50%17。然而,一个成功的长基因敲入盒使用取得尚未实现。这大概是因为人力资源活动很低蚕的生殖细胞17并建议常规敲入方法由人力资源也不乐观。因此,我们设想的想法应用MMEJ-mediated TAL-PITCh系统在蚕。
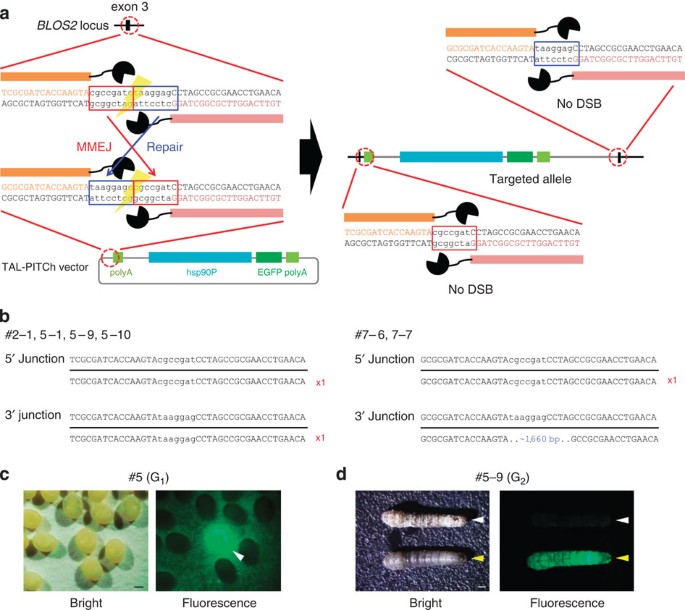
我们有针对性的蚕BLOS2基因,因为它的高效淘汰赛之前使用取得成就18。信使核糖核酸(mRNA)取得对第三外显子的设计BLOS2基因注入TAL-PITCh向量窝藏在一起一半promoter-enhanced绿色荧光蛋白(EGFP)表达盒19(图2一个),EGFP表达他们的后代被检查。值得注意的是,G1胚胎显示强劲EGFP表达(图2 c;补充图6;补充表2)。这个结果表明,敲入成功发生在G0配子。我们检查每个EGFP-positive G的基因型1个人和发现TAL-PITCh向量集成到BLOS2轨迹在六个蠕虫(图2 b;补充图7;补充表2)。其中四个显示精确的集成,而两人~ 1660 - bp额外的序列包含一个部分EGFP序列和基因序列的下游~ 2.6 kb取得目标站点的3′端(图2 b;补充表2)。目标融入BLOS2轨迹进一步支持的事实,这些人表现出一种油性皮肤,破坏造成的表型BLOS2基因18(图2 d)。因此,我们得出结论,在蚕TAL-PITCh系统非常有效。
(一个)TAL-PITCh的图解说明b . mori BLOS2轨迹。橙色和粉色的字母显示左右取得目标网站,分别。红色和蓝色框表示microhomologous序列。hsp90P,一半启动子。(b从6 G)撞倒等位基因序列1蠕虫(# 2、# 5 - 1 # 5 - 9,# 5 - 10,# 7和# 7)。打算把序列显示在顶部。取得目标站点大写字母所示。红色字母显示正确撞倒等位基因。蓝色的字母表示插入。(c)具有亮和荧光显微镜的图像G1胚胎在# 5批。强大的EGFP的表达可以观察到在假定的敲入胚胎,根据的活动一半启动子(箭头)。比例尺,0.5毫米。(d)具有亮和荧光显微镜的图像G2幼虫来自# 5 - 9批。白色和黄色箭头表明野生型和敲入蚕,分别。注意,敲入幼虫显示了油性皮肤表型。规模的酒吧,1毫米。
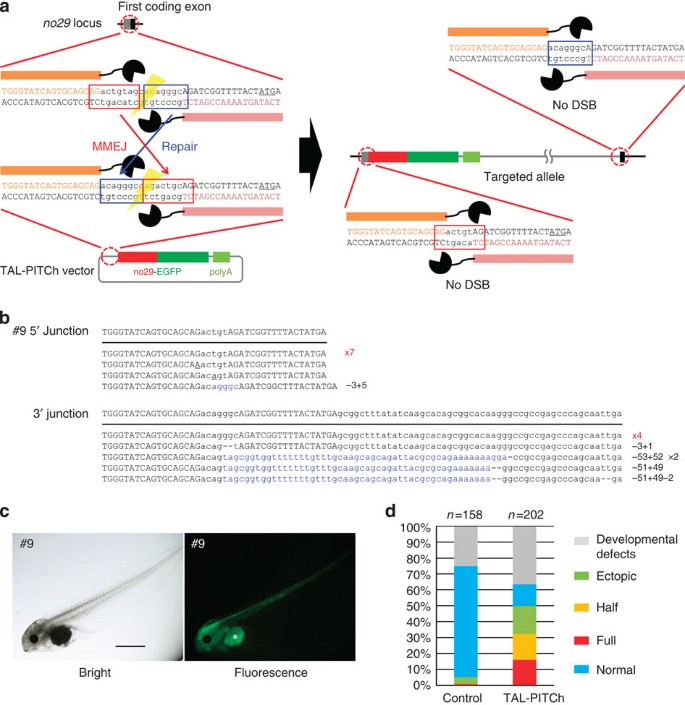
我们随后尝试EGFP敲入内源性基因位点x光滑的脊椎动物的胚胎作为一个模型,因为在青蛙包括基因敲入x光滑的尚未实现,尽管有针对性的突变可以有效地执行使用取得20.,21。因此,我们第一个目标no29轨迹,组蛋白伴侣paralogues之一22在x光滑的使用TAL-PITCh (图3)。在这种情况下,我们设计取得的起始密码子no29基因和打no29- - - - - -EGFP融合互补DNA。尽管的空间表达模式no29在早期的发展x光滑的从未阐明,我们观察到一个明显的表达趋势在中枢神经系统(图3 c;补充图8 b)。总的来说,~ 15%的胚胎注射取得mrna和TAL-PITCh向量显示充分表达,另一个15%的胚胎表达式显示一半,也就是说,左边一半或右半边的身体,在中枢神经系统(图3 d)。三人预期大小的扩增子的5′和3′连接测序,和所有三个精确定位等位基因至少在5′结。3′结,然而,并不是所有的个体包含精确定位等位基因(# 1、9和10;图3 b;补充图8 a, b)。
(一个)TAL-PITCh的图解说明x光滑的no29轨迹。橙色和粉色的字母显示左右取得目标网站,分别。红色和蓝色框表示microhomologous序列。起始密码子展露无遗。(b从胚胎# 9)撞倒等位基因序列。打算把序列显示在顶部。取得目标站点大写字母所示。红色字母显示正确撞倒等位基因。蓝色的字母表示插入。破折号表示删除。替换下划线。(c)和荧光显微镜图像具有亮胚胎# 9。星号表明蛋黄自体荧光。规模的酒吧,1毫米。(d)的表型比例控制胚胎和TAL-PITChed胚胎。的矢量控制,取得目标站点是被用来代替TAL-PITCh向量。除了异常发达的胚胎,表型被分成四组(满,一半,异位和正常)根据EGFP的表达区域。总数量的个人顶部的每个图所示。
我们下一个演示了TAL-PITCh-mediated在活的有机体内基因敲入的角蛋白(fgk;鳍和鳃角蛋白)轨迹x光滑的,在某种程度上类似于人类细胞(图4)。的EGFP基因插入前内源性表达一个终止密码子fgk- - - - - -EGFP融合基因。关于fgk,转基因x光滑的胚胎有报道显示具体表达式鳍和鳃23。符合这个报告,我们获得了一些胚胎显示荧光专门本地化的鳍(图4 b)和吉尔(图4摄氏度)精确的5′和3′路口,尽管其中一个也包含微妙的突变在连接(图4 b, c)。此外,熔化的表情使我们观察细胞骨架定位(图4 b, c)。
在人类细胞CRIS-PITCh设计和应用
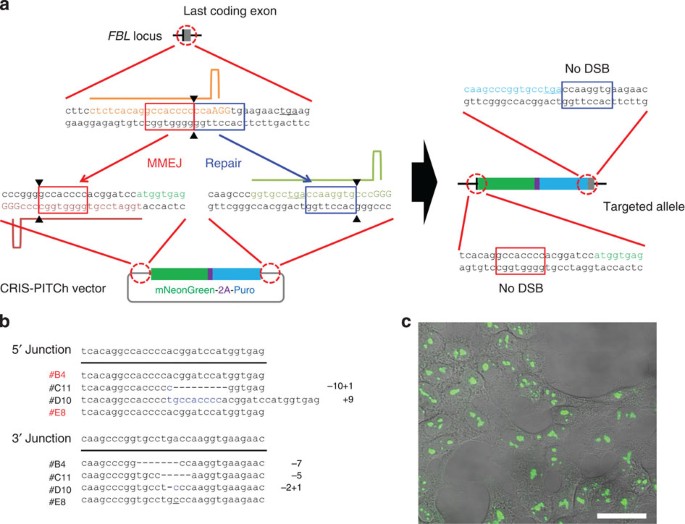
音调系统的另一个重要方面是CRISPR / Cas9可以使用,而不是取得。因此,我们的目标FBL轨迹HEK293T细胞使用CRISPR / Cas9-mediated音高(CRIS-PITCh) (图5左面板)。诱导双边带的原则取得和CRISPR / Cas9完全不同;因此,我们修改了针对战略(图5)。在CRIS-PITCh,三个指南rna (gRNAs)和Cas9核酸酶应该coexpressed,和两个不同的gRNA目标站点应该添加到CRIS-PITCh向量。策略用于TAL-PITCh可以,当然,也适用于CRIS-PITCh;然而,有几个原因改善系统:删除不必要的向量骨干和废除限制gRNA目标序列(有关详细信息,请参阅讨论)。使用这个改进CRIS-PITCh系统启用磁带集成,我们可以产生把细胞没有任何额外的序列(图5右面板)。转染后CRISPR / Cas9和CRIS-PITCh向量,其次是嘌呤霉素选择和单细胞克隆、基因组DNA提取和撞倒等位基因被PCR(放大补充图9)。DNA测序显示的两个四个测序克隆包含精确加入5′连接,虽然他们已经替换,插入或删除3′端(图5 b)。核仁的本地化的荧光观察,类似于TAL-PITCh实验(图5度)。
最后,我们调查是否TAL-PITChed CRIS-PITChed细胞克隆包含非目标突变,特别是因为CRISPR / Cas9系统据报道能导致大量非目标突变在人类细胞系,如U2OS、K562和HEK293细胞24,25,26。的六大潜在的非目标网站测序TAL-PITChed HEK293T细胞克隆(# H4和#代替图1 b和补充图1),前三个潜在的非目标网站的每个gRNA测序CRIS-PITChed HEK293T细胞克隆(# B4和# E8图5 b和补充图9 b)。我们的救援,测序脱靶候选人中没有一个是突变(补充表3和图4)。
讨论
随着HR-independent敲入策略报告到目前为止,我们的投球策略可以灵活的在细胞和动物基因敲入。在这项研究中,我们检查了使用CRIS-PITCh full-plasmid集成使用TAL-PITCh和盒式集成。然而,盒式集成使用TAL-PITCh没有矢量骨干和full-plasmid集成使用CRIS-PITCh也是可能的。当执行使用TAL-PITCh盒式集成,取得两个目标站点应该添加盒两端(补充图10)。左边一半的间隔序列的基因组取得目标站点应放置在适当的间隔的一半地区的左翼取得网站TAL-PITCh向量,和一半的间隔序列的基因组取得目标站点应该放在左边一半的间隔区域右边取得网站的向量。另外,额外Cre-loxP——或者Flp-FRT-mediated切除后建立撞倒细胞或minicircle DNA的一代27构建质粒载体也可以执行后,在前面的讨论研究10。
有两种方法可以执行使用CRIS-PITCh full-plasmid集成,和限制目标基因组序列的变化。当gRNA CRIS-PITCh向量有义链设计,顺序限制是5′-GGNNNNNNGG-3′,如果microhomologous序列设置为8英国石油公司(补充图11)。然而,当gRNA CRIS-PITCh向量对反义链设计,顺序限制是5′-CCGG-3′,如果microhomologous序列设置为8英国石油公司(补充图11 b)。修改microhomology序列的长度可以改变这些限制的目标序列。
其他技巧TAL-PITCh和CRIS-PITCh如下所述。关于TAL-PITCh,左右取得目标网站TAL-PITCh可以打乱。如果他们被打乱,有针对性的等位基因的5′端将包含左取得网站,缩短间隔和倒置的取得。目标等位基因的3′端将包含正确的网站取得,缩短间隔和反向权利取得的网站。通过执行洗牌,我们可以完全避免再切取得目标等位基因,如果我们使用取得包含heterodimeric FokI核酸酶域28,29日,30.。关于CRIS-PITCh-mediated盒式集成,左右gRNA目标网站CRIS-PITCh向量应该为了目标反义和链,分别如图所示图5。否则,其他序列的限制比原protospacer相邻基因CRISPR / Cas9目标站点的主题将出现。当目标基因的反义链CRISPR / Cas9目标站点,左右gRNA目标网站CRIS-PITCh向量应该设计目标感和反义链,分别以最小化的局限性基因组CRISPR / Cas9目标站点。
TAL-PITCh和CRIS-PITCh实验,5′路口有一个高的趋势是使用microhomologous序列加入准确;然而,3′路口被MMEJ不一定加入。这种趋势可能会依赖于选择方法,我们采用了在这个研究。嘌呤霉素选择用于这项研究可以排除在图片克隆;然而,3′结被认为没有什么影响的选择。的情况下都正确连接需要加入药物或荧光选择,很可能观察到的偏见在这项研究将会消失。此外,增强MMEJ修复途径和/或抑制NHEJ修复机械也可能增加目标与精确的连接和集成减少NHEJ-dependent错误的集成,包括假阳性克隆,如# F3和# B10补充图1 b,# 2和# 6补充图3 b和# G10补充图9,据说有意外撞倒等位基因;例如,集成concatemerized向量,与大删除或添加和随机整合集成。还需要进一步的研究来阐明整合的机制,提高其准确性。
总的来说,我们证明TAL-PITCh-mediated基因敲入可以应用在人类细胞和其他动物,建议策略的广泛适用性。我们所知,这是第一个报告显示有针对性的插入可以通过短microhomologies发生,在培养细胞和动物。此外,我们证明了成功CRIS-PITCh-mediated在人类细胞没有携带基因敲入一个向量序列支柱。我们预料我们系统将提高基因组工程技术的作用在各种细胞和生物体,特别是那些在基因敲入是困难的,因为较低的人力资源效率。
方法
建设取得质粒
人类细胞和青蛙的实验中,一个两步金门组装方法使用铂门取得工具包(Addgene;猫# 1000000043)15是用于构造铂取得质粒含有homodimer-type FokI核酸酶域。短暂、单端dna重复被组装成中间数组向量。组装重复数组随后被插入到最终目的地向量,ptCMV-153/47-VR。蚕的实验,金门取得和TAL效应工具包2.0 (Addgene;猫# 1000000024)31日被用来构造取得(BLTS-5A BLTS-4B18),和重复数组插入支架质粒,pBlue-TAL18。
建设CRISPR / Cas9质粒
影城CRISPR / Cas9装配系统32被用来构建一体化CRISPR / Cas9质粒。简而言之,pX330向量(Addgene;质粒42230)是修改统一多个gRNA-expressing磁带到单个向量使用金门装配方法。gRNA模板合成寡核苷酸,退火并插入相应的向量。的列表所示使用的寡核苷酸补充表5。金门大会被用来组装构造向量成一个一体化的CRISPR / Cas9向量FBL基因,称为pX330A -FBL3 grnas窝藏三个gRNA磁带和一个Cas9录音带。
球场的建设和人力资源向量
TAL-PITCh, CRIS-PITCh和人力资源向量构造使用PCR和在融合克隆(Clontech)或标准的分子克隆方法。full-plasmid序列所示补充图12。
细胞培养和转染
从写明ATCC HEK293T和海拉细胞,维持在杜尔贝科修改鹰与10%胎牛血清的培养基补充。Lipofectamine第(生命技术)和Opti-MEM(技术)被用来使转染质粒,根据供应商的协议。质粒浓度,细胞数量和菜肴使用如下:200 ng ptCMV左右取得向量和TAL-PITCh向量1×105细胞实验使用six-well板图1和补充图2;1.6μg ptCMV左右取得向量和TAL-PITCh或人力资源向量为5×105细胞使用100毫米盘的实验补充图5;400 ng pX330A -FBL3 grnas CRISPR / Cas9向量和200 ng CRIS-PITCh向量1×105使用six-well板实验的细胞图5。转染后细胞在上述生长培养基培养3天,然后选择1μg毫升−1嘌呤霉素为6天。DNA测序,显微镜和南部印迹,选定的细胞克隆的使用限制在96孔板稀释法。
信使rna的合成和显微镜下注射蚕实验
mMessage 1 T7超工具包(技术)被用来合成b . mori BLOS2取得mRNA (BLTS-5A和BLTS-4B18)。信使rna与氯化锂沉淀,用70%的乙醇清洗三次,风干和溶解在0.5毫米磷酸盐缓冲剂(pH值7.0)包含5毫米氯化钾。取得mRNA (250 ngμl−1每个μl)和500 ng−1捐赠者向量是蚕的注入胚胎w1- - - - - -患产后抑郁症应变之间收集1 -和2 h后产卵在合胞体preblastderm阶段。注射后,打开密封胶和胚胎孵化25°C。孵化蚕幼虫饲养在一个人造的饮食(日本Nosan Kogyo) 25°C下12 h光明/黑暗光周期。每个注入个人交叉与身体蠕虫。EGFP的表达G1个人观察在胚胎阶段,EGFP-positive胚胎选择进行进一步的饲养。G1成年人是交叉的含水量滞育的压力。
信使rna的合成和显微镜下注射青蛙实验
受精x光滑的鸡蛋是来自野生型成人注射人体绒毛膜促性腺激素(日本人名制药)。鸡蛋dejellied有2%半胱氨酸和清洗在0.1×Marc的铃声(MMR)修改。洗蛋转移到5%聚蔗糖(Sigma-Aldrich) 0.3×MMR co-injected有一双取得mrna (250 pg),合成使用mMessage 1 T7超设备(技术),和TAL-PITCh向量(100 pg)在一个细胞阶段使用Nanoject II(德拉蒙德)。注射胚胎饲养游泳舞台0.1×MMR 20°C。动物是维护和使用按照广岛大学实验动物的使用和护理指南。
显微镜
对人类细胞实验,细胞被转移到collagen-coated杯底24-well板块在PBS和4%多聚甲醛固定。荧光观察和细胞图像捕获使用488 nm激光共焦激光扫描显微镜(奥林巴斯阵线- 1000 d)。为b .森使用荧光实验,观察荧光立体显微镜(奥林巴斯SZX16)。为x光滑的使用荧光实验,观察荧光立体显微镜(徕卡MZ10F)。
基因组PCR和DNA测序
DNeasy血液和组织工具包(试剂盒)用于从细胞中提取基因组DNA丸,青蛙胚胎和蚕幼虫或成人。基因组PCR进行外汇(Toyobo),使用KOD KOD FX Neo (Toyobo)或洛杉矶Taq(豆类)中列出的引物补充表6。人类细胞和蚕实验中,PCR产品受到直接的DNA测序。为x光滑的实验中,PCR克隆产品,转化为细菌使用威尼斯平底渔船TA克隆工具与PCR2.1威尼斯平底渔船(技术)。随后,菌落PCR产品用作DNA测序模板。DNA测序进行使用ABI 3130 xl基因分析仪(技术)BigDye终结者v3.1循环测序工具(技术)。
非目标分析
PROGNOS工具(http://baolab.bme.gatech.edu/cgi-bin/prognos/prognos.cgi)33被用来识别潜在的非目标网站的FBL取得。最大不匹配/ half-site和间隔的长度设置为6和12 - 24,分别。CRISPR设计工具(http://crispr.mit.edu/)25被用来识别潜在的非目标网站的三个gRNAs基因位点和CRIS-PITCh向量。每个候选基因组区域网站中列出被使用引物PCR扩增补充表7直接测序和序列证实了。
印迹分析
南部印迹是根据之前的报告34有一些修改,如下所述。与PstI Five-μg整除的基因组DNA被消化,和2μg和1μg外和西班牙芒果探针,分别解决在0.8%琼脂糖凝胶。Digoxigenin-labelled DNA探针是由PCR使用KOD FX Neo (Toyobo)和挖掘DNA标签(罗氏)混合引物中列出补充表6。膜转移(Hybond-N +;通用电气医疗集团),紫外线交联(120 mJ厘米−2),pre-hybridization和杂交根据指令进行挖掘简单Hyb颗粒(罗氏)。CDP-Star检测试剂(罗氏)是用于开发的膜,在制造商的指示。化学发光信号检测使用Amersham Hyperfilm发射极耦合逻辑(通用电气医疗集团)。
引用
卡罗尔,d基因组工程与通道核酸酶。为基础。学生物化学启。83年,409 - 439 (2014)。
金,h &金,j . s .指南与可编程的核酸酶基因工程。Nat,启麝猫。15,321 - 334 (2014)。
陈,f . et al .高频基因组编辑使用ssDNA寡核苷酸与锌指核酸酶。Nat方法。8,753 - 755 (2011)。
Miyaoka, y . et al .隔离的单碱基genome-edited人类“诱导多能性”细胞没有抗生素的选择。Nat方法。11,291 - 293 (2014)。
Hockemeyer, d . et al。有效的表达和沉默目标使用锌指核酸酶基因在人类的ESCs和万能。生物科技Nat。》。27,851 - 857 (2009)。
Hockemeyer, d . et al .基因工程对人类多能细胞核酸酶使用故事。生物科技Nat。》。29日,731 - 734 (2011)。
美国奥兰多j . et al .锌指nuclease-driven有限的目标是使用捐赠者的集成到哺乳动物的基因组染色体同源性。核酸Res。38e152 (2010)。
Cristea, s . et al .体内转基因捐助者促进nuclease-mediated目标集成的乳沟。Biotechnol。Bioeng。110年,871 - 880 (2013)。
奥氏小体,t . O。,Duroure, K., De Cian, A., Concordet, J. P. & Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair.基因组Res。24,142 - 153 (2014)。
马雷斯卡、M。,Lin, V. G., Guo, N. & Yang, Y. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining.基因组Res。23,539 - 546 (2013)。
Bae, S。,Kweon, J., Kim, H. S. & Kim, J. S. Microhomology-based choice of Cas9 nuclease target sites.Nat方法。11,705 - 706 (2014)。
美国借债过度,m & Lee大肠MMEJ修复(导演剪辑版)的双链断裂:删除序列和替代的结局。趋势麝猫。24,529 - 538 (2008)。
Taleei, r & Nikjoo h .生化DSB-repair模式哺乳动物细胞在细胞周期G1和年代早期阶段。Mutat。Res。756年,206 - 212 (2013)。
米勒,j . c . et al .核酸酶故事架构高效基因组编辑。生物科技Nat。》。29日,143 - 148 (2011)。
佐久间,t . et al .重复的模式non-RVD dna结合模块提高取得活动的变化。科学。代表。33379 (2013)。
沙:c . et al,明亮的绿色荧光蛋白单体的来自文昌鱼lanceolatum。Nat方法。10,407 - 409 (2013)。
邪神,T。,Kiuchi, T. & Takasu, Y. Recent progress in genome engineering techniques in the silkworm,家蚕。Dev。增长是不同的。5614-25 (2014)。
Takasu, y . et al。有效的取得建设家蚕基因打靶。《公共科学图书馆•综合》8e73458 (2013)。
Tsubota, t . et al .小说强烈的识别和无处不在的启动子/增强蚕,家蚕。G34,1347 - 1357 (2014)。
铃木k . t . et al .高效率取得使F0功能分析,有针对性的基因中断非洲爪蟾蜍光滑的胚胎。医学杂志。开放2,448 - 452 (2013)。
Sakane, y等。针对多个和paralogous基因的突变非洲爪蟾蜍光滑的使用两对转录activator-like效应核酸酶。Dev。增长是不同的。56,108 - 114 (2014)。
Motoi, N。,Hasebe, T., Suzuki, K. T. & Ishizuya-Oka, A. Spatiotemporal expression profile of no29/nucleophosmin3 in the intestine of非洲爪蟾蜍光滑的在蜕变。细胞组织Res。344年,445 - 453 (2011)。
铃木k . t . et al .小说类型的描述我角蛋白基因和代转基因线荧光报告基因由其启动子/增强剂非洲爪蟾蜍光滑的。Dev。直流发电机239年,3172 - 3181 (2010)。
傅,y . et al。高频脱靶诱变引起CRISPR-Cas核酸酶在人类细胞。生物科技Nat。》。31日,822 - 826 (2013)。
许,p . d . et al。DNA靶向特异性RNA-guided Cas9核酸酶。生物科技Nat。》。31日,827 - 832 (2013)。
Cradick, t·J。,好吧,e . J。,Antico, C. J. & Bao, G. CRISPR/Cas9 systems targeting β-globin andCCR5基因大量非目标的活动。核酸Res。41,9584 - 9592 (2013)。
凯,m·A。,He, C. Y. & Chen, Z. Y. A robust system for production of minicircle DNA vectors.生物科技Nat。》。28,1287 - 1289 (2010)。
米勒,j . c . et al .改善锌指核酸酶结构高度特定基因组编辑。生物科技Nat。》。25,778 - 785 (2007)。
Szczepek, m . et al。基于结构的二聚作用界面的设计降低了锌指核酸酶的毒性。生物科技Nat。》。25,786 - 793 (2007)。
Doyon, y . et al。加强与改善zinc-finger-nuclease活动预留heterodimeric架构。Nat方法。8,74 - 79 (2011)。
Cermak, t . et al。高效的设计和组装定制的取得和其他TAL effector-based构造DNA的目标。核酸Res。39e82 (2011)。
佐久间,T。,Nishikawa, A., Kume, S., Chayama, K. & Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system.科学。代表。45400 (2014)。
很好,e . J。,Cradick, t·J。赵,c . L。,Lin, Y. & Bao, G. An online bioinformatics tool predicts zinc finger and TALE nuclease off-target cleavage.核酸Res。42e42 (2014)。
落,h . et al . TALEN-mediated单个碱基对编辑的上游基因间的变异的识别BUB1B个人电脑(MVA)综合症的病因。Proc。《科学。美国111年,1461 - 1466 (2014)。
确认
我们感谢博士Hiroshi落(广岛大学、广岛、日本)共享合成mNeonGreen cDNA、博士Keiro Uchino(国家农业科学研究所、茨城、日本)注入蚕,洋子博士Takasu(国家农业科学研究所、茨城、日本)对取得信使rna合成的教诲,Kaoru中村先生和行长三泽先生(国家农业科学研究所、茨城、日本)饲养蚕和聪女士川(国家农业科学研究所、茨城、日本)的技术援助。这项工作是支持的日本促进社会科学(K.T.S. 25890014 t。s。, 25890014),农业部、林业和渔业,尼亚斯战略研究基金会。
作者信息
作者和联系
贡献
电汇,T.S. and K.T.S. designed the work. S.N., T.T., Y.S., S.K., T.S. and K.T.S. performed the experiments. T.T., T.S. and K.T.S. wrote the manuscript with support from all the authors. N.S., M.O., T.D. and H.S. provided instructions. T.Y. supervised the work.
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
补充信息
补充信息
补充图1 - 12、补充表1 - 7。(PDF 6304 kb)
权利和权限
这项工作是基于知识共享署名4.0国际许可证。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在信贷额度;如果材料不包括在Creative Commons许可下,用户需要获得许可证持有人的许可复制的材料。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/
关于这篇文章
引用这篇文章
Nakade, S。,Tsubota, T., Sakane, Y.et al。Microhomology-mediated end-joining-dependent供体DNA在细胞和动物使用的集成取得和CRISPR / Cas9。Nat Commun55560 (2014)。https://doi.org/10.1038/ncomms6560
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/ncomms6560
本文引用的
软件:一个高效的基因整合方法供MMEJ抑制啮齿动物和灵长目动物胚胎
基因组生物学(2023)
在治疗CRISPR-Cas9基因组编辑最新进展:机制和应用程序
分子生物医学(2023)
基因编辑“铅笔”而不是“剪刀”人类的多能干细胞
干细胞研究与治疗(2023)
有针对性的使用集成在人类细胞DNA双链断裂CRISPR-associated转座酶
自然生物技术(2023)
针对DNA修复途径B02和诺考达唑小分子来改善CRIS-PITCh盒式集成CHO-K1细胞介导的
科学报告(2023)