亲爱的编辑,gydF4y2Ba
COVID-19大流行已经持续了近两年半,新的SARS-CoV-2关注变种(VOCs)不断出现,这推动了广泛中和抗体的开发。gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba据报道,delta (B.1.617.2谱系)和Omicron (BA.1和BA.2)等变体对目前的一些治疗性抗体表现出免疫逃避。gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba
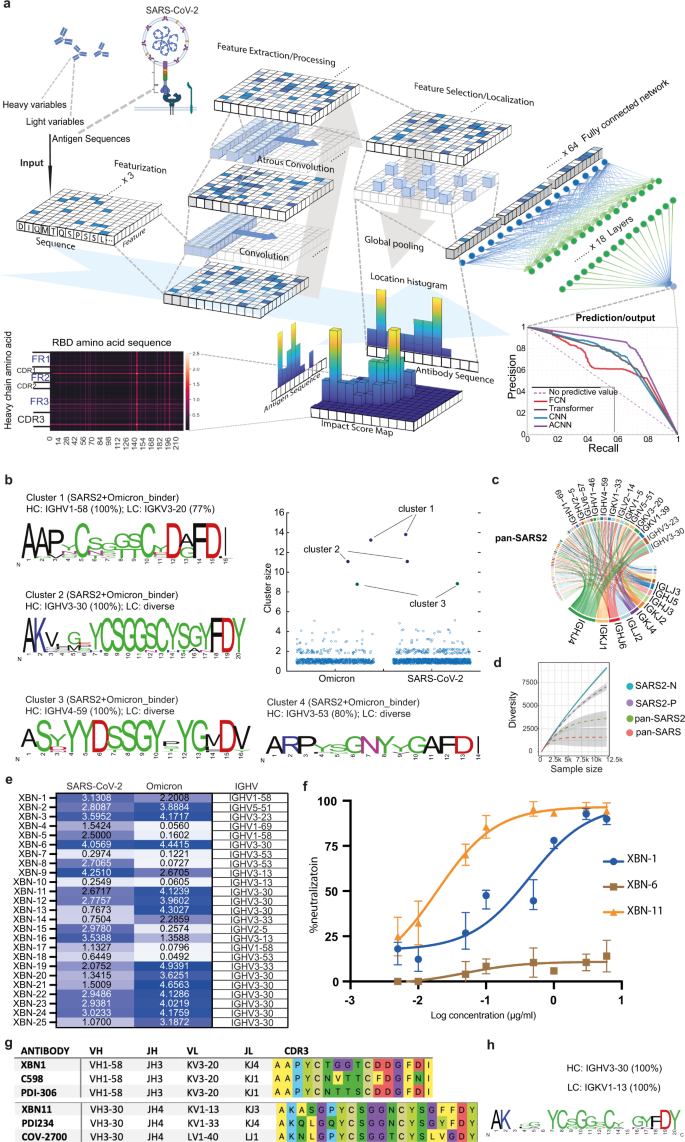
不断发展的SARS-CoV-2需要快速预测抗体与新变体的结合,并开发广泛中和的抗体。考虑到深度学习在抗体工程和优化中的应用,我们想知道针对SARS-CoV-2变体的广泛反应性抗体是否可以通过深度学习快速设计和生成。在这里,我们报告一个阿特劳斯卷积神经网络(ACNN)的发展。gydF4y2Ba4gydF4y2Ba基于深度学习框架:交叉反应性B细胞受体网络(XBCR-net),可以直接从单细胞BCR序列预测针对SARS-CoV-2和VOCs的广泛反应性抗体。XBCR-net由两部分组成,第一部分通过三分支ACNN提取抗体-抗原相互作用的相关特征,第二部分通过残差结构多层感知器(residual structural Multi-Layer Perceptron)预测抗体与抗原(14个不同的RBD序列)的结合概率(图。gydF4y2Ba1gydF4y2Ba;gydF4y2Ba补充信息gydF4y2Ba,无花果。gydF4y2BaS1agydF4y2Ba).对基于acnn的XBCR-net预测SARS-CoV-2结合的性能进行了评估,结果表明,与其他框架相比,XBCR-net的准确率、精密度和召回值均显著提高(图2)。gydF4y2Ba1gydF4y2Ba;gydF4y2Ba补充信息gydF4y2Ba,无花果。gydF4y2Ba印地gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba提取VH、VL和RBD序列的氨基酸序列特征,进行局部定位和max-pooled,连接在一起作为全连接层的输入。利用多层感知机(Multi-Layer Perceptron)处理潜伏空间中的活性特征,预测抗体与多种抗原的结合概率。在局部直方图影响评分图上计算VH、VL和RBD的影响评分,表示指定氨基酸在VH、VL (gydF4y2BaygydF4y2Ba轴)和RBD (gydF4y2BaxgydF4y2Ba轴)。通过ACNN(紫色)、Transformer(灰色)、FCN(红色)和CNN(蓝色)的Precision-Recall曲线对预测结果进行评价。gydF4y2BabgydF4y2Ba预测的SARS-CoV-2和Omicron变体结合剂的HCDR3序列采用80%序列相似度聚类。簇大小表示簇中BCR序列的数量。对于每个展开的集群,HCDR3序列被可视化为序列标识图,其中gydF4y2BaygydF4y2Ba-轴表示单个氨基酸在中相应位置的频率gydF4y2BaxgydF4y2Ba设在。主导VH基因的频率列在标志上方。gydF4y2BacgydF4y2Ba显示泛sars2序列的特定V区到J区配对编码的抗体频率的Circos图。gydF4y2BadgydF4y2Ba分析了四组BCR曲目的多样性,这与每组的样本数有关。gydF4y2BaegydF4y2Ba通过ELISA检测预测的交叉反应抗体与SARS-CoV-2的RBD和Omicron变体(左图)的结合。具有代表性的外径读数绘制为热图,范围为0.05至5.0,外径0.1为临界值(gydF4y2BangydF4y2Ba=每组3名)。gydF4y2BafgydF4y2Ba通过荧光素酶8次稀释的读数,生成SARS-CoV-2 Omicron变体(BA.1) XBN-1、XBN-6和XBN-11单抗伪病毒中和曲线(gydF4y2BangydF4y2Ba= 3)。gydF4y2BaggydF4y2BaXBN-1和XBN-11的HCDR3序列与已发表的研究中最收敛的抗sars - cov -2抗体一致。gydF4y2BahgydF4y2Ba泛sars组的优势簇(由IGHV3-30和IGKV1-13编码)的HCDR3序列频率如图所示。gydF4y2Ba
为评估XBCR-net对未见VOCs的适应性,采用Omicron新变体(BA.1、BA.2和BA.4)的RBD和142种抗Omicron单克隆抗体(包括治疗性抗体LY-CoV016、AZD-1061、REGN10933和S309)进行测试。gydF4y2Ba5gydF4y2BaXBCR-net预测142种结合物中有102种为阳性,142种非结合物(抗sars - cov -2抗体,不与Omicron结合)中有116种为阴性,说明XBCR-net在预测与Omicron结合抗体时的实用性(gydF4y2Ba补充信息gydF4y2Ba、表gydF4y2BaS1gydF4y2Ba、数据gydF4y2BaS3gydF4y2Ba).gydF4y2Ba
然后,我们使用XBCR-net从COVID患者(GSE171703)的单细胞BCR数据集中预测野生型(WT) SARS-CoV-2和VOC结合物,这些患者未被Omicron变体感染。gydF4y2Ba6gydF4y2Ba基于预测SARS-CoV-2结合剂和Omicron变体结合剂80% HCDR3序列相似性,我们鉴定了153和89个簇(图5)。gydF4y2Ba1 bgydF4y2Ba).3个簇的大小大于8,预计对SARS-CoV-2和Omicron变体具有交叉反应。优势聚类(聚类1)与IGHV1-58编码的一种经过充分研究的公共克隆型高度收敛,包括治疗性抗体Tixagevimab,据报道Tixagevimab可中和SARS-CoV-2。gydF4y2Ba7gydF4y2Ba另外两个簇(簇2和簇3)也分别属于由IGHV3-30(如治疗性抗体REGN 10987)和IGHV4-59 v区(如交叉反应性抗体47D11)编码的公共抗sars - cov -2克隆型(图3)。gydF4y2Ba1 bgydF4y2Ba).在一些研究中也描述了簇4抗体,如COV2-2733和COV2-2752,它们与SARS-CoV-2结合,但不与SARS-CoV结合。gydF4y2Ba8gydF4y2Ba
XBCR-net预测,6743个BCRs中有336个对WT型SARS-CoV的RBD区域及其VOCs(泛sars2,包括alpha、beta、delta和gamma变体)具有交叉反应,而其中只有54个对SARS-CoV的RBD(泛sars)具有交叉反应。泛sars -2的V-J区IGHV3-30、IGHV3-23和IGKV1-39基因使用率略高(图5)。gydF4y2Ba1 cgydF4y2Ba).与泛sars抗体序列相比,在泛sars抗体序列中观察到IGHV3-30的使用更偏倚,多样性更低(图2)。gydF4y2Ba1 dgydF4y2Ba;gydF4y2Ba补充信息gydF4y2Ba,无花果。gydF4y2BaS2a bgydF4y2Ba).gydF4y2Ba
由于我们在预测的交叉反应性RBD结合剂中观察到IGHV3-30的偏倚使用和IGHV3-30簇的扩大(图2)。gydF4y2Ba1 cgydF4y2Ba;gydF4y2Ba补充信息gydF4y2Ba,无花果。gydF4y2BaS2a-cgydF4y2Ba),我们从过滤的抗体列表中选择了10个IGHV3-30抗体和15个不同IGHV用法的抗体(描述在gydF4y2Ba补充信息gydF4y2Ba).与阴性对照抗体相比,所有25单抗在1 μg/mL时均能与WT SARS-CoV-2的RBD结合。与Omicron验证数据集一致,25个单克隆抗体中有20个对SARS-CoV-2 Omicron变体的RBD具有1 μg/mL的交叉反应。gydF4y2Ba1 egydF4y2Ba).有趣的是,我们研究中所有的IGHV3-30抗体都能结合Omicron变体(图。gydF4y2Ba1 egydF4y2Ba).为了进一步实证验证XBCR-net,我们将其应用于克隆的25株SARS-CoV结合单克隆抗体。在预测与SARS-CoV发生交叉反应的8个单克隆抗体中,有6个与SARS-CoV的RBD显著结合(gydF4y2Ba补充信息gydF4y2Ba,无花果。gydF4y2BaS3gydF4y2Ba).这些结果证明了XBCR-net在不需要额外训练数据的情况下,可以推断出BCR的交叉反应性。gydF4y2Ba
据报道,SARS-CoV-2 Omicron变体可以逃避一些治疗性单抗药物的中和。然后,我们测试了这些交叉反应单克隆抗体对SARS-CoV-2的Omicron和delta变体的中和能力。XBN-1与IC对delta (B.1.617.2)和Omicron (BA.1)均有中和活性gydF4y2Ba50gydF4y2Ba分别为7 ng/mL和418 ng/mL。XBN-6用IC中和delta (D614G)gydF4y2Ba50gydF4y2Ba而XBN-11在IC作用下对Omicron (BA.1)中和gydF4y2Ba50gydF4y2Ba17ng /mL(图;gydF4y2Ba1 fgydF4y2Ba;gydF4y2Ba补充信息gydF4y2Ba、表gydF4y2BaS2gydF4y2Ba).gydF4y2Ba
由于SARS-CoV-2在不断进化,因此需要迅速更新新变体的治疗方法,以便临床决策。gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba我们的XBCR-net在获得RBD序列后,可以快速预测新发现的SARS-CoV-2变体的抗体结合。我们克隆的IGHV1-58单抗与已发表的中和SARS-CoV-2的抗体PDI-306和C598具有收敛性。我们克隆的IGHV3-30单抗显示出与已发布的冠状病毒抗体PDI234和COV2-2700的重链收敛(图2)。gydF4y2Ba1gydF4y2Bag). PDI234和COV2-2700不与SARS-CoV结合,gydF4y2Ba8gydF4y2Ba这表明HCDR3和不同轻链(IGKV1-13)上的关键突变可以使IGHV3-30克隆型单抗交叉反应。通过XBCR-net的预测,我们发现IGHV3-30和IGKV1-13编码的聚类衍生的单抗除SARS-CoV-2外,还能与SARS-CoV和Omicron变体结合(图2)。gydF4y2Ba1 egydF4y2Bah;gydF4y2Ba补充信息gydF4y2Ba,无花果。gydF4y2BaS3gydF4y2Ba),表明我们发现的IGHV3-30、IGKV1-13编码簇可以进一步发展成为广泛中和SARS-CoV和SARS-CoV-2的抗体。总之,我们的XBCR-net可以预测针对新发现的SARS-CoV-2变体的广泛反应性抗体,而无需事先了解新的变体特异性抗体,有助于快速生成针对SARS-CoV-2变体和其他新出现的病毒的抗体。gydF4y2Ba
数据可用性gydF4y2Ba
神经网络模型已经上传到githubgydF4y2Bahttps://github.com/jianqingzheng/XBCR-netgydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
施密特,F.等。gydF4y2Ba心血管病。j .地中海。gydF4y2Ba386gydF4y2Ba, 599-601(2022)。gydF4y2Ba
曹毅,等。gydF4y2Ba自然gydF4y2Ba602gydF4y2Ba, 657-663(2022)。gydF4y2Ba
Dejnirattisai, W.等。gydF4y2Ba细胞gydF4y2Ba185gydF4y2Ba, 467 - 484。e15(2022).
周晓燕,郑建强,李培平,杨国忠gydF4y2Ba2020年IEEE机器人与自动化国际会议(ICRA)gydF4y2Ba8455 - 8461(2020)。gydF4y2Ba
雷布尔德,m.i.j,科瓦尔津克,A,马克斯,C. &迪恩,C. M.。gydF4y2Ba生物信息学gydF4y2Ba37gydF4y2Ba, 734-735(2021)。gydF4y2Ba
杜根,H. L.等人。gydF4y2Ba免疫力gydF4y2Ba54gydF4y2Ba, 1290 - 1303。e7(2021)。gydF4y2Ba
Bruel, T.等。gydF4y2BaNat,地中海。gydF4y2Ba28gydF4y2Ba, 1297-1302(2022)。gydF4y2Ba
佐斯特,S. J.等。gydF4y2BaNat,地中海。gydF4y2Ba26gydF4y2Ba, 1422-1427(2020)。gydF4y2Ba
刘,L.等。gydF4y2Ba自然gydF4y2Ba602gydF4y2Ba, 676-681(2022)。gydF4y2Ba
Shan, S.等。gydF4y2BaProc。国家的。学会科学。美国gydF4y2Ba119gydF4y2Ba, e2122954119(2022)。gydF4y2Ba
确认gydF4y2Ba
国家自然科学基金(81788101)和中国科学院医学科学创新基金(2021-I2M-1-017)资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
X.C.和H.L.设计并监督了这项研究;洪磊、江志忠、薛峰、张志良、程伟进行了实验和生物信息学分析;j.z., Y.C, M.Z.和X.F.建立并优化了神经网络;洪磊、J.Z、X.F、z.l、x.c对数据进行分析并撰写了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
补充信息gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
娄宏,郑俊,方兴。gydF4y2Baet al。gydF4y2Ba基于深度学习的快速生成针对SARS-CoV-2及其Omicron变体的广泛反应性抗体。gydF4y2Ba细胞ResgydF4y2Ba33gydF4y2Ba, 80-82(2023)。https://doi.org/10.1038/s41422-022-00727-6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41422-022-00727-6gydF4y2Ba