摘要gydF4y2Ba
通过组蛋白修饰对转录控制的定量表征一直受到许多计算研究的挑战,但大多数研究只关注启动子周围狭窄和线性的基因组区域,有改进的空间。我们提出了Chromoformer,一种基于变压器的三维染色质构象感知深度学习架构,在基因调控中组蛋白编码的定量破译中实现了最先进的性能。Chromoformer结构的核心本质在于注意操作的三种变体,每一种都专门用于模拟转录调节的个体层次,涉及从核心启动子到通过三维染色质相互作用与启动子接触的远端元件。对Chromoformer的深入解释表明,它自适应地利用了与转录起始和延伸相关的组蛋白修饰之间的长期依赖性。我们还表明,转录工厂和Polycomb基团体的定量动力学可以被Chromoformer捕获。总之,我们的研究突出了基于注意力的表观基因组复杂相互作用深度建模的巨大优势。gydF4y2Ba
简介gydF4y2Ba
基因表达的控制由不同组的调控因子进行,包括转录因子、辅激活因子、辅抑制因子以及基因组序列元件。然而,这些因素之间相互作用的基本前提是组蛋白尾共价修饰或组蛋白修饰(HMs)在相关基因组区域的适当配置,因为它们在染色质可及性调控中起着关键作用。因此,可以认为,大量的HMs及其组合编码了附近基因组区域的调控潜力。gydF4y2Ba
这个概念被称为“组蛋白编码假说”。gydF4y2Ba1gydF4y2Ba.已经有许多计算和定量的方法来破解由HMs编码的基因表达的调控密码。其中大部分是预测模型,利用转录起始位点(tss)周围启动子的HMs水平来预测相应基因的表达水平。值得注意的是,最近的研究表明,与传统的机器学习模型相比,深度学习模型在这项任务中的表现更优越gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba.gydF4y2Ba
迄今为止,深度学习已经在计算生物学的各个领域取得了显著的突破,从结合的特性到DNA和RNA结合蛋白的特异性gydF4y2Ba4 gydF4y2Ba针对基于氨基酸序列预测蛋白质结构这一长期存在的问题gydF4y2Ba5克ydF4y2Ba.如果没有新型模型架构的发明,以及它们在复杂生物学问题上的巧妙应用,深度学习在生物学领域的这些成功是不可能实现的。从这个意义上说,组蛋白编码的高度复杂性确实使其成为深度学习的一个很好的目标,正如现有方法所示,但它们仍然构成了两个主要的限制,这促使了新方法的发展。gydF4y2Ba
一是他们只能利用tss周围狭窄的基因组窗口。这是因为这些模型所基于的深度学习架构,如卷积神经网络(cnn)和循环神经网络(RNNs),在建模长序列中的依赖关系时并不有效。cnn在学习数据的局部模式方面是高度专门的,但对它们来说,学习模式之间的遥远依赖关系是一个挑战。虽然RNN体系结构是为了建模顺序数据而开发的,但在清楚地捕获长期依赖关系方面也存在困难,因为嵌入在单个位置的信息会逐渐稀释,并在模型计算沿着两个遥远位置之间的位置移动时受到污染。事实上,高级形式的RNN细胞,如门控循环单元gydF4y2Ba6gydF4y2Ba或长短期记忆(LSTM)gydF4y2Ba7gydF4y2Ba在一定程度上改善了这一问题,但长序列建模中由于递归而固有的低效率仍然存在。gydF4y2Ba
其次,大多数深度学习模型都没有考虑远端gydF4y2Ba独联体gydF4y2Ba-由三维(3D)染色质折叠介导的调控,尽管人们已经广泛知道核心启动子和远端启动子之间的物理相互作用gydF4y2Ba独联体gydF4y2Ba-调控元件对基因表达起关键调控作用gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba.换句话说,组蛋白编码传递的调控信息不仅可以在局部传播,还可以通过三维染色质相互作用在遥远的基因组位点之间跳跃gydF4y2Ba10gydF4y2Ba.幸运的是,最近高通量测量技术如Hi-C的进步gydF4y2Ba11gydF4y2Ba成功地在千碱基尺度上提供了3D染色质相互作用的精细分辨率视图,并为我们提供了前所未有的机会来利用这些有价值的信息来建模基因调控的全面视图。很少有新兴的研究明确地将3D染色质相互作用考虑到预测基因表达。GC-MERGE就是这样一个例子gydF4y2Ba12gydF4y2Ba图神经网络(GNN)在相互作用的基因组区域之间传播信息,以预测基因的表达水平。虽然这是一个概念验证模型,不能应用于没有任何染色质相互作用的基因,并且只能执行10 kbp基因组箱水平的预测,但不是在基因水平,它仍然强调了远端基因组区域的表观基因组环境建模以及启动子的前景。gydF4y2Ba
同时,一个名为transformer的深度学习模型架构,最初是为自然语言处理而开发的gydF4y2Ba13gydF4y2Ba在理解DNA序列的潜在语法方面显示出巨大的潜力gydF4y2Ba14gydF4y2Ba,氨基酸序列gydF4y2Ba15gydF4y2Ba,甚至他们的路线gydF4y2Ba16gydF4y2Ba.特别是,在本研究中,我们注意到变压器架构的两个主要功能非常适合解决上述两个挑战。首先,变压器可以精确地模拟序列数据中的长期依赖关系。这是通过向输入序列添加位置编码来实现的。这些包含位置信息的输入特征被独立处理,并输入到随后的自注意模块中,该模块计算输入特征之间的全成对依赖关系。因此,可以捕获长期依赖关系,而不受位于对之间的特征的干扰。其次,变压器体系结构还可以应用于对无序实体集及其之间的交互进行建模。值得注意的是,这对于大多数深度学习架构来说并不简单,因为组成它们的操作取决于输入位置。另一方面,组成变压器的操作基本上是置换不变的。输入特征之间的相互作用只在自注意操作中考虑,所有其他操作都以位置方式完成,因此它们可以应用于一个无序特征集的模型。 Together, these two strengths of the transformer architecture make it a promising choice for the quantitative modeling of histone codes by allowing us to utilize wider genomic windows near TSSs and histone codes at multiple distal regulatory regions simultaneously.
在这里,我们提出了一个名为Chromoformer的基于变压器的深度学习架构,基于tss宽邻域的HMs以及位于远端调控元件的HMs来预测基因表达水平。Chromoformer基于三种反映三维基因调控层次的自注意操作变体的模型架构,在基因表达预测方面取得了比其他深度学习模型更好的预测能力。此外,通过对transformer架构的使用如何促进模型的卓越性能的全面调查,我们证明了TSS附近表观遗传背景的远程建模和远端调控区域的同时集成建模实际上可以提高性能。最后,我们表明,我们可以为定量效应绘制人工智能驱动的假设gydF4y2Ba独联体gydF4y2Ba-通过Chromoformer学习到的调控状态的潜在嵌入动态的解释,细胞核内的两个子域,转录工厂和沉默中心的调控。gydF4y2Ba
结果gydF4y2Ba
显色仪采用三级变压器结构,体现了基因三维调控的层次性gydF4y2Ba
显色仪的核心设计原则是双重的。一种是从核心启动子的HMs中提取尽可能多的近端调控信息,另一种是整合远端组蛋白编码,其信息通过3D染色质相互作用传输到核心启动子。充分利用变压器结构来模拟复杂的动力学gydF4y2Ba独联体gydF4y2Ba由于涉及多层调控,我们从概念上将基因调控分解为三层结构:(1)gydF4y2Ba独联体gydF4y2Ba(2)核心启动子与假定启动子之间的三维成对相互作用gydF4y2Ba独联体gydF4y2Ba-调控区域(pcre)和(3)三维成对相互作用所施加的集体调控效应。为了在计算上模拟这种层次结构,我们引入了三个基于变压器的子模块,分别称为Embedding、Pairwise Interaction和Regulation transformer,它们专门用于按照复杂度增加的顺序学习基因表达调控的各自语法。gydF4y2Ba
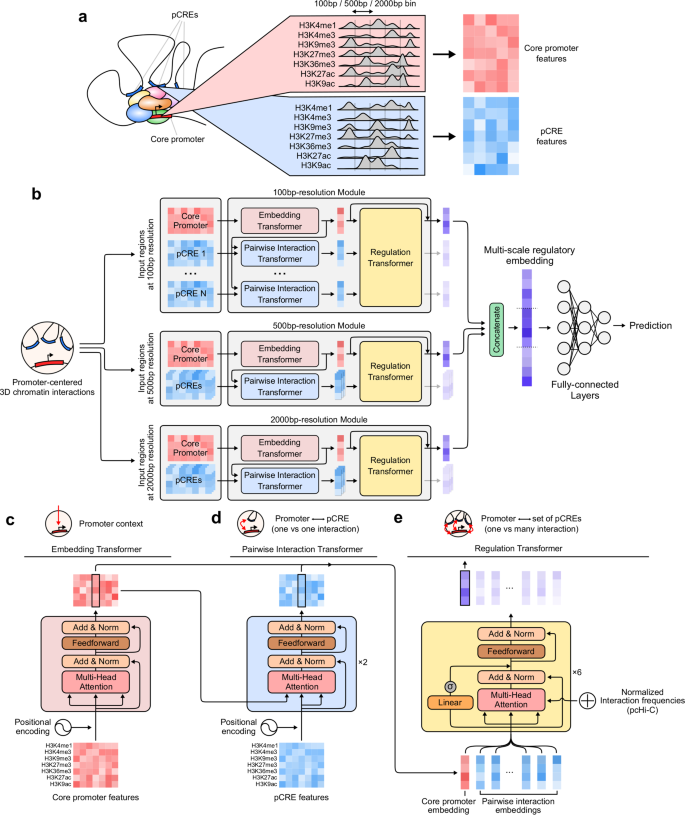
在说明模型架构之前,我们简要描述了整个研究中使用的输入特征。显色仪使用来自组蛋白ChIP-seq实验的读取深度值对七种主要HMs (H3K4me1、H3K4me3、H3K9me3、H3K27me3、H3K36me3、H3K27ac和H3K9ac)进行训练(补充图)。gydF4y2Ba1gydF4y2Baa).在tss两侧的40 kbp区域内,对固定大小的容器读取深度进行平均和log2转换(图2)。gydF4y2Ba1gydF4y2Ba和补充图。gydF4y2Ba1gydF4y2Bab).考虑远端gydF4y2Ba独联体gydF4y2Ba-调控,我们还利用了已知与相应细胞类型的核心启动子相互作用的pcre上的HM信号(补充图。gydF4y2Ba1gydF4y2Bac).为此,使用公开的启动子捕获Hi-C (pcHi-C)数据集,获得了每个核心启动子的一组经实验验证的pcregydF4y2Ba17gydF4y2Ba.三维染色质相互作用在HindIII限制性内切片段的分辨率下进行了表征。这些相互作用具有足够的高分辨率,这些片段的中位数和平均长度分别为4797 bp和5640 bp,其中约95%的片段小于10 kbp (Supplementary Fig。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba输入功能。为了使用组蛋白修饰(HMs)水平来预测基因的表达,我们从核心启动子和假定的HMs中提取了分组平均信号gydF4y2Ba独联体gydF4y2Ba-调控区域(pcre)。gydF4y2BabgydF4y2BaChromoformer架构。三个独立的模块被用来产生基因表达调控的多尺度表示。每个模块以不同分辨率输入HM特征,以产生反映核心启动子调控状态的嵌入向量。gydF4y2BacgydF4y2Ba嵌入变压器架构。核心启动子特征的位置编码HM信号通过自注意转化为核心启动子嵌入。gydF4y2BadgydF4y2Ba成对交互变压器架构。利用pcre的位置编码HM信号,通过编码器-解码器注意将核心启动子嵌入转化为成对交互嵌入。gydF4y2BaegydF4y2Ba调节变压器结构。利用核心启动子、成对交互嵌入和门控自注意的一整套方法,调节转换器学习pcre如何集体调节核心启动子。为了引导模型更加关注频繁发生的三维交互,在自注意亲和矩阵中加入归一化交互频率向量。gydF4y2Ba
本研究中使用的完整模型架构如图所示。gydF4y2Ba1 bgydF4y2Ba.在最高级别上,它由三个独立的模块组成,每个模块以不同的分辨率接受输入特征,并反过来在核心启动子处产生调控状态的嵌入向量。将得到的3个调控嵌入物连接起来,形成一个多尺度调控嵌入物,随后将其送入全连接层,以预测基因的表达水平。使用多尺度调节嵌入比使用任何单一分辨率调节嵌入具有更好的性能,并且所有三种分辨率的组合比任何两种分辨率的组合具有更高的性能提升(补充图。gydF4y2Ba3.gydF4y2Ba).同时,利用自注意操作将不同分辨率下的规则嵌入进行组合gydF4y2Ba13gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba与拼接相比似乎没有显著优势,因此为了模型的简单性,我们决定拼接监管嵌入(补充图。gydF4y2Ba4 gydF4y2Ba).嵌入变压器(图。gydF4y2Ba1 cgydF4y2Ba)学习作用于TSS直接附近的组蛋白编码,并产生一个固定大小的向量,总结该区域的表观遗传状态。这个子模块本身的工作原理与现有的基于HM的基因表达预测的机器学习模型非常相似,但我们期望使用变压器架构将允许模型在核心启动子HM上下文的大范围视图(高达40 kbp)内精确识别相关信号,而不会有任何性能下降。接下来,得到的核心启动子嵌入被Pairwise Interaction变压器进一步更新(图2)。gydF4y2Ba1 dgydF4y2Ba)在成对的情况下gydF4y2Ba独联体gydF4y2Ba-启动子和pcre之间的调控相互作用。这个模块使用编码器-解码器注意层,而不是像嵌入转换器中那样使用典型的自注意层。由于启动子的活性是由与pCRE的接触调节的,因此选择编码器-解码器框架来通过解码给定pCRE特征的启动子嵌入来反映这一点。我们称所得到的嵌入向量为成对交互嵌入,因为它们携带了启动子和pcre之间一对一关系的信息。最后,调节变压器(图。gydF4y2Ba1 egydF4y2Ba)接受由核心启动子和成对交互嵌入组成的并集,最后将其整合生成调控嵌入。这个模块建模的整个景观gydF4y2Ba独联体gydF4y2Ba-使用门控自我注意层进行调节。将归一化的交互频率(参见补充方法1)注入到自我注意评分矩阵中,以指导模型确定交互的优先级。模型的详细解释见方法和补充注1。gydF4y2Ba
显色器优于现有的基于表观遗传特征的基因表达预测深度模型gydF4y2Ba
我们使用各自研究中提出的最佳设置,用三个基线深度学习模型对Chromoformer的性能进行了基准测试。我们首先训练DeepChromegydF4y2Ba2gydF4y2Ba,一种卷积神经网络,通过卷积滤波器的权重学习HMs的局部组合来预测基因表达。我们还训练了attentivchromegydF4y2Ba3.gydF4y2Ba和DeepDiffgydF4y2Ba21gydF4y2Ba模型。前者结合LSTM和全局注意力机制增强模型的可解释性,后者将其扩展到预测一对细胞类型之间基因表达的折叠变化。最后,Kang等提出了一种基于hmm的混合CNN-RNN模型。gydF4y2Ba22gydF4y2Ba(HM-CRNN)也被用于比较。它通过CNN学习并捕获有意义的HMs局部组合,并通过RNN理解它们的顺序依赖关系。gydF4y2Ba
通过三种不同的基因表达预测任务:二元基因表达状态分类、表达水平回归和表达折叠变化回归,综合评估Chromoformer的预测性能。在二元基因表达状态分类任务中,模型预测某个基因的表达水平是否高于中位数。这个问题的提法是由Singh等人首先提出的。gydF4y2Ba2gydF4y2Ba,到目前为止,它已被许多研究广泛采用,包括上述三个基线研究。在表达水平回归任务中,训练模型预测log2转换的RPKM值,表达折叠变化回归任务评估性能以预测每个基因在两种细胞类型之间的表达折叠变化。对于每个任务,我们设计了一个定制的Chromoformer模型变体(图2)。gydF4y2Ba2gydF4y2Ba),所有这些都是基于Chromoformer的多尺度骨干(图;gydF4y2Ba1中gydF4y2Ba).针对二元基因表达状态分类任务,建立了染色体前分类器(Chromoformer-clf)。它有一个具有完全连接层的分类头,该分类头产生一个二维概率向量,表示高表达或低表达的概率。chromoformer -回归器(Chromoformer-reg)设计用于表达水平回归任务,使用一个回归头产生单个标量。用于折叠变化回归的显色仪(Chromoformer-diff)采用Siamese神经网络架构来接受来自两种不同细胞类型的HM剖面(补充图)。gydF4y2Ba5克ydF4y2Baa).两条Chromoformer骨干共享权值,使两条HM剖面嵌入同一潜空间,并由后续回归头将其差异非线性转化为折变值。gydF4y2Ba
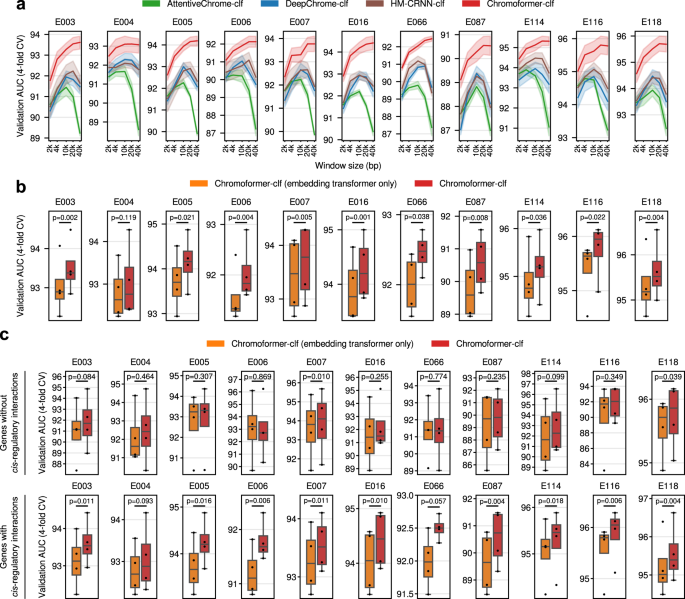
一个gydF4y2Ba本研究评估了三种不同的显色剂。Chromoformer-classifier (Chromoformer-clf)预测二元基因表达标签。Chromoformer-reg预测基因的表达水平(以log2 RPKM为单位)。Chromoformer-diff预测每个基因在两种不同细胞类型的HM配置下基因表达的log2 fold变化。gydF4y2BabgydF4y2Ba交叉验证(gydF4y2BangydF4y2Ba= 4)与仅利用核心启动子特征的基准深度学习模型相比,Chromoformer-clf的性能。gydF4y2BacgydF4y2Ba交叉验证的比较(gydF4y2BangydF4y2Ba= 4) GC-MERGE的性能,这是一种利用3D的图神经网络模型gydF4y2Ba独联体gydF4y2Ba监管的交互。为了进行公平的比较,Chromoformer-clf模型只使用GC-MERGE可以预测的一个基因子集进行重新训练。gydF4y2BadgydF4y2Ba交叉验证(gydF4y2BangydF4y2Ba= 4)与基准深度学习模型相比,Chromoformer-reg的性能。对基准模型的预测头进行了修改,以产生单个标量值而不是二进制标签。gydF4y2BaegydF4y2Ba与基准深度学习模型相比,Chromoformer-diff的交叉验证(n=4)性能。基于分类概率比值的折叠变化预测的评估显示为参考(表示为DeepChrome-clf, attentivchrome -clf, HM-CRNN-clf和Chromoformer-clf)。gydF4y2BafgydF4y2BaDeepDiff与Chromoformer-diff的两两性能比较。在gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BaegydF4y2Ba中心线表示中位数,上下方框界限表示上四分位数和下四分位数,胡须表示1.5×四分位数范围。AUC,接受者工作特性曲线下面积;简历,交叉验证。源数据作为源数据文件提供。gydF4y2Ba
在路线图表观基因组学分析的127种细胞类型中,评估了11种细胞类型的模型性能gydF4y2Ba23gydF4y2Ba和ENCODE项目gydF4y2Ba24gydF4y2Ba.之所以选择这11种细胞类型,是因为每种细胞类型的所有基因表达谱、7种主要HMs的ChIP-seq数据和pcHi-C相互作用谱都是公开可用的。将18,955个基因分成4组进行4倍交叉验证(CV),每组分别由5045、4751、4605和4554个基因组成。对于每一个CV折叠,每个集都成为一个保留的验证集,而其他三个集则用于模型训练。为了避免通过涉及启动子-启动子相互作用的3D染色质折叠将不必要的信息从训练集泄漏到验证集,我们确保不同集中的两个基因不位于同一染色体上。gydF4y2Ba
结果,我们的多尺度Chromoformer模型在所有三个任务中都比现有的基线深度学习模型取得了显著的性能提升,这表明所提出的模型架构在建模基因表达的调控层次方面是成功的(图2)。gydF4y2Ba2 b-fgydF4y2Ba和补充图。gydF4y2Ba5克ydF4y2Bab,gydF4y2Ba6gydF4y2BaA b和gydF4y2Ba7gydF4y2Ba).这些结果在所有11种被检测的细胞类型中得到了一致的再现。具体而言,对于二元基因表达状态分类任务,Chromoformer-clf在接受者工作特征曲线下面积(ROC-AUC)方面较现有基线深度学习模型取得了显著的性能提升(图。gydF4y2Ba2 bgydF4y2Ba)、精度、平均精度(补充图。gydF4y2Ba6 a、bgydF4y2Ba).此外,我们发现Chromoformer-clf产生的预测概率与实际表达水平呈非常高的正相关(补充图。gydF4y2Ba6gydF4y2Bac).这些对定量表达水平校正良好的预测概率支持使用二元分类公式对HMs进行定量建模。Chromoformer-clf也远远优于GC-MERGE, GC-MERGE是一种使用三维染色质相互作用预测基因表达的GNN(图2)。gydF4y2Ba2摄氏度gydF4y2Ba).重要的是,GC-MERGE只能预测参与至少一种染色质相互作用的基因。此外,GC-MERGE只能以10 kbp基因组箱为单位预测基因表达,因此当两个或多个基因存在于同一个箱子中时,GC-MERGE无法进行基因预测。因此,我们对Chromoformer进行了重新训练,选择了可以通过GC-MERGE预测表达的基因子集,以进行公平的比较。同时,Chromoformer-reg在Pearson相关系数方面优于基准模型的回归变量(图。gydF4y2Ba二维gydF4y2Ba),gydF4y2BaR \ ({} ^ {2} \)gydF4y2Ba(补充图。gydF4y2Ba7gydF4y2Ba).相比于最先进的DeepDiff模型,显色前diff也有更好的性能。gydF4y2Ba2 egydF4y2Ba, f).值得注意的是,它也比使用分类模型预测概率与预测折叠变化之比的回归性能好得多(图。gydF4y2Ba2 egydF4y2Ba).总的来说,这些结果表明显色结构在表观遗传基因调控预测中的有效性。gydF4y2Ba
训练与大窗口大小和gydF4y2Ba独联体gydF4y2Ba-调控相互作用有助于Chromoformer的性能改善gydF4y2Ba
为了将显色器的性能分析为单个因素的贡献,我们首先考察了在TSS周围建模高达40 kbp的大范围窗口的效果。通过逐渐增加TSS附近的窗口大小从2 kbp到40 kbp,我们观察到我们的模型的性能持续增加,而其他深度学习模型在窗口大小大于10 kbp时表现出相当大的性能下降(图2)。gydF4y2Ba3gydF4y2Ba).更大的窗口更容易包含其他基因的tss,这就影响了模型的性能,因为模型更容易对那些不相关的tss产生虚假关注。这是因为每个基因组位置的注意力得分是根据全局上下文向量计算的,而不考虑到目标TSS的绝对距离。然而,在变压器架构的情况下,带有位置编码的缩放点积注意允许在所有位置对之间计算注意分数,而无需引入全局上下文向量,从而允许我们查询输入窗口中我们感兴趣的特定TSS所关注的位置。因此,这些结果强调了变压器结构在精确定位组蛋白编码方面的力量,这些组蛋白编码与TSS周围大范围窗口内基因表达的调控相关。gydF4y2Ba
一个gydF4y2BaTSS前后输入窗口大小对模型性能的影响。显色仪和其他基准模型分别针对5种不同的窗口大小(2 kbp、4 kbp、10 kbp、20 kbp和40 kbp)进行训练,而所有其他训练程序保持不变。粗体线表示每个窗口大小的4倍交叉验证的平均验证AUC,而阴影表示它们的标准误差。gydF4y2BabgydF4y2Ba取远端效果gydF4y2Ba独联体gydF4y2Ba-考虑到pCREs的规定。我们训练的烧蚀显色模型只有嵌入变压器,因此不能合并gydF4y2Ba独联体gydF4y2Ba-核心启动子和pcre之间的调控信息。结果的交叉验证(gydF4y2BangydF4y2Ba= 4)性能与完整的显色器模型进行比较。gydF4y2BacgydF4y2Ba交叉验证的比较(gydF4y2BangydF4y2Ba= 4)没有或有已知染色质相互作用的基因子集的表现。仅针对未知的基因亚群,计算了Embedding transformer-only Chromoformer和完整Chromoformer的ROC-AUC评分gydF4y2Ba独联体gydF4y2Ba-调控相互作用(Upper),以及至少一种已知的基因gydF4y2Ba独联体gydF4y2Ba监管的交互。gydF4y2BaPgydF4y2Ba-values来自双面配对gydF4y2BatgydF4y2Ba-tests显示。在箱线图中,中心线表示中位数,上下箱限表示上四分位数和下四分位数,胡须表示1.5×四分位数范围。源数据作为源数据文件提供。gydF4y2Ba
我们接下来检查远端是否合并gydF4y2Ba独联体gydF4y2Ba与监管部门的交互作用真正促进了性能的提高。为此,我们从Chromoformer模型中消除了成对交互和调节变压器,以查看它在不使用远端训练时的表现如何gydF4y2Ba独联体gydF4y2Ba监管的交互。我们重新训练了烧蚀后的显色器模型,并将其与完整的显色器模型在ROC-AUC方面的性能进行了比较(图2)。gydF4y2Ba3 bgydF4y2Ba).这表明,完整形式的显色器在大多数所分析的细胞类型中表现出显著的高性能(11种中的10种),这意味着将远端pcre及其相互作用纳入深度建模有助于学习控制基因表达的表观遗传因素。进一步支持这种改进是由于建模的具体原因gydF4y2Ba独联体gydF4y2Ba-调控相互作用,但并不仅仅是因为模型的复杂性,我们分别研究了启动子不与任何pCREs相互作用的基因的建模性能。由于没有生物学上有意义的信息会通过Pairwise Interaction and Regulation transformer传递到这些基因的核心启动子嵌入,我们可以辨别导致性能提高的主要因素是基因组区域之间的信息传递还是模型复杂性本身的增加。正如预期的那样,我们观察到Chromoformer对大多数细胞类型没有显着改善(图。gydF4y2Ba3 cgydF4y2Ba).此外,对于至少有一种与pCREs相互作用的基因,性能的提高仍然是显著的(图。gydF4y2Ba3 cgydF4y2Ba).此外,tad内相互作用对性能的贡献大于tad间相互作用,再次支持远端生物学相关性gydF4y2Ba独联体gydF4y2Ba显色仪训练中的-调节相互作用(补充图;gydF4y2Ba8gydF4y2Ba).总之,它表明的贡献gydF4y2Ba独联体gydF4y2Ba-调控建模远远大于来自更深层次的建模能力的增加。gydF4y2Ba
显色原体学会关注基因体的远端转录延伸信号gydF4y2Ba
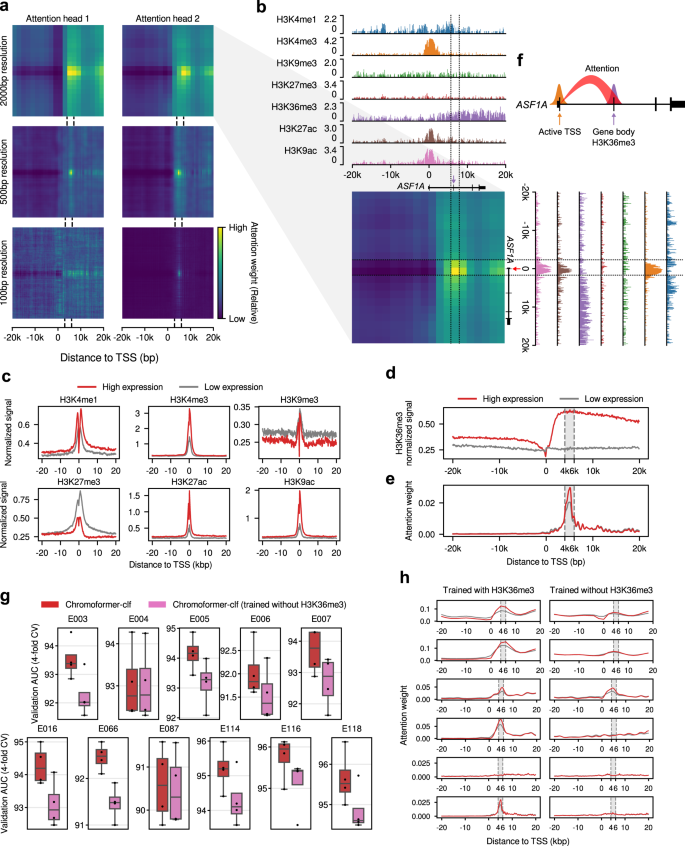
考虑到Chromoformer模型与其他深度学习模型相比性能的提高,以及核心启动子区域大范围建模的成功,我们随后询问嵌入变压器是否有任何独特的HMs捕获模式。由于嵌入转换器内的自注意层被设计为理解核心启动子上HMs的依赖关系,我们假设Chromoformer模型所意识到的任何此类依赖关系都可以通过它产生的自注意映射来揭示。因此,将Chromoformer-clf的Embedding transformer在预测过程中产生的注意权值可视化,以分析模型的内部行为。数字gydF4y2Ba4gydF4y2Ba给出了在预测抗沉默函数表达式1a时注意权重的实例快照(gydF4y2BaASF1AgydF4y2BaH1细胞中的组蛋白伴侣(表观基因组标识符E003)。有趣的是,我们观察到大多数注意头明显地对TSS下游4-6 kbp给予了强烈的注意,而不是任何其他特定位置(图2)。gydF4y2Ba4gydF4y2Ba, b).这是相当出乎意料的,因为已知HMs传递的大多数调控信息都沉积在TSS附近,转录因子的结合和转录启动主要发生在这里。根据这一概念,HMs的平均信号大多在TSS处表现出其特征模式(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).具体而言,H3K4甲基化和组蛋白乙酰化与较高水平的基因表达相关,而高水平的H3K9me3和H3K27me3修饰与低水平的基因表达相关。gydF4y2Ba
一个gydF4y2Ba预测H1细胞中ASF1A表达的代表性自我注意权重矩阵。每个热图显示了每对基因组箱的关注权重。虚线表示TSS下游4-6 kbp区域。gydF4y2BabgydF4y2Ba对于2kbp分辨率的嵌入变压器,注意头2学习的注意权重的详细描述。表示七个HMs归一化信号的基因组轨迹与注意力权重矩阵对齐。虚线划分了TSS下游4-6 kbp的区域。红色箭头表示TSS,紫色箭头表示位于TSS下游4-6 kbp区域内的外显子。gydF4y2BacgydF4y2Ba除H3K36me3外HMs的平均信号。根据信号的表达标签(高/低表达)分别取平均。gydF4y2BadgydF4y2Ba平均H3K36me3信号。gydF4y2BaegydF4y2Ba100 bp分辨率箱子的第二个注意头的平均注意权重。灰色阴影表示TSS下游4-6 kbp区域。gydF4y2BafgydF4y2Ba说明嵌入变压器行为的示意图。gydF4y2BaggydF4y2Ba交叉验证(gydF4y2BangydF4y2Ba= 4) H3K36me3特征烧蚀性能。中心线表示中位数,上下方框界限表示上四分位数和下四分位数,胡须表示1.5×四分位数范围。gydF4y2BahgydF4y2BaTSS bin的注意权重。红色和灰色线分别表示中位数以上和中位数以下表达的基因的平均注意力权重。源数据作为源数据文件提供。gydF4y2Ba
与之形成鲜明对比的是,两类基因(即高表达和低表达)的H3K36me3平均信号在TSS处无明显差异,而在TSS下游4 ~ 6 kbp区域差异最大(图2)。gydF4y2Ba4 dgydF4y2Ba).H3K36me3是通过SETD2组蛋白甲基转移酶在H3K36上添加甲基而建立的,并且已知SETD2被招募到RNA聚合酶的c端结构域,与转录延伸一致gydF4y2Ba25gydF4y2Ba.因此,H3K36me3被广泛认为是一种转录伸长相关的HM,主要标记活性转录基因的体。含有TSS的容器施加的平均注意权重恰好在TSS下游的4-6 kbp区域达到最大值(图2)。gydF4y2Ba4 egydF4y2Ba),这表明该模型经过了很好的优化,可以专注于H3K36me3信号方面最具鉴别性的基因组区域。有趣的是,对TSS下游4-6 kbp区域的关注程度对于高表达的基因要远远大于低表达的基因(图2)。gydF4y2Ba4 egydF4y2Ba).换句话说,根据TSS直接附近的组蛋白上下文,训练Embedding transformer自适应控制对基因体HMs的关注量,如图所示。gydF4y2Ba4 fgydF4y2Ba.这些自我注意权重的模式在所检查的细胞类型中高度一致(补充图)。gydF4y2Ba9gydF4y2Ba).嵌入转换器的这种行为的一种解释是,模型寻求补充证据,以加强其对基因表达的初始猜测的置信度,这是基于TSS附近的HM状态。从这个意义上说,H3K36me3是一个非常适合扮演这样一个角色的候选人,因为它的辨别能力存在于其他hm没有显示出很大的变化的地方,因此在这些区域中,H3K36me3是唯一的清晰信号。特征消融实验进一步支持了H3K36me3在下游基因体中的重要性。当H3K36me3信号被排除在模型训练之外,仅将其他6个hm作为输入特征时,我们观察到性能显著下降(图2)。gydF4y2Ba4 ggydF4y2Ba).此外,我们发现嵌入变压器主要对4-6 kbp下游区域失去了特定的注意(图4-6 kbp)。gydF4y2Ba4 hgydF4y2Ba).gydF4y2Ba
值得注意的是,与任何其他单个HMs的消融相比,H3K36me3消融对模型性能的影响最大。gydF4y2Ba10gydF4y2Baa).这意味着H3K36me3的分布可能不容易被其他hm推断出来,因为它与其他hm的空间相关性较低(补充图)。gydF4y2Ba10 bgydF4y2Ba).此外,虽然烧蚀与chromhmm定义的50个染色质状态相对应的其他HMs的组合通常导致性能较差,但H3K36me3的作用几乎是独立的(补充图。gydF4y2Ba10 c, dgydF4y2Ba).我们还注意到,烧蚀两个增强子标记H3K4me1和H3K27ac并不足以显著降低显色剂的性能,而进一步烧蚀H3K4me3和H3K9ac会导致性能急剧下降(补充图。gydF4y2Ba10 cgydF4y2Ba).这意味着活动HMs的空间相关性或冗余(补充图。gydF4y2Ba10 bgydF4y2Ba),有效地弥补了其他在职医疗服务经理所传达的规管信息的缺失。此外,这可能是由于H3K4me3或H3K9ac标记的活性启动子之间通过启动子-启动子相互作用的补偿,提示存在富含增强子的转录中枢。然而,去除H3K4me1和H3K27ac加上额外的HMs通常表现出高性能的退化,而不去除H3K36me3,这表明HMs标记增强子的整体重要性。gydF4y2Ba
在这个问题设置中,代表转录延长的特征的重要性似乎特别重要,因为该模型被训练为预测RNA-seq测量的mRNA的稳态水平。mRNA的稳态水平不仅由转录起始决定,还由多种因素决定,包括转录延伸率和mRNA的稳定性。根据一项比较用于基因表达预测任务的不同测量技术的研究gydF4y2Ba26gydF4y2Ba通过基因表达帽分析(CAGE)预测表达水平被证明比预测基于rna序列的表达水平更容易。这项研究还表明,H3K36me3可以预测rna -seq的表达水平,而包括H3K4me3在内的核心启动子HMs对CAGE测量更有用。他们暗示隐藏的因素,包括转录延长的有效性,存在于RNA-seq测量中,并且它们不能简单地仅用核心启动子特征来解释。因此,我们推测Chromoformer模型的优越性能可能来自于能够模拟转录延伸率,转录延伸率在基因体中以H3K36me3的形式留下痕迹。这些结果,基于Embedding transformer的高度可解释性,总体上表明Embedding transformer了解了决定TSS附近转录活性的组蛋白编码与代表基因体,特别是TSS下游4 ~ 6 kbp基因体转录伸长的高水平H3K36me3之间的远距离相关性。此外,这在一定程度上解释了为什么Chromoformer的性能随着窗口大小的增加而持续增加,因为该模型还可以从基因体中收集到基因表达的额外证据。gydF4y2Ba
分析gydF4y2Ba独联体gydF4y2Ba-regulome通过gydF4y2Ba独联体gydF4y2Ba- Chromoformer预测的监管影响gydF4y2Ba
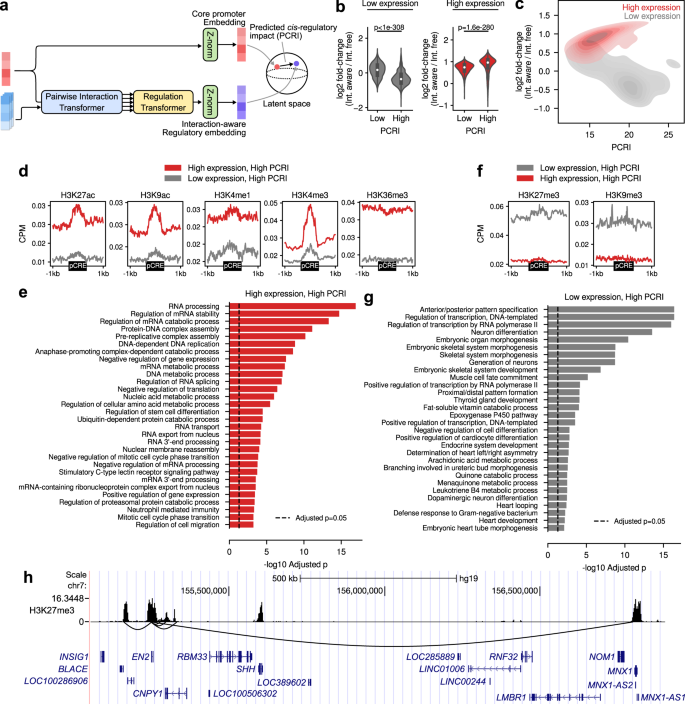
然后我们检查了建模的效果gydF4y2Ba独联体gydF4y2Ba-通过远端pcre的详细调节。由于已经表明,包含成对相互作用和调节变压器导致更好的整体性能,我们寻求对调节相互作用建模的基因水平效应的更详细的解释。为此,我们设计了一种基因水平的测量方法来量化基因突变的预测影响gydF4y2Ba独联体gydF4y2Ba-基于Chromoformer学习的潜在表示的调节。具体而言,我们测量了由Pairwise Interaction and Regulation变压器产生的相互作用感知调控嵌入与由embedding变压器产生的原始核心启动子嵌入之间的欧氏距离(图2)。gydF4y2Ba5gydF4y2Ba).为了方便起见,我们把这个量称为“预测量”gydF4y2Ba独联体gydF4y2Ba-监管影响(PCRI)”。gydF4y2Ba
一个gydF4y2Ba说明PCRI计算的示意图。gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2BaH1细胞中有互作意识和无互作的Chromoformer-reg预测基因表达的log2 fold-change与PCRI的关系(表观基因组标识符E003)。低PCRI组和高PCRI组按PCRI中位数进行划分。在gydF4y2BabgydF4y2Ba,gydF4y2BapgydF4y2Ba- PCRI中位数以下基因的双侧Wilcoxon秩和检验值(gydF4y2BangydF4y2Ba低表达组和高表达组均为4739)和中位数以上的PCRI (gydF4y2BangydF4y2Ba低表达组为4738,高表达组为4739)。在violinplot内的方框中,白点表示中位数,上下方框界限表示上四分位数和下四分位数。gydF4y2BadgydF4y2Ba平均HM信号在pcrs附近,与高表达基因和低表达基因中PCRI最高的前1000个基因相互作用。gydF4y2BaegydF4y2BaH1细胞高表达基因中PCRI最高的前1000个基因的功能富集分析Benjamini-Hochberg调整了Fisher的精确值gydF4y2BapgydF4y2Ba-值以负对数刻度表示。gydF4y2BafgydF4y2Ba平均HM信号在pcrs附近,与高表达基因和低表达基因中PCRI最高的前1000个基因相互作用。gydF4y2BaggydF4y2BaH1细胞低表达基因中PCRI最高的前1000个基因的功能富集分析Benjamini-Hochberg调整了Fisher的精确值gydF4y2BapgydF4y2Ba-值以负对数刻度表示。gydF4y2BahgydF4y2Ba有代表性的基因组区表现为抑制gydF4y2Ba独联体gydF4y2Ba-监管相互作用gydF4y2BaEN2gydF4y2Ba.H3K27me3信号轨迹下方的黑色曲线显示了以核心启动子为中心的三维染色质相互作用gydF4y2BaEN2gydF4y2Ba.NCBI RefSeq基因注释显示在底部。源数据作为源数据文件提供。gydF4y2Ba
为了确保PCRI真正反映潜在向量表示的动态以及调控相互作用的影响,我们首先询问它最终如何影响预测结果概率。测量每个基因在相互作用敏感的Chromoformer-reg和不相互作用的Chromoformer-reg之间预测基因表达的折叠变化,以确定对预测概率的扰动量。因此,在H1细胞的低表达组和高表达组中,PCRI在一半以上和一半以下的两组基因的折叠变化有显著差异(E003)(图。gydF4y2Ba5 bgydF4y2Ba).同样,高表达基因的PCRI与fold-change呈正相关(Spearman 's r = 0.39,gydF4y2BapgydF4y2Ba< 10gydF4y2Ba−308gydF4y2Ba)与低表达基因的PCRI和fold-change呈负相关(Spearman 's r =−0.54,gydF4y2BapgydF4y2Ba< 10gydF4y2Ba−308gydF4y2Ba)(图。gydF4y2Ba5度gydF4y2Ba).综上所述,高表达基因的高PCRI使这些基因的预测表达更高,低表达基因的高PCRI导致预测表达更低。gydF4y2Ba
为了更深入地了解Chromoformer模型如何准确地识别与基因表达的激活或抑制相关的pCREs,我们分析了在PCRI方面分配给最高极端基因的pCREs的特征,即预测远端影响最大的基因gydF4y2Ba独联体gydF4y2Ba监管。我们从4倍CV的每一倍中收集了250个PCRI值最高的高表达基因,并检测了与这1000个基因相关的PCRI附近的HMs的平均信号。结果,与低表达基因相比,具有高PCRI的高表达基因的pcrs平均显示与转录激活相关的HMs水平增加(图2)。gydF4y2Ba5 dgydF4y2Ba,为H1细胞)。特别是,HMs代表增强子(H3K27ac、H3K9ac和H3K4me1)、活性启动子(H3K4me3和H3K9ac)和活性基因体(H3K36me3)被富集。这种与最大的转录激活相关的基因组元素的广泛富集意味着显色细胞学习了转录工厂的存在,活性基因和增强子聚集在一起以进行有效的转录gydF4y2Ba27gydF4y2Ba.基于这一观察,我们通过检查那些聚集在假定的转录工厂的基因是否表现出特定生物功能的富集来寻找额外的生物学证据。有趣的是,它们在家务活动中高度富集,包括mRNA剪接、DNA复制、核糖体生物发生和DNA损伤反应(图2)。gydF4y2Ba5 egydF4y2Ba).我们还观察到细胞类型特异性功能的富集,如干细胞(E003和E016)和永生化细胞系(E114, E116和E118)中的端粒维持,间充质干细胞(E006)中的细胞形态发生和细胞外结构组织,肝细胞(E066)和肝细胞癌(E118)中的铁稳态(补充图)。gydF4y2Ba11gydF4y2Ba).综上所述,可以推测Chromoformer反映了细胞倾向于通过将必需基因隔离在包含多个增强子的转录活性亚室中来确保其功能和生存的重要基因的稳健表达gydF4y2Ba28gydF4y2Ba.另一方面,高PCRI低表达基因的pcrs平均表现出H3K27me3和H3K9me3等抑制标记水平的增加(图2)。gydF4y2Ba5 fgydF4y2Ba),这意味着Chromoformer也检测到抑制性调控信息从pcre转移到核心启动子。我们推测这些pcre代表转录沉默子,因为之前的研究已经显示了远端h3k27me3丰富区域在转录抑制中的潜在功能gydF4y2Ba29gydF4y2Ba.预测有较强抑制的前1000个基因gydF4y2Ba独联体gydF4y2Ba-调控对发育功能表现出极大的富集(图;gydF4y2Ba5克gydF4y2Ba).抑制性的一个典型例子gydF4y2Ba独联体gydF4y2Ba-调控相互作用如图所示(图;gydF4y2Ba5 hgydF4y2Ba)适用于Engrailed Homeobox 2 (gydF4y2BaEN2gydF4y2Ba).正如预期的那样,许多pcre显示出高H3K27me3信号。值得注意的是,其中一个pcre位于1.5 Mbp之外gydF4y2BaEN2gydF4y2Ba, pCRE跨越运动神经元和胰腺Homeobox 1的核心启动子(gydF4y2BaMNX1gydF4y2Ba),这是另一个与发育相关的同源盒转录因子。这两个相距遥远但相互作用的基因之间的功能相似性意味着沉默中枢的存在,在那里发育基因和沉默基因通过3D染色质折叠被隔离在一起。gydF4y2Ba
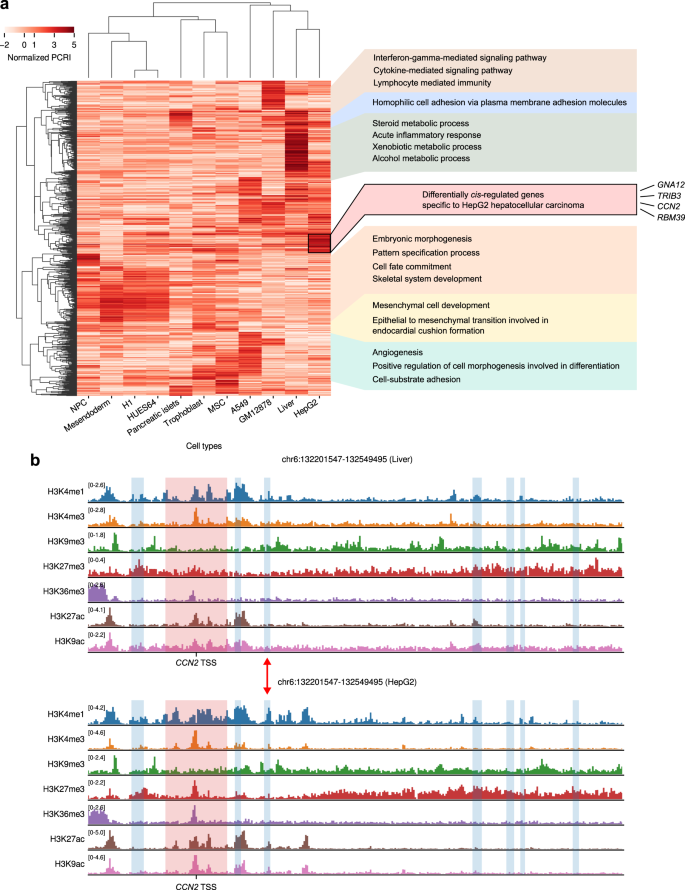
假设整个PCRI值的集合可以看作是一个gydF4y2Ba独联体gydF4y2Ba-规则,我们进一步询问我们是否可以检测到差异gydF4y2Ba独联体gydF4y2Ba-细胞类型之间的调节,并从组蛋白编码的角度揭示其潜在基础。我们选择了在本研究中使用的11种细胞类型中标准化PCRI(方法)方差最高的前1000个基因(补充图)。gydF4y2Ba12gydF4y2Ba),并根据其PCRI值进行分层聚类。正如预期的那样,相似的细胞类型聚集在一起,相应的细胞类型特异性功能被高pcr值突出(图。gydF4y2Ba6gydF4y2Ba).值得注意的是,我们观察到gydF4y2Ba独联体gydF4y2Ba-健康肝组织和HepG2 HCC细胞的规则紧密聚集在一起,但也发现一小部分基因受到HepG2特异性的影响gydF4y2Ba独联体gydF4y2Ba监管(无花果。gydF4y2Ba6gydF4y2Ba,黑匣子)。在分析中,我们没有发现任何与其他聚类基因集不同的显著丰富的生物学术语,因此可以推测,它们代表了癌症特异性异常顺式调控以随机方式发生的结果。为了支持这一观点,我们可以确定四个单独的基因(gydF4y2BaGNA12gydF4y2Ba,gydF4y2BaTRIB3gydF4y2Ba,gydF4y2BaCCN2gydF4y2Ba而且gydF4y2BaRBM39gydF4y2Ba)与HCC密切相关gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,这可以被认为是异常顺式调控的表观遗传命中。这四种基因在HCC中的表达分别比健康肝细胞高9.3-、6.0-、4.1-和3.7倍,与pcr的趋势一致(补充图。gydF4y2Ba13gydF4y2Ba).为了解释为什么显色仪预测这些基因的高PCRI值,我们可视化地检查了基因周围的组蛋白修饰景观。例如,Fig。gydF4y2Ba6 bgydF4y2Ba显示周围组蛋白修饰的景观gydF4y2BaCCN2gydF4y2Ba在健康的肝脏和HCC细胞中比较两种图像显示了一个假定的仅在HCC中活跃的增强子区域(图2)。gydF4y2Ba6 bgydF4y2Ba(红色箭头),这可能解释了较高的PCRI以及较高的表达gydF4y2BaCCN2gydF4y2Ba在肝细胞癌。强调以差分顺调节组分析的形式对Chromoformer模型预测进行深入解释,可以揭示恶性基因表达的表观基因组起源。gydF4y2Ba
显色原蛋白在转录工厂学习加性转录激活,在多梳结合沉默枢纽学习开关样抑制gydF4y2Ba
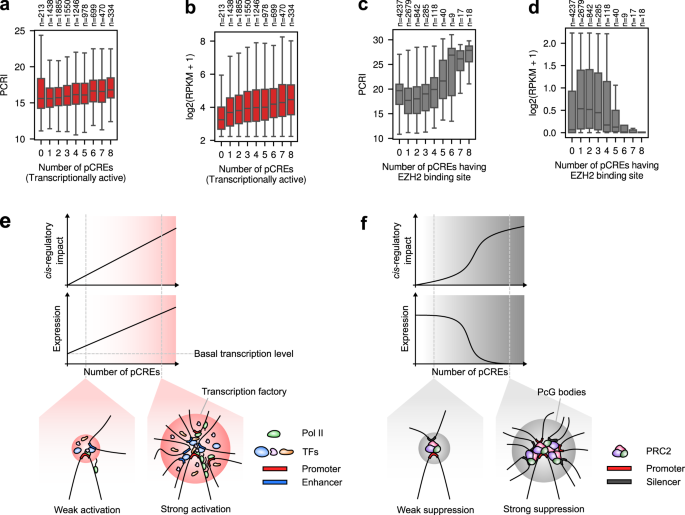
为了评估两个调控中心,即转录工厂和沉默中心的数量特征,我们分析了与转录激活或抑制相关的PCRIs水平和pcrs数量之间的相关性。首先,为了定义整个基因组的转录活性区域,我们利用了路线图表观基因组学项目中提供的基于chrommm的染色质状态注释。在18个状态中,代表活跃的TSS、活跃的基因体或增强子的基因组间隔被认为是活跃区域,我们为每个基因计算与识别的活跃区域重叠的pCREs的数量。我们发现转录活性pcrs的数量与PCRI呈中度但显著的正相关(Spearman 's r = 0.15,gydF4y2BapgydF4y2Ba= 1.3 × 10gydF4y2Ba−42gydF4y2Ba)在H1细胞中(图;gydF4y2Ba7一个gydF4y2Ba).由于基因的表达水平也随着转录活性pCREs数量的增加而增加(图。gydF4y2Ba7 bgydF4y2Ba),这意味着Chromoformer学习了转录工厂中pcre的加性动力学,以激活基因表达(图。gydF4y2Ba7 egydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba转录活性pcrs的数量与PCRI之间的关联。gydF4y2BabgydF4y2Ba转录活性pCREs的数量与基因表达水平之间的关联。gydF4y2BacgydF4y2Ba携带EZH2结合位点的pCREs数量与PCRI之间的关系。gydF4y2BadgydF4y2Ba含有EZH2结合位点的pCREs数量与基因表达水平之间的关系。在整个a-d中,具有相应数量的pCREs的基因的数量显示在图的上方。在箱形图中,中心线表示中位数,上下框限表示上下四分位数,胡须表示1.5×四分位数范围。gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba的监管动态所提出的假设模型的插图gydF4y2BaegydF4y2Ba转录工厂及gydF4y2BafgydF4y2Ba静音中心显示。Pol II, RNA聚合酶II;PcG体、Polycomb群体;Polycomb抑制性复合体2;源数据作为源数据文件提供。gydF4y2Ba
对于沉默中枢,我们认为与Polycomb基团(PcG)蛋白相关的pcre是功能性沉默剂。除了作为一种靶向H3K27的组蛋白甲基转移酶的酶促作用外,最近已经证明Polycomb抑制性复合体2 (PRC2)作为抑制性的中介gydF4y2Ba独联体gydF4y2Ba-调节沉默剂和发育启动子之间的相互作用,促进PcG体的形成gydF4y2Ba35gydF4y2Ba.为了确定PRC2结合消声器的数量,我们利用了来自ENCODE的PRC2三个核心亚基中的两个,即EZH2和SUZ12的ChIP-seq峰。因此,我们发现随着H1细胞中与EZH2结合位点重叠的PCRI数量的增加,PCRI呈逻辑增长(图2)。gydF4y2Ba7 cgydF4y2Ba, d),这表明开关样转录抑制(图;gydF4y2Ba7 fgydF4y2Ba)由prc2介导的沉默中枢由Chromoformer学习。SUZ12结合也可以得到类似的结果(补充图。gydF4y2Ba14 a、bgydF4y2Ba).值得注意的是,当测试与PRC2无关的非特异性pcre数量时,这一趋势完全消失。gydF4y2Ba14摄氏度gydF4y2Ba),这表明prc2介导的沉默具有高度特异性。我们进一步询问,除了H1之外,在哪些细胞类型中观察到prc2介导的沉默的开关样动态。因此,我们在中胚层、神经祖细胞和HUES64细胞中发现了这种模式(补充图。gydF4y2Ba15gydF4y2Ba).有趣的是,这些细胞类型已经被证明具有密切的相似性,当各种细胞类型基于它们的H3K27me3标记在被抑制的Polycomb染色质状态下聚集在一起时gydF4y2Ba23gydF4y2Ba.因此,我们的研究结果强调了1D表观基因组状态的相似性,特别是与polycomb相关的H3K27me3标记,可以通过功能影响的相似性来概括gydF4y2Ba独联体gydF4y2Ba交互。我们还检查了与Polycomb抑制性复合体1 (PRC1)相关的pcre是否具有不同的基因沉默模式,并观察到相似的模式(补充图)。gydF4y2Ba16gydF4y2Ba).这可能反映了PRC1和PRC2在Polycomb响应元件定位中的协同性gydF4y2Ba36gydF4y2Ba,但我们不能排除该模型遗漏了一些受PRC1-或prc2特异性影响的基因的可能性gydF4y2Ba独联体gydF4y2Ba沉默。gydF4y2Ba
迄今为止,集体效应远端gydF4y2Ba独联体gydF4y2Ba基因表达的调控元素仍然不完全清楚,但尽管如此,开拓性的工作利用现代技术,如STARR-seqgydF4y2Ba37gydF4y2Ba或CRISPRi-FlowFISHgydF4y2Ba38gydF4y2Ba当然,我们可以深入了解它们的动态。有趣的是,从训练过的Chromoformer模型的解释中得出的观察结果,该模型经过优化以捕获的定量特征gydF4y2Ba独联体gydF4y2Ba-监管,高度认同这类研究的最新观点。我们对活性pCREs的加性转录激活的观察,总结了之前关于增强子活性定量表征的研究结果gydF4y2Ba果蝇gydF4y2Ba.这种可加性的潜在机制可以用交互中枢或启动子竞争模型来解释gydF4y2Ba39gydF4y2Ba.前者假设启动子与多个具有独立贡献的增强子之间存在多途径相互作用,而后者假设启动子与增强子之间存在一对一的相互作用,并证明启动子与任何增强子之间的接触概率随着候选增强子数量的增加而增加。相反,PcG体的转录沉默的数量性质与PcG结合的沉默子的数量还没有完全被描述。gydF4y2Ba
我们对显色剂的解释导致假设存在一定的局部浓度阈值,以使PcG体充分发挥其抑制功能(图2)。gydF4y2Ba7 cgydF4y2Ba).这可能与其他抑制性表观遗传因子的协同作用有关,包括PcGs新加入的HMs和其他染色质重塑因子诱导的DNA甲基化。无论如何,这一假设的实验验证和决定pcg介导的基因沉默临界点的生物学因素的进一步表征将极大地提高我们对基因表达精确调控的理解。总之,这些结果证明了Chromoformer和深度学习模型在计算生物学领域的新定量假设推导中的效用,最终将促进实验验证,从而获得新的科学发现。gydF4y2Ba
讨论gydF4y2Ba
在本研究中,我们提出了一个名为Chromoformer的基于变压器的深度学习架构,以模拟组蛋白编码在基因表达调控中的定量作用。显色仪通过建立基因的三级层次模型,大大提高了基因表达预测的性能gydF4y2Ba独联体gydF4y2Ba-涉及核心启动子和pcre的监管。通过对自我注意权重、潜在嵌入动力学和几个特征消融研究的分析,我们还对变色模型的行为提供了深入的生物学解释。由于转换器能够理解序列中的遥远依赖关系,Chromoformer可以成功地学习关注基因体内与基因表达相关的HMs在高表达和低表达基因之间最具区别的特定区域。有趣的是,对基因体的关注程度取决于TSS的表观遗传背景,这意味着显色器模型捕获了放置在TSS和基因体上的HMs的遥远依赖性。另一方面,通过使用变压器对无序特征集中的成对关系进行建模,Chromoformer可以了解组蛋白编码介导的信息如何通过三维染色质折叠从pcre传播到核心启动子以调节基因表达。对该模型学习的组蛋白编码的潜在表达的分析强调,管家基因和细胞类型特异性基因的表达通过与增强子的相互作用得到加强,而发育基因的表达主要通过与prc2结合的沉默子相互作用而受到抑制。gydF4y2Ba
我们明确地使用预编译的3D染色质相互作用知识来指导Chromoformer学习。通过显式地将这些实验测量的相互作用频率注入到自我注意评分矩阵中,用于优先考虑将参与模型训练的pcre。然而,似乎也可以仅从基因组序列信息推断pcre和核心启动子之间的相互作用频率。这是因为的特异性gydF4y2Ba独联体gydF4y2Ba-调控相互作用在很大程度上取决于DNA结合蛋白对DNA序列基序的识别,包括转录因子或ccctc结合因子(CTCFs),其功能是隔离3D基因组构象的绝缘体。因此,这些嵌入在基因组中的结合基序可以作为隐藏的词汇表,允许仅根据DNA序列推断所需的染色质构象。来自最近命名为Enformer的模型的结果强烈支持,当使用更广泛的序列信息时,这种从头排序的pcre更有效gydF4y2Ba40gydF4y2Ba,因此,通过使用变压器架构整合基因组和表观基因组特征,为基因表达调控的完全数据驱动建模提供了令人兴奋的可能性。我们将这种基于变压器的多组学集成作为进一步的工作。gydF4y2Ba
嵌入转换器从活跃的TSS跳到基因体的注意力表明,放置在基因体上的HMs在预测稳态基因表达水平时确实是有用的,如果不是最关键的信息的话。从这一结果中,我们可以考虑使用分布在单个基因中的组蛋白编码的整个图谱可能进一步提高稳态mRNA水平的预测准确性。此外,由于H3K36me3在外显子上的富集程度远高于内含子,利用全长基因注释将是另一种有效的模型训练指导方法。由于基因长度和外显子-内含子分布显示出巨大的变异性,我们需要一些巧妙的生物学先验知识的表示。同样,转换器架构将是最强大的选择之一,因为可以灵活地应用掩码来处理变长输入,也可以扩展位置编码以形成复合编码,同时包含基因组位置信息和基因结构注释。gydF4y2Ba
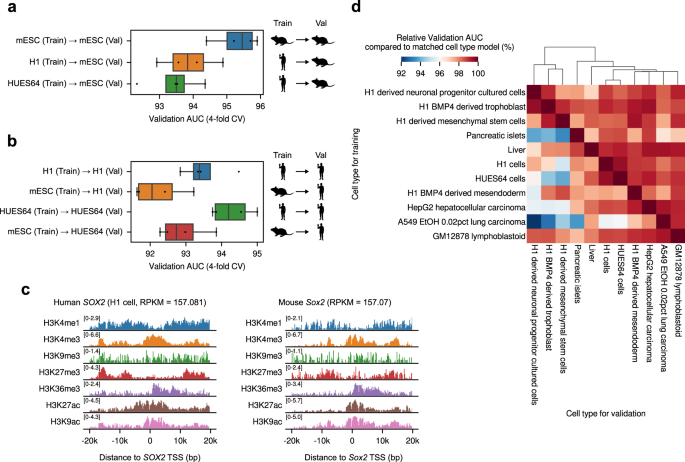
针对Chromoformer模型提出的训练方案是高度可扩展的。例如,我们展示了Chromoformer模型可以使用相关组蛋白ChIP-seq训练来自人类以外物种的细胞类型,即小鼠胚胎干细胞gydF4y2Ba24gydF4y2Ba和Hi-C剖面gydF4y2Ba41gydF4y2Ba,两种间组蛋白编码语法的整体相似性通过跨种预测性能来证明(图;gydF4y2Ba8 a - cgydF4y2Ba).跨细胞类型预测实验也表明,在一种细胞类型上训练的Chromoformer模型在一定程度上仍然适用于其他细胞类型(相对验证AUC > 92%),相似细胞类型的交叉预测性能更高(图2)。gydF4y2Ba8 dgydF4y2Ba).这意味着,以细胞类型特异性方式训练的Chromoformer不仅学习了基因调控的细胞类型特异性特征,而且仍然捕获了可普遍应用于其他细胞类型的一般规则。显色训练可以扩展到包含任何额外的表观基因组特征,如果它可以表示为全基因组信号值的数组。这些特征包括转录因子ChIP-seq信号或用于隔间识别的第一主成分(PC1)信号。如前所述,CTCF结合是3D基因组结构的关键决定因素,启动子-近端CTCF结合也在通过远端增强子-启动子相互作用的基因激活中得到了强调gydF4y2Ba42gydF4y2Ba,如补充图所概括。gydF4y2Ba17一个gydF4y2Ba.我们可以证明,加入CTCF后,Chromoformer-clf的性能得到了边际但一致的提高(补充图。gydF4y2Ba17 bgydF4y2Ba),而Embedding transformer-only Chromoformer-clf模型的增幅更大(补充图17c)。另一方面,将细胞类型特异性的基因组区隔化状态告知ChromoformergydF4y2Ba43gydF4y2Ba使用PC1值作为附加特征并没有导致显著的整体性能增益(补充图。gydF4y2Ba18一个gydF4y2Ba).即使区隔化与基因表达水平相关(补充图。gydF4y2Ba18 b, cgydF4y2Ba),由于关联的绝对水平(Pearson 's correlation coefficient 0.12-0.19)不够大,我们认为区区水平特征的预测能力并没有超过基因水平的HM特征。gydF4y2Ba
a、bgydF4y2BaChromoformer-clf模型的跨种预测性能。gydF4y2Ba一个gydF4y2Ba交叉验证(gydF4y2BangydF4y2Ba= 4)用es -布鲁斯4小鼠胚胎干细胞(mESC)或人胚胎干细胞(hESCs)训练的Chromoformer-clf模型的性能显示用于预测(gydF4y2Ba一个gydF4y2Ba) mESC基因表达及(gydF4y2BabgydF4y2Ba) hESC基因表达。在箱线图中,中心线表示中位数,上下箱限表示上下四分位数,胡须表示1.5×四分位数范围。c人体周围log2转化组蛋白修饰信号gydF4y2BaSOX2gydF4y2BaTSS和鼠标gydF4y2BaSox2gydF4y2BaTSS。gydF4y2BadgydF4y2Ba跨细胞型基因表达预测性能。颜色表示与匹配的细胞类型Chromoformer-clf模型相比的相对验证AUC(即针对相同的细胞类型进行训练和评估)。瓦尔,验证。源数据作为源数据文件提供。gydF4y2Ba
总之,Chromoformer是另一个范例应用,强调了变压器架构在生物序列建模中的巨大潜力。该研究还强调了开发有效嵌入生物先验知识的专业深度学习架构的重要性,不仅可以提高预测任务的性能,还可以定量表征生物实体之间的复杂关系。gydF4y2Ba
方法gydF4y2Ba
启动子中心的三维染色质相互作用gydF4y2Ba
实验验证的核心启动子- pcre相互作用对信息需要训练显色模型。在这项研究中,我们使用了存放在3DIV数据库中的公开数据gydF4y2Ba17gydF4y2Ba以启动子为中心的长距离染色质相互作用,由针对各种组织类型的pcHi-C实验综合编制。HindIII酶切片段的中位长度为4797 bp,平均长度为5640 bp。我们还可以从数据库中获得DNA片段之间的归一化相互作用频率。获得的频率已经经过两步归一化处理,这说明了可捕获性和依赖于背景信号的距离。虽然可以根据每个相互作用提供的估计FDR值选择显著相互作用,但如果归一化频率大于1.5,我们将相互作用视为显著相互作用,以提高显色仪训练期间染色质相互作用的敏感性。注意,归一化频率为1.5表示相互作用和背景信号之间的比值为1.5。gydF4y2Ba
培训数据准备gydF4y2Ba
7个主要HMs (H3K4me1, H3K4me3, H3K9me3, H3K27me3, H3K36me3, H3K27ac和H3K9ac)的巩固ChIP-seq读取比对从路线图表观基因组计划中获得gydF4y2Ba23gydF4y2BatagAlign格式。这种对齐数据可以形成一个高度同质的训练数据集,因为读取被截断到36 bp,以减少由于测序实验差异引起的读取长度偏差,并且它们被下采样到最多3000万个读取,以均匀化读取深度。在使用Sambamba v0.6.8对对齐进行排序和索引之后gydF4y2Ba44gydF4y2Ba,沿着hg19参考基因组计算每个碱基位置的读取深度gydF4y2BagenomecovgydF4y2Ba命令gydF4y2Ba45gydF4y2Ba.对于每个给定的核心启动子或pcre,我们计算了7个HMs的log2转换binned平均信号,沿着大小为100bp、500bp和2 kbp的完全覆盖区域的非重叠基因组窗口,并将这些值作为我们模型的输入特征。由于pCRE的大小在我们的设置中不是固定的,我们用零填充的pCRE特征矩阵使它们的大小与核心启动子特征矩阵的大小一致。具体来说,我们将矩阵居中对齐,并将适当大小的零矩阵附加到输入矩阵的左侧和右侧。为了确定预测目标标签,还从路线图表观基因组学项目下载了标准化基因表达水平(RPKM)。使用RefSeq注释来确定每个基因的TSS。总共选择了18,955个适当注释并有表达测量的基因进行模型训练和评估。整个数据采集和预处理管道在蛇制工作流管理系统v6.5.3中实现gydF4y2Ba46gydF4y2Ba.gydF4y2Ba
三个显色变异体(显色变异体clf、显色变异体reg和显色变异体diff)的基因预测目标如下:最简单的是,log2转换的RPKM值用于Chromoformer-reg训练。对于Chromoformer-diff训练,我们使用两种细胞类型之间RPKM值的log2 fold-change作为预测目标。为了获得用于Chromoformer-clf训练的二进制目标标签,我们将每种细胞类型中所有基因的中位数表达值作为阈值,分配具有高(1)或低表达(0)两种标签之一的基因。换句话说,表达高于中位数的基因被分配为标签“1”,其他基因被分配为标签“0”。这种基因表达的二元分类公式已被广泛应用于各种用于基因表达建模的机器学习方法。请注意,这些标签(“1”和“0”)没有数量意义,而只是表示二进制分类标签的序数索引。即“0”表示该基因分配给第一类,“1”表示该基因分配给第二类。gydF4y2Ba
选择用于模型训练的细胞类型gydF4y2Ba
对于Chromoformer-clf和Chromoformer-reg模型训练,我们只选择了路线图表观基因组学项目中分析的细胞类型的子集,这些细胞类型的基因表达、HMs和3D染色质相互作用的整体概况是可用的。由于我们使用的3D染色质相互作用配置文件不是路线图表观基因组学的官方结果,而是从独立来源获得的,因此我们手动匹配了3DIV数据库中的表观基因组id (eid)和细胞类型助记符gydF4y2Ba17gydF4y2Ba.结果,以下11种细胞类型被选择用于嗜色原细胞训练:H1细胞(E003, H1), H1 BMP4来源的中胚层细胞(E004, ME), H1 BMP4来源的滋养细胞(E005, TB), H1来源的间充质干细胞(E006, MSC), H1来源的神经元祖细胞培养细胞(E007, NPC), HUES64细胞(E016, H1),肝脏(E066, LI11),胰岛(E087, PA), A549 EtOH 0.02pct肺癌(E114, LG), GM12878淋巴母细胞(E116, GM)和HepG2肝细胞癌(E118, LI11)。gydF4y2Ba
显色器模型结构gydF4y2Ba
显色仪由基于变压器编码器的三个模块组成:嵌入、成对交互和调节变压器。gydF4y2Ba
嵌入变压器有一个编码器层,该编码器层取一个二进制平均信号矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{\ rm输入}}}}\)gydF4y2Ba并将其总结为核心启动子嵌入矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba它由固定大小的潜在嵌入向量组成。之前gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{输入 }}}}}}}}\)gydF4y2Ba是馈送进模块的,七维的输入特征为每个gydF4y2Ba\ (n \)gydF4y2Ba容器首先线性投影到的维度gydF4y2Ba\ ({d }_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba(= 128),然后是位置编码矩阵gydF4y2Ba\({{{{{{{\ 男朋友{P }}}}}}}}\)gydF4y2Ba的维度gydF4y2Ba\ (n \ * {d }_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba以元素的方式添加到相同维度的输入特征矩阵中。gydF4y2Ba\({{{{{{{{\ 男朋友{P }}}}}}}}}_{{ ij}} \)gydF4y2Ba定义如下。gydF4y2Ba
值得注意的是,任何两行向量之间的内积(例如,gydF4y2Ba\ \ ()gydF4y2Bath和gydF4y2Ba\ (b \)gydF4y2Ba-th行)的位置编码矩阵gydF4y2Ba\({{{{{{{\ 男朋友{P }}}}}}}}\)gydF4y2Ba,即gydF4y2Ba\({{{{{{{{\ 男朋友{P }}}}}}}}}_{ } \)gydF4y2Ba而且gydF4y2Ba\({{{{{{{{\ 男朋友{P }}}}}}}}}_{ b} \)gydF4y2Ba,只取决于位置距离gydF4y2Ba\ ({|} - {b |} \)gydF4y2Ba在两个向量之间。因此,通过在输入特征矩阵中添加位置编码矩阵,可以在接下来的多头注意层中识别任意两个特征之间的相对距离。embed transformer中的多头注意层利用自注意机制来捕获不同位置的HM配置之间的相互依赖性,这些相互依赖性有助于调节基因表达。重要的是,这些操作是针对多个头部分别进行的,因此模型可以捕捉输入特征之间相互依赖的不同方面。变压器架构中的自注意操作是一种特殊的缩放点积注意,其中查询、键和值矩阵起源于相同的特征序列。具体来说,是位置编码的输入维度特征矩阵gydF4y2Ba\ (n \ * {d }_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba是线性投影产生三个矩阵吗gydF4y2Ba\({{{{{{{{\ 男朋友{问 }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba,gydF4y2Ba\({{{{{{{{\ 男朋友{K }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba而且gydF4y2Ba\({{{{{{{{\ 男朋友{V }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba的维度gydF4y2Ba\(n\times {d^{\prime}}_{{{{{{{\rm{emb}}}}}}}}\)gydF4y2Ba,在语义上分别表示查询、键和值矩阵。gydF4y2Ba\ (^ {d {\ ' } }_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba设置为64。的gydF4y2Ba\ (n \ n \)gydF4y2Ba矩阵的乘积gydF4y2Ba\({{{{{{{{\ 男朋友{问 }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba而且gydF4y2Ba\({{{{{{{{\ 男朋友{K }}}}}}}}}_{{{{{{{{\ rm {emb }}}}}}}}}^{ T} \)gydF4y2Ba被称为成对亲和矩阵,因为矩阵的每个元素都等价于对应的一对向量之间的点积gydF4y2Ba\({{{{{{{{\ 男朋友{问 }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba而且gydF4y2Ba\({{{{{{{{\ 男朋友{K }}}}}}}}}_{{{{{{{{\ rm {emb }}}}}}}}}^{ T} \)gydF4y2Ba.它表示输入序列中两个位置之间的亲和量。两两亲和矩阵除以gydF4y2Ba\ \√{{d }_{{{{{{{{\ rm {emb }}}}}}}}}^{{\ '}}} \)gydF4y2Ba运用softmax函数将自注意亲和力转换为每行和为1的权重。价值矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{V }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba与得到的注意权重矩阵相乘,最终得到自注意操作的输出。缩放点积的整个过程可以总结如下:gydF4y2Ba
在嵌入变压器中,上述自注意操作由单独完成gydF4y2Ba\({米 }_{{{{{{{{\ rm {emb }}}}}}}}}\)gydF4y2Ba(= 2)个正面和结果gydF4y2Ba\({米 }_{{{{{{{{\ rm {emb }}}}}}}}}\)gydF4y2Ba维数向量gydF4y2Ba\ ({d }_{{{{{{{{\ rm {emb }}}}}}}}}^{{\ '}} \)gydF4y2Ba连接起来形成一个单一的gydF4y2Ba\ ({d }_{{{{{{{{\ rm {emb }}}}}}}}}^{{\ '}} \ * {m }_{{{{{{{{\ rm {emb }}}}}}}}}={ d }_{{{{{{{{\ rm {emb }}}}}}}}}\)gydF4y2Ba(= 128)维向量,以保持输入特征的维数。随后,通过残差连接添加自我注意前的特征输入序列,并进行层归一化。结果再经过线性投影层变成维数gydF4y2Ba\({\三角洲 }_{{{{{{{{\ rm {emb }}}}}}}}}\)gydF4y2Ba(= 128),非线性激活由整流线性单元(ReLU)和最终线性投影成的维数gydF4y2Ba\ ({d }_{{{{{{{{\ rm {emb }}}}}}}}}\)gydF4y2Ba.这一系列涉及线性投影、非线性激活和另一个线性投影的操作包括一个位置前馈层。gydF4y2Ba
再经过残差连接和层归一化,得到核心启动子嵌入矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba最终产生。gydF4y2Ba
成对交互变压器由两个堆叠层组成,根据其与每个pCRE的成对交互来更新核心启动子嵌入,并生成成对交互嵌入矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba.嵌入转换器中编码器-解码器注意与自注意操作的区别在于,编码器-解码器注意分别构建查询矩阵和键值矩阵。具体来说,查询矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{问 }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba是从gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba(或gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba由第一层产生),而键和值矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{K }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba而且gydF4y2Ba\({{{{{{{{\ 男朋友{V }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba是由位置编码的pCRE HM特征构建的gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{嗯 }}}}}}}}\)gydF4y2Ba.简而言之,编码器-解码器注意的查询、键和值矩阵可以总结如下,其中LinearNoBias表示没有偏差的线性投影函数。gydF4y2Ba
然后在核心启动子查询和pCRE键值之间进行缩放点积注意,如下所示gydF4y2Ba\ (^ {d {{{\ ' } }}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba= 64和gydF4y2Ba\ ({d }_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba= 128:gydF4y2Ba
其余的操作,包括位置前馈层的配合gydF4y2Ba\({\三角洲 }_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba(= 256),残差连接和层归一化与Embedding transformer相同,最终产生成对交互嵌入gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba.gydF4y2Ba
为了避免过多的计算负载,并使训练批处理在训练期间适合单个图形处理单元(GPU)的内存,我们只考虑最多gydF4y2Ba\({我}_{{\马克斯}}\)gydF4y2Ba(= 8)每个核心启动子的pcr。为了确定参与训练的pCRE集合,所有候选pCRE根据其与核心启动子的归一化相互作用频率进行优先排序,因为相互作用频率越高的pCRE可能在预测相应基因的表达方面信息越丰富。gydF4y2Ba
调节变压器由六层堆叠组成,具有门控自注意机制。调节变压器的关键功能是更新gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba连同整套的gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba的同时,最终产生监管嵌入gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{注册 }}}}}}}}\)gydF4y2Ba.为此,提取出精确表示相关TSS所在基因组仓的单个嵌入向量gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm {emb }}}}}}}}\)gydF4y2Ba而且gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{一对 }}}}}}}}\)gydF4y2Ba的年代。然后,将它们并排连接,形成一个复合输入矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{排版 }}}}}}}}\)gydF4y2Ba的维度gydF4y2Ba\ \离开({我}_{{{\马克斯}}}+ 1 \)\ * {d }_{{{{{{{{\ mathrm {emb }}}}}}}}}\)gydF4y2Ba(回想一下,gydF4y2Ba\ ({d }_{{{{{{{\ rm {emb }}}}}}}}={ d }_{{{{{{{{\ rm{一对 }}}}}}}}}\)gydF4y2Ba= 128)。具体来说,它们是在嵌入矩阵中点的向量。注意对于小于的基因gydF4y2Ba\({我}_{{{\马克斯}}}\)gydF4y2Ba顺式-监管互动,剩下的gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{排版 }}}}}}}}\)gydF4y2Ba用虚拟零向量填充。规则转换器不需要位置编码,因为它不假定嵌入之间的任何预定义顺序。我们只确定复合输入矩阵的第一行向量是核心启动子嵌入。这个无序的嵌入集被馈送到一个门控的自注意机制,以允许模型决定它将在多大程度上积极利用携带交互信息的转换嵌入。除了查询、键和值矩阵之外,门控自注意还引入了门矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{G }}}}}}}}}_{{{{{{{\ rm{注册 }}}}}}}}\)gydF4y2Ba它学习信息传输的量。这四个gydF4y2Ba\ \离开({我}_{{{\马克斯}}}+ 1 \)\ * {d ^ {\ ' } }_{{{{{{{{\ rm{注册 }}}}}}}}}\)gydF4y2Ba门控自注意操作的矩阵计算如下,其中gydF4y2Ba\ ({d }_{{{{{{{{\ rm{注册 }}}}}}}}}^{{\ '}} \)gydF4y2Ba= 32:gydF4y2Ba
此外,我们增加了一个归一化相互作用频率的向量gydF4y2Ba\({{{{{{{\ 男朋友{f }}}}}}}}\)gydF4y2Ba在相应的核心启动子- pcre对之间作为自注意矩阵的偏置项,以告知模型成对相互作用的相对亲和性。一个gydF4y2Ba\(\左({我}_{{{\马克斯}}}+ 1 \)\ \倍左({我}_{{{\马克斯}}}+ 1 \)\)gydF4y2Ba偏差矩阵gydF4y2Ba\({{{{{{{\ 男朋友{B }}}}}}}}\)gydF4y2Ba的第一行是谁的gydF4y2Ba\({{{{{{{\ 男朋友{f }}}}}}}}\)gydF4y2Ba其他的值都是0。综上所述,调节变压器的注意操作可写成:gydF4y2Ba
在哪里gydF4y2Ba\γ(\ \)gydF4y2Ba,gydF4y2Baσ\ (\ \)gydF4y2Ba而且gydF4y2Ba\(\保监会\)gydF4y2Ba分别表示一个可学习的标量系数、sigmoid函数和Hadamard积。gydF4y2Ba
我们连接了三个gydF4y2Ba\({{{{{{{{\ 男朋友{X }}}}}}}}}_{{{{{{{\ rm{注册 }}}}}}}}\)gydF4y2Ba是由独立模块分别从100 bp、500 bp和2 kbp分辨率的输入中学习得出的。只有连接的矩阵的第一行,它表示gydF4y2Ba独联体gydF4y2Ba提取核心启动子的-调控感知嵌入载体,送入全连接头。在三种不同的Chromoformer模型(Chromoformer-clf, Chromoformer-reg和Chromoformer-diff)中,全连接的头部都有一个128维的ReLU激活隐藏层。Chromoformer-clf的全连接头产生一个二维输出,表示每个二进制表达式标签的两个预测对数,而Chromoformer-reg的全连接头产生一个表示log2转换的基因表达值的单个标量。在Chromoformer-diff中,完全连接的头部被来自每种细胞类型的两个多尺度调控嵌入的级联向量所供养,并产生一个表示基因表达的log2折叠变化的单个标量。此外,Chromoformer-diff采用两个辅助任务预测每种细胞类型log2转化基因表达的绝对水平(补充图。gydF4y2Ba5gydF4y2Ba).所有的Chromoformer变体都是使用PyTorch v1.9.0实现的gydF4y2Ba47gydF4y2Ba.gydF4y2Ba
模型培训与评价gydF4y2Ba
使用AdamW优化器对Chromoformer模型的所有变体进行10个epoch的训练gydF4y2Ba48gydF4y2Ba最后选取上一个纪元的模型作为最终模型。初始学习速率选择为3 × 10gydF4y2Ba−5gydF4y2Ba并且在每个纪元之后都减少了13%,因此在五个纪元之后都可以大约缩小到其值的一半。在Chromoformer-clf中,将预测概率与单热编码二元基因表达标签之间的交叉熵作为损失函数。在Chromoformer-reg和Chromoformer-diff中,使用预测标量与目标值之间的均方误差(MSE)作为损失函数。批大小固定为64。所有基准深度学习模型的实现都是从各自作者提供的官方代码库中获得的。为了训练基准模型,我们应用了之前为每个基准模型确定的最优超参数。gydF4y2Ba
对于GC-MERGE训练,我们需要修改HM信号的输入表示形式和gydF4y2Ba独联体gydF4y2Ba-根据要求进行监管互动。计算整个基因组中每个10 kbp bin的ChIP-seq读取深度gydF4y2BamulticovgydF4y2Ba利用pcHi-C实验结果确定这些10 kbp基因组箱之间的相互作用频率。由于GC-MERGE预测是针对每个10 kbp的箱子进行的,而不是针对每个基因,当同一个箱子中有两个或多个基因时,就会产生歧义。通过在每个箱子中选择一个具有代表性的基因,并将其分配给该箱子中最常出现的标签,可以解决这种模糊性。这是以减少可预测基因的数量为代价的。为了进行尽可能公平的比较,我们通过重新训练Chromoformer模型,使用GC-MERGE可预测表达的相同基因集,对GC-MERGE和Chromoformer进行了4倍CV。gydF4y2Ba
嵌入式变压器自注意分析gydF4y2Ba
每个嵌入变压器有两个独立的注意头,因此产生两个相应的自注意成对亲和矩阵gydF4y2Ba\({{{{{{{{\ 男朋友{问 }}}}}}}}}_{{{{{{{{\ rm {emb }}}}}}}}}{{{{{{{{\ 男朋友{K }}}}}}}}}_{{{{{{{{\ rm {emb }}}}}}}}}^{ T} / \√{{d }_{{{{{{{{\ rm {emb }}}}}}}}}^{{\ '}}} \)gydF4y2Ba对于每个输入。由于整个模型由三个独立的单分辨率模块组成,我们总共可以提取6个自我注意权重矩阵。我们可视化了softmax归一化的成对亲和力,即自我注意权重。gydF4y2Ba4 gydF4y2Ba.注意,所有的自我注意权重都是在验证集的基因推理时获得的。gydF4y2Ba
预测的计算gydF4y2Ba独联体gydF4y2Ba-监管影响、规范化和聚类gydF4y2Ba
为了计算预测的顺式调控影响(PCRI),我们首先将多分辨率核心启动子嵌入定义为由三种不同输入分辨率产生的单个核心启动子嵌入的拼接。然后,将PCRI定义为每个基因的多分辨率核心启动子嵌入与多分辨率调控嵌入之间的欧氏距离。重要的是,我们在计算欧几里得距离之前标准化了每个嵌入向量,以校正由变换本身引起的全局位移。与自注意分析类似,我们对每个验证集中的基因计算了正文中讨论的整个PCRI值,以确保模型没有显式地学习到潜在嵌入所反映的最优转换gydF4y2Ba独联体gydF4y2Ba-对这些基因的调控。gydF4y2Ba
为了根据PCRI值对细胞类型和基因进行聚类,这些值事先进行了标准化(即Z-score标准化)。得到的归一化PCRI值一般在零点附近呈钟形分布(补充图。gydF4y2Ba12gydF4y2Ba).采用PCRI值跨细胞方差最高的前1000个基因进行具有相关相似度的平均连锁层次聚类。gydF4y2Ba
高pcr基因的功能富集分析gydF4y2Ba
对于四个验证折叠中的每一个,我们分别为每个二元标签识别出具有最高pcr值的前250个基因。四个基因集结合的功能富集分析用enrichment r完成gydF4y2Ba49gydF4y2Ba.基因本体生物学过程术语与Benjamini-Hochberg进行了调整gydF4y2BapgydF4y2Ba-值<0.05为显著富集项。gydF4y2Ba
多梳相关pcre的定义gydF4y2Ba
为了定义与Polycomb结合区域相关的pcre,我们使用了在ENCODE中公开的H1细胞中确定的Polycomb亚基的不可复制发现率(IDR)阈值ChIP-seq峰值。具体来说,我们下载了PRC2的EZH2和SUZ12 ChIP-seq峰值,以及PRC1的RNF2和CBX8 ChIP-seq峰值(补充表)gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
小鼠胚胎干细胞数据处理gydF4y2Ba
为了评估Chromoformer对人类以外物种的效用,我们从es - brue4小鼠胚胎干细胞(mESC)系的原始组蛋白ChIP-seq reads中处理了ENCODE参考表观基因组(补充表)gydF4y2Ba2gydF4y2Ba).为了与人类数据一致,处理管道遵循路线图表观基因组计划,如下所述。下载FASTQ文件后,使用bwa v0.7.17-r1188首先将组蛋白ChIP-seq读对mm9参考基因组gydF4y2Ba50gydF4y2Ba.为了标准化读取长度的影响,每个对齐的读取被截断到36 bp。此外,读取深度通过子采样读取对齐到300万个读取进行归一化。处理后的比对转换为全基因组读深度信号gydF4y2Babedtools genomecovgydF4y2Ba.此外,为了确定用于显色器训练的启动子- pcre相互作用,我们使用了公开可用的mESC Hi-C相互作用矩阵的归一化相互作用频率gydF4y2Ba41gydF4y2Ba.es -布鲁斯4细胞的大量RNA-seq基因表达谱也从ENCODE文件ENCFF166EXS中获得[gydF4y2Bahttps://www.encodeproject.org/experiments/ENCSR000CGUgydF4y2Ba].使用Gencode vM1基因注释来确定每个基因的转录位点和启动子区域。gydF4y2Ba
CTCF ChIP-seq数据处理gydF4y2Ba
为了检验在模型训练中包含CTCF信号的效果,我们从ENCODE中获得了原始CTCF ChIP-seq读取。ENCODE中只有5种细胞类型有CTCF ChIP-seq数据:H1细胞(E003)、H1衍生的神经元祖细胞培养细胞(E007)、A549肺癌细胞(E114)、GM12878淋巴母细胞(E116)和HepG2肝细胞癌细胞(E118)(补充表)gydF4y2Ba3.gydF4y2Ba).CTCF ChIP-seq reads的处理方式与mESC组蛋白ChIP-seq reads完全相同(见上文),但使用hg19参考基因组。gydF4y2Ba
报告总结gydF4y2Ba
实验设计的进一步信息可在gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
本研究中使用的Histone ChIP-seq read比对、mRNA表达谱和chromHMM染色质状态从Roadmap Epigenomics Web Portal下载gydF4y2Ba23gydF4y2Ba[gydF4y2Bahttps://egg2.wustl.edu/roadmap/web_portal/index.htmlgydF4y2Ba].以启动子为中心Hi-C实验的归一化相互作用频率来自3DIV数据库hg19 pcHi-C数据的收集[gydF4y2Bahttp://3div.kr/gydF4y2Ba]在组织助记符H1, ME, TB, MSC, NPC, LI11, PA, LG和GM下。TF针对PRC1/2亚基和CTCF的ChIP-seq读取也从ENCODE下载gydF4y2Ba24gydF4y2Ba在补充表格所列的加入编号下gydF4y2Ba1gydF4y2Ba而且gydF4y2Ba3.gydF4y2Ba.TAD和基因组区隔信息(PC1值)从Schmitt等人的补充材料中下载。gydF4y2Ba43gydF4y2Ba原始组蛋白ChIP-seq reads和es -布鲁斯4小鼠胚胎干细胞数据的mRNA表达谱也从ENCODE下载gydF4y2Ba24gydF4y2Ba在补充表所列的编号下gydF4y2Ba2gydF4y2Ba.es -布鲁斯4 mRNA表达谱的接入码为ENCFF166EXS [gydF4y2Bahttps://www.encodeproject.org/experiments/ENCSR000CGUgydF4y2Ba].es -布鲁斯4单元的Hi-C相互作用频率矩阵从Dixon等人的数据库中下载。gydF4y2Ba41gydF4y2BaNCBI RefSeq基因注释从UCSC表格浏览器下载[gydF4y2Bahttps://genome.ucsc.edu/cgi-bin/hgTablesgydF4y2Ba].Gencode vM1基因注释从Gencode [gydF4y2Bahttps://www.gencodegenes.org/mouse/release_M1.htmlgydF4y2Ba].gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
Chromoformer模型的源代码可在GitHub存储库[gydF4y2Bahttps://github.com/dohlee/chromoformergydF4y2Ba]在doi:10.5281/zenodo.7151966gydF4y2Ba51gydF4y2Ba.Chromoformer-clf模型的预训练权重可在Figshare上获得,doi:10.6084/m9.figshare.19424807.v1gydF4y2Ba52gydF4y2Ba.基准模型的代码实现从相应的代码库下载:DeepChrome [gydF4y2Bahttps://github.com/QData/DeepChromegydF4y2Ba], attentivchrome [gydF4y2Bahttps://github.com/QData/AttentiveChromegydF4y2Ba]、DeepDiff [gydF4y2Bahttps://github.com/QData/DeepDiffChromegydF4y2Ba], gc-merge [gydF4y2Bahttps://github.com/rsinghlab/GC-MERGEgydF4y2Ba],以及HM-CRNN [gydF4y2Bahttps://github.com/pptnz/deeply-learning-regulatory-latent-spacegydF4y2Ba].gydF4y2Ba
参考文献gydF4y2Ba
Jenuwein, T. & Allis, C. D.翻译组蛋白密码。gydF4y2Ba科学gydF4y2Ba293gydF4y2Ba, 1074-1080(2001)。gydF4y2Ba
Singh, R, Lanchantin, J., Robins, G. & Qi, Y. DeepChrome:从组蛋白修饰预测基因表达的深度学习。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba, i639-i648(2016)。gydF4y2Ba
Singh, R, Lanchantin, J, Sekhon, A. & Qi, Y.参加和预测:通过染色质的选择性注意了解基因调控。gydF4y2Ba神经导过程系统。gydF4y2Ba30.gydF4y2Ba, 6785-6795(2017)。gydF4y2Ba
Alipanahi, B., Delong, A., Weirauch, m.t. & Frey, B. J.通过深度学习预测DNA和rna结合蛋白的序列特异性。gydF4y2Ba生物科技Nat。》。gydF4y2Ba33gydF4y2Ba, 831-838(2015)。gydF4y2Ba
Jumper, J.等。高度准确的蛋白质结构预测AlphaFold。gydF4y2Ba自然gydF4y2Ba596gydF4y2Ba, 583-589(2021)。gydF4y2Ba
Cho, K.等。使用RNN编码器-解码器学习短语表示,用于统计机器翻译。在gydF4y2Ba2014年自然语言处理经验方法会议论文集gydF4y2Ba.1724 - 1734(2014)。gydF4y2Ba
何克瑞特,史密德胡伯,J.长短期记忆。gydF4y2Ba神经第一版。gydF4y2Ba9gydF4y2Ba, 1735-1780(1997)。gydF4y2Ba
Shlyueva, D., Stampfel, G. & Stark, A.转录增强子:从特性到全基因组预测。gydF4y2BaNat. Rev. GenetgydF4y2Ba15gydF4y2Ba, 272-286(2014)。gydF4y2Ba
高金,梁德华,任斌。三维基因组在转录调控和多能性中的作用。gydF4y2Ba细胞干细胞gydF4y2Ba14gydF4y2Ba, 762-775(2014)。gydF4y2Ba
hamston, N. & Lenhard, B.染色质和远程基因调控的表观遗传特征。gydF4y2Ba核酸测定。gydF4y2Ba41gydF4y2Ba, 7185-7199(2013)。gydF4y2Ba
Lieberman-Aiden, E.等人。对远距离相互作用的全面测绘揭示了人类基因组的折叠原理。gydF4y2Ba科学gydF4y2Ba326gydF4y2Ba, 289-293(2009)。gydF4y2Ba
bess, J., Loinaz, X., Patel, S., Larschan, E. & Singh, R.利用图卷积网络集成远程调控相互作用来预测基因表达。gydF4y2BaJ.计算生物学。gydF4y2Ba29gydF4y2Ba, 409-424(2022)。gydF4y2Ba
瓦斯瓦尼,A.等人。你所需要的就是关注。在gydF4y2Ba神经信息处理系统的进展gydF4y2Ba(2017)。gydF4y2Ba
季勇,周志,刘浩,戴武里,R. V. DNABERT:基于变形模型的dna语言预训练双向编码器表示。gydF4y2Ba生物信息学gydF4y2Ba37gydF4y2Ba, 2112-2120(2021)。gydF4y2Ba
里夫斯等人。生物结构和功能是通过将无监督学习扩展到2.5亿个蛋白质序列而产生的。gydF4y2Ba国家科学院学报美国gydF4y2Ba118gydF4y2Ba, e2016239118(2021)。gydF4y2Ba
Rao, R.等人。MSA变压器。在gydF4y2Ba机器学习国际会议gydF4y2Ba.8844 - 8856(2021)。gydF4y2Ba
荣格等人。人类基因组中以启动子为中心的远程染色质相互作用的概要。gydF4y2BaNat麝猫。gydF4y2Ba51gydF4y2Ba, 1442-1449(2019)。gydF4y2Ba
Bahdanau, D., Cho, K. Bengio, Y.神经机器翻译通过联合学习对齐和翻译。在gydF4y2Ba第三届学习表征国际会议gydF4y2Ba(2015)。gydF4y2Ba
陈德良M.-T。,Ph一个m, H. Manning C. D. Effective approaches to attention-based neural machine translation. In2015年自然语言处理经验方法会议论文集gydF4y2Ba.1412 - 1421(2015)。gydF4y2Ba
林,Z.等。一种结构化的自我注意句子嵌入。在gydF4y2Ba第五届学习表征国际会议gydF4y2Ba(2017)。gydF4y2Ba
Sekhon, A., Singh, R. & Qi, Y. DeepDiff:从组蛋白修饰预测差分基因表达的深度学习。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, i891-i900(2018)。gydF4y2Ba
Kang, M., Lee, S., Lee, D. & Kim, S.基于多注意深度学习的细胞类型特异性基因调控机制研究。gydF4y2Ba麝猫面前gydF4y2Ba11gydF4y2Ba, 869(2020)。gydF4y2Ba
表观基因组学联盟路线图。et al。111个参考人类表观基因组的综合分析。gydF4y2Ba自然gydF4y2Ba518gydF4y2Ba, 317-330(2015)。gydF4y2Ba
Encode项目联盟。人类基因组中DNA元素的综合百科全书。gydF4y2Ba自然gydF4y2Ba489gydF4y2Ba, 57-74(2012)。gydF4y2Ba
Yoh, S. M., Lucas, J. S. & Jones, K. A. Iws1:Spt6:CTD复合物控制共转录mRNA的生物合成和HYPB/ setd2介导的组蛋白H3K36甲基化。gydF4y2BaDev的基因。gydF4y2Ba22gydF4y2Ba, 3422-3434(2008)。gydF4y2Ba
董,等。利用不同细胞环境中的染色质特征建模基因表达。gydF4y2Ba基因组医学杂志。gydF4y2Ba13gydF4y2Ba, 1-17(2012)。gydF4y2Ba
萨瑟兰,H. & Bickmore, W. A.转录工厂:工会中的基因表达?gydF4y2BaNat. Rev. GenetgydF4y2Ba10gydF4y2Ba, 457-466(2009)。gydF4y2Ba
弗兰克尔等人。由明显冗余的转录增强子所赋予的表型鲁棒性。gydF4y2Ba自然gydF4y2Ba466gydF4y2Ba, 490-493(2010)。gydF4y2Ba
蔡,Y.等。富含h3k27me3的基因组区域可以作为沉默剂,通过染色质相互作用抑制基因表达。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 719(2021)。gydF4y2Ba
杨,Y. M.等。Gα12在肝细胞癌中的过表达通过hnf4 α失活降低microRNA-122的表达,从而导致c-Met的诱导。gydF4y2BaOncotargetgydF4y2Ba6gydF4y2Ba, 19555 - 19069(2015)。gydF4y2Ba
徐,C.等。rna结合蛋白39:一个有希望的癌症治疗靶点。gydF4y2Ba细胞死亡迪斯科。gydF4y2Ba7gydF4y2Ba, 214(2021)。gydF4y2Ba
荣德杰,纳,沈勇,纳,李俊伟,CAPER的分子克隆和表征,一种新的激活蛋白-1和雌激素受体的共激活剂。gydF4y2Ba生物。化学。gydF4y2Ba277gydF4y2Ba, 1229-1234(2002)。gydF4y2Ba
王晓军,李芳芳,张玉军,蒋敏,任文华,TRIB3促进肝细胞癌生长,预测不良预后。gydF4y2Ba癌症Biomark。gydF4y2Ba29gydF4y2Ba, 307-315(2020)。gydF4y2Ba
贾强,董强,秦林。CCN:微环境中影响肝细胞癌转移的核心调控蛋白?gydF4y2BaOncotargetgydF4y2Ba7gydF4y2Ba, 1203-1214(2016)。gydF4y2Ba
Ngan, C. Y.等。染色质相互作用分析阐明了prc2结合沉默蛋白在小鼠发育中的作用。gydF4y2BaNat麝猫。gydF4y2Ba52gydF4y2Ba, 264-272(2020)。gydF4y2Ba
卡恩,t.g.等。PRC1和PRC2招募Polycomb响应元素的相互依赖性。gydF4y2Ba核酸测定gydF4y2Ba44gydF4y2Ba, 10132-10149(2016)。gydF4y2Ba
阿诺德,c.d.等人。STARR-seq鉴定的全基因组定量增强子活性图。gydF4y2Ba科学gydF4y2Ba339gydF4y2Ba, 1074-1077(2013)。gydF4y2Ba
Fulco, c.p.等。来自数千个CRISPR扰动的增强子-启动子调控的接触活性模型。gydF4y2BaNat麝猫。gydF4y2Ba51gydF4y2Ba, 1664-1669(2019)。gydF4y2Ba
Schoenfelder, S. & Fraser, P.基因表达控制中的远程增强子-启动子接触。gydF4y2BaNat. Rev. GenetgydF4y2Ba20.gydF4y2Ba, 437-455(2019)。gydF4y2Ba
Avsec, Z.等。通过整合远程相互作用,从序列中有效预测基因表达。gydF4y2BaNat方法。gydF4y2Ba18gydF4y2Ba, 1196-1203(2021)。gydF4y2Ba
狄克逊,J. R.等。通过染色质相互作用分析确定的哺乳动物基因组的拓扑结构域。gydF4y2Ba自然gydF4y2Ba485gydF4y2Ba, 376-380(2012)。gydF4y2Ba
Kubo, N.等。启动子-近端CTCF结合促进远端增强子依赖的基因激活。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba28gydF4y2Ba, 152-161(2021)。gydF4y2Ba
施密特,a.d.等人。染色质接触图的纲要揭示了人类基因组中的空间活跃区域。gydF4y2Ba细胞的代表。gydF4y2Ba17gydF4y2Ba, 2042-2059(2016)。gydF4y2Ba
Tarasov, A., Vilella, A. J., Cuppen, E., Nijman, I. J. & Prins, P. Sambamba: NGS对齐格式的快速处理。gydF4y2Ba生物信息学gydF4y2Ba31gydF4y2Ba, 2032-2034(2015)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
莫尔德,F.等。使用Snakemake进行可持续数据分析。gydF4y2BaF1000ResgydF4y2Ba10gydF4y2Ba, 33(2021)。gydF4y2Ba
Paszke等人。PyTorch:一个命令式的高性能深度学习库。在gydF4y2Ba神经信息处理系统研究进展gydF4y2Ba(2019)。gydF4y2Ba
洛什奇洛夫,I.赫特,F.解耦权衰减正则化。在gydF4y2Ba学习表征国际会议gydF4y2Ba(2019)。gydF4y2Ba
库列绍夫,m.v.等。enrichment:一个全面的基因集富集分析web服务器2016年更新。gydF4y2Ba核酸测定gydF4y2Ba44gydF4y2Ba, w90-w97(2016)。gydF4y2Ba
李,H. & Durbin R.快速和准确的短读对齐与Burrows-Wheeler变换。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 1754-1760(2009)。gydF4y2Ba
Lee, D. Yang, J. Kim S.显色器(1.0版)(Zenodo, 2022);gydF4y2Bahttps://doi.org/10.5281/zenodo.7151966gydF4y2Ba.gydF4y2Ba
李。D., Yang, J. Kim S.预训练显色器权重(Figshare, 2022);gydF4y2Bahttps://doi.org/10.6084/m9.figshare.19424807.v1gydF4y2Ba.gydF4y2Ba
确认gydF4y2Ba
本研究得到了国家研究基金会(NRF)培育新型后基因组产业的协作基因组计划的支持,由科学和信息通信技术部(MSIT)资助(NRF- 2014m3c9a3063541),韩国政府(MSIT)资助的信息与通信技术规划与评估研究所(IITP) [NO. 1]。2021-0-01343,人工智能研究生院计划(首尔国立大学)],以及由科学和信息通信技术部资助的国家研究基金会(NRF)的生物与医疗技术发展计划(NRF- 2019m3e5d3073375和NRF- 2022m3e5f3085677) (S. K.)。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
D.L.、J.Y.和S.K.构思了实验,D.L.和J.Y.进行了实验,D.L.、J.Y.和S.K.分析了实验结果。所有作者都审阅了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然通讯gydF4y2Ba感谢Jim Clauwaert, Ramana Davuluri和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Lee, D, Yang, J. & Kim, S.使用transformer学习具有大基因组窗口和三维染色质相互作用的组蛋白编码。gydF4y2BaNat CommungydF4y2Ba13gydF4y2Ba, 6678(2022)。https://doi.org/10.1038/s41467-022-34152-5gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467-022-34152-5gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba