摘要gydF4y2Ba
三维基因组结构在基因表达、细胞分化和病理条件中起着关键作用。阐明精细尺度的染色质结构,特别是调控元件之间的相互作用,对于理解基因表达的时空调控至关重要。在本研究中,我们报道了Hi-TrAC作为一种近距离无结扎、可靠且敏感的技术,可在高分辨率上分析调控元件之间的全基因组染色质相互作用。Hi-TrAC以单核小体分辨率检测可达区域之间的染色质环。通过近50万个已识别的环,我们揭示了整个基因组中调控元件的全面相互作用网络。整合转录因子的染色质结合谱后,我们发现内聚蛋白复合物和CTCF负责组织远程染色质环,与结构域的形成有关;而ZNF143和HCFC1参与在调控元件之间构建短程染色质环,直接调控基因表达。因此,我们引入了一种方法来识别一个微妙而全面的顺式调控元件网络,揭示了转录因子在组织染色质环进行基因组组织和基因表达的复杂性和劳动分工。gydF4y2Ba
简介gydF4y2Ba
基因组被组织成高阶染色质结构gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2 gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.每条染色体在细胞核中占据一个独立的区域gydF4y2Ba7gydF4y2Ba.基于活性和非活性相的空间分离,染色质分别被划分为A和B区室gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba.自关联染色质组装成约1mb大小的拓扑关联域(TADs)gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,其中包含大小为几百kb的嵌套子tadsgydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba.染色质结构域由染色质相互作用环组成,这些相互作用环由CTCF和内聚蛋白复合物通过环挤压过程组织gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba.转录调控元件之间的相互作用对于协调基因表达非常重要gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba.了解详细的增强子-启动子相互作用网络对于理解细胞活动的微调是重要的gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba.一种敏感和有效的技术是非常需要阐明全基因组精细结构,特别是增强子-启动子相互作用。人们普遍认为内聚蛋白复合体催化染色质折叠成由CTCF结合锚定的环gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,而其他几个染色质因子,包括ZNF143和YY1,已被证明促进染色质环的形成gydF4y2Ba8gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba.然而,目前尚不清楚这些结构蛋白是如何在基因组组织的不同尺度上编排染色质环的。gydF4y2Ba
在这项研究中,我们开发了一种被称为Hi-TrAC(高灵敏度转座酶介导的染色质分析)的高灵敏度技术,并将其应用于三种不同小鼠和人类细胞类型中Tn5可达染色质区域的染色质环路分析。通过将Hi-TrAC数据中的调控相互作用网络与数百个转录因子ChIP-seq数据整合,我们发现CTCF和内聚蛋白参与了远距离染色质相互作用,而HCFC1和ZNF143参与了短程染色质环的形成。gydF4y2Ba
结果gydF4y2Ba
Hi-TrAC技术改进gydF4y2Ba
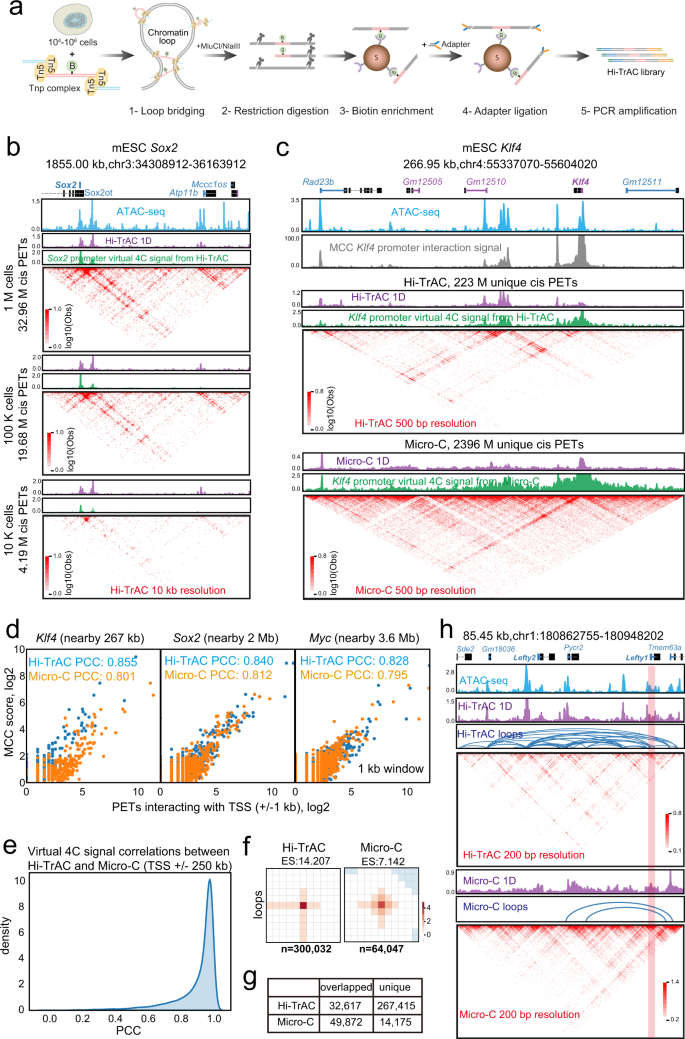
Hi-TrAC起源于Trac-looping(转座酶介导的染色质环分析)方法,并经过了实质性的改进gydF4y2Ba59gydF4y2Ba.Hi-TrAC利用了DNA转座酶Tn5利用特殊设计的二价连接子共价桥接近端开放染色质区域的能力,从而消除了基于3c(染色体构象捕获)技术所需的染色质碎片和近距离连接步骤(图5)。gydF4y2Ba1gydF4y2Ba、方法)。我们还设计了一种策略,消除了trac -loop中大量的滚转周期扩增和稀释结扎,使我们能够将1亿个细胞的起始材料减少到1万个细胞,并将文库构建时间从7天缩短到2天。gydF4y2Ba
一个gydF4y2BaHi-TrAC实验方案。在甲醛固定细胞中使用tnp生物素化的二价ME连接复合物桥接染色质环后,DNA被限制性内切酶MluCI和NlaIII切割。在通用适配器结扎后,使用链霉亲和素珠和pcr扩增来富集桥接的基因组区域。gydF4y2BabgydF4y2BaHi-TrAC可重复检测周围的相互作用gydF4y2BaSox2gydF4y2Ba基因座10gydF4y2Ba6gydF4y2Ba, 10gydF4y2Ba5gydF4y2Ba,和10gydF4y2Ba4gydF4y2BaE14灯头制。ATAC-seq数据来源于GSM1830114gydF4y2Ba114gydF4y2Ba.Hi-TrAC虚拟4c信号仅通过保持pet与的+ /−1 kb TSS区域相互作用产生gydF4y2BaSox2gydF4y2Ba基因,显示为堆积的1D信号。相互作用的宠物被显示为类似4c信号下方的圆点。基因组注释显示在面板的顶部。使用cLoops2绘图模块进行可视化。gydF4y2BacgydF4y2Ba周围相互作用的比较gydF4y2BaKlf4gydF4y2Ba来自汇集MCC的基因位点gydF4y2Ba62gydF4y2Ba、Hi-TrAC和Micro-CgydF4y2Ba60gydF4y2BamESCs中的数据。仅使用Hi-TrAC和Micro-C的染色体内pet进行比较。gydF4y2BadgydF4y2BaMCC探测到的相互作用与周围Hi-TrAC或Micro-C数据虚4c信号的相关性分析gydF4y2BaKlf4gydF4y2Ba,gydF4y2BaSox2gydF4y2Ba,gydF4y2BaMycgydF4y2Ba将视点设置为TSS的+ /−1 kb。源数据作为源数据文件提供。gydF4y2BaegydF4y2BaHi-TrAC和Micro-C虚4c信号在所有蛋白编码基因启动子区域的相关性分布。将启动子定义为TSS上下游+ /−1Kb区域作为视点。只保留任何一端位于启动子区域的pet,将tss上下行250Kb区域作为比较区域。PCC代表Pearson相关系数。gydF4y2BafgydF4y2BaHi-TrAC和Micro-C环的聚合分析。Hi-TrAC循环是由cLoops2 callLoops模块调用的,需要至少20个pet(补充数据gydF4y2Ba2 gydF4y2Ba),而Micro-C循环被HiCCUPS调用。gydF4y2BaggydF4y2BaHi-TrAC和Micro-C循环的重叠。gydF4y2BahgydF4y2Ba基因组浏览器的快照gydF4y2Ba左撇子gydF4y2Ba显示Hi-TrAC和Micro-C检测到的ATAC-seq峰和染色质环的分布以及相互作用矩阵,分辨率为200 bp。gydF4y2Ba
Hi-TrAC在检测染色质环方面优于其他技术gydF4y2Ba
我们首先用最先进的Hi-C变体Micro-C和Micro-Capture-C (MCC)对Hi-TrAC进行了基准测试,它们可以生成高分辨率的染色质结构图gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba.从0.01、0.1和100万个小鼠胚胎干细胞(mESCs)开始(补充数据)gydF4y2Ba1克ydF4y2Ba),以gydF4y2BaSox2gydF4y2BaHi-TrAC可重复检测到相似的染色质相互作用谱(图2)。gydF4y2Ba1 bgydF4y2Ba).为了与MCC数据进行比较,我们提取了所有连接到的启动子的成对端标签(pet)gydF4y2BaKlf4gydF4y2Ba,gydF4y2BaSox2gydF4y2Ba,gydF4y2BaMycgydF4y2Ba从Hi-TrAC和Micro-C数据中提取基因,并将其作为虚拟4c信号显示。Hi-TrAC在这些启动子上检测到的染色质相互作用与MCC和Micro-C检测到的染色质相互作用具有高度可比性,无论是视觉检查还是定量相关分析(图2)。gydF4y2Ba1 c, dgydF4y2Ba).此外,Hi-TrAC和Micro-C数据中的虚拟4c信号在基因组中的所有基因启动子上都高度相关(图2)。gydF4y2Ba1 egydF4y2Ba).Hi-TrAC具有更高的信噪比,与MCC的相关性甚至高于Micro-C,能够检测到更多染色质精细结构的细节(图。gydF4y2Ba1 c, dgydF4y2Ba和补充图。gydF4y2Ba1gydF4y2Ba).这些结果表明Hi-TrAC是一种可靠的检测染色质调控区域之间相互作用的技术。值得注意的是,所有这些染色质环都属于可达染色质区域,这表明染色质可达性是Hi-TrAC检测染色质环所必需的。gydF4y2Ba
Hi-TrAC在检测不同基因组距离的相互作用时比Micro-C表现得更敏感一些(补充图。gydF4y2Ba1 bgydF4y2Ba).在Hi-TrAC中,我们从2.23亿个独特的染色体内pet (cis-PETs)中鉴定出300k个显著染色质环,而在Micro-C中,从2.96亿个独特的cis-PETs中鉴定出64k个染色质环(补充数据)gydF4y2Ba2 gydF4y2Ba).聚合分析显示,Hi-TrAC环路比Micro-C环路表现出更高的富集评分(ES),表明Hi-TrAC数据具有更高的信噪比(图2)。gydF4y2Ba1 fgydF4y2Ba).大约80%的Micro-C环被Hi-TrAC识别,而只有11%的Hi-TrAC环被Micro-C覆盖(图2)。gydF4y2Ba1克gydF4y2Ba和补充图。gydF4y2Ba1 cgydF4y2Ba).举例来说,在功能重要基因的位点,包括gydF4y2BaMycgydF4y2Ba(补充图。gydF4y2Ba1gydF4y2Ba),gydF4y2Ba左撇子gydF4y2Ba(无花果。gydF4y2Ba1 hgydF4y2Ba),gydF4y2BaNanoggydF4y2Ba(gydF4y2Ba补充图。gydF4y2Ba1 dgydF4y2Ba),由于Hi-TrAC具有较高的信噪比,因此可以识别出更显著的环路。尤其是在gydF4y2BaLefty1gydF4y2Ba而且gydF4y2BaNanoggydF4y2BaHi-TrAC只能识别特定的环路,而Micro-C则无法识别(图2)。gydF4y2Ba1 hgydF4y2Ba和补充图。gydF4y2Ba1 dgydF4y2Ba).通常,Micro-C唯一环为远端较弱环(补充图)。gydF4y2Ba1 e, fgydF4y2Ba).大多数microc(76%)和Hi-TrAC(92%)环锚都富集在可接近的染色质区域,以ATAC-seq峰为特征(补充图)。gydF4y2Ba1克gydF4y2Ba)其余未显示抑制性染色质的特征(补充图。gydF4y2Ba1 hgydF4y2Ba),表明这些技术检测到的显著染色质环主要存在于可及调控元件之间。总之,这些结果表明Hi-TrAC是一种检测活性调控区域之间染色质环的敏感方法。gydF4y2Ba
为了进一步评估Hi-TrAC在阐明染色质结构方面的性能,我们将Hi-TrAC应用于GM12878细胞,这是一种人类细胞系,其基因组结构已被各种技术广泛研究(补充数据gydF4y2Ba1克ydF4y2Ba).为了获得一个全面的相互作用图,我们汇集了所有实验重复的数据,得到了8.22亿个原始reads和1.17亿个独特的染色体内pet(补充数据gydF4y2Ba1克ydF4y2Ba).如不同分辨率的二维(2D)热图所示,我们将Hi-TrAC生成的基因组结构图与原位Hi-C构建的可用图进行了比较gydF4y2Ba8gydF4y2Ba, CTCFgydF4y2Ba32gydF4y2Ba、RAD21佳petgydF4y2Ba51gydF4y2Ba,gydF4y2Ba63gydF4y2Ba,捕获Hi-CgydF4y2Ba64gydF4y2Ba, H3K27ac HiChIPgydF4y2Ba65gydF4y2Ba,内聚HiChIPgydF4y2Ba66gydF4y2Ba,以及HiCARgydF4y2Ba67gydF4y2Ba(补充图。gydF4y2Ba2 gydF4y2Ba).在测序深度较低的情况下,Hi-TrAC在不同分辨率下均能清晰检测到染色质畴状结构和环;特别是在识别重要的染色质环时,Hi-TrAC数据比其他方法有更高的信噪比;即使在200 bp分辨率下,也可以观察到超级增强子的精细结构细节,这在其他技术生成的图中是不清楚的(补充图。gydF4y2Ba2 gydF4y2Ba).HiCAR是最近开发的一种近距离连接依赖技术,可通过Tn5检测染色质相互作用。通过比较已发表的HiCAR数据和我们的GM12878细胞Hi-TrAC数据,我们发现虽然这两种方法在检测tads样结构方面表现相似(补充图。gydF4y2Ba2 bgydF4y2Ba), Hi-TrAC在染色质环检测数量(91,042 vs. 48,515)和富集分数显示的信噪比(20.798 vs. 3.402)方面优于HiCAR(补充图。gydF4y2Ba3 hgydF4y2Ba而且gydF4y2Ba5度gydF4y2Ba).gydF4y2Ba
为了系统地比较这些技术可以实现的全基因组最高分辨率,我们计算了不同仓位大小的pet的覆盖率。在超过50%的pet不是单例pet的阈值下,只有H3K27ac HiChIP可以实现与Hi-TrAC相似的分辨率。gydF4y2Ba3.gydF4y2Ba).然后,我们对Hi-TrAC数据进行子采样,以估计达到所需分辨率所需的测序深度。分析表明,6000万个染色体内pet,大约3亿个原始reads,可以实现全基因组1 kb分辨率(补充图)。gydF4y2Ba4gydF4y2Ba),精细尺度的结构也可以在200 bp分辨率的基因组区域子集中识别,包括超级增强子(补充图。gydF4y2Ba4 b, cgydF4y2Ba).gydF4y2Ba
Hi-TrAC检测顺式调控元件的综合相互作用网络gydF4y2Ba
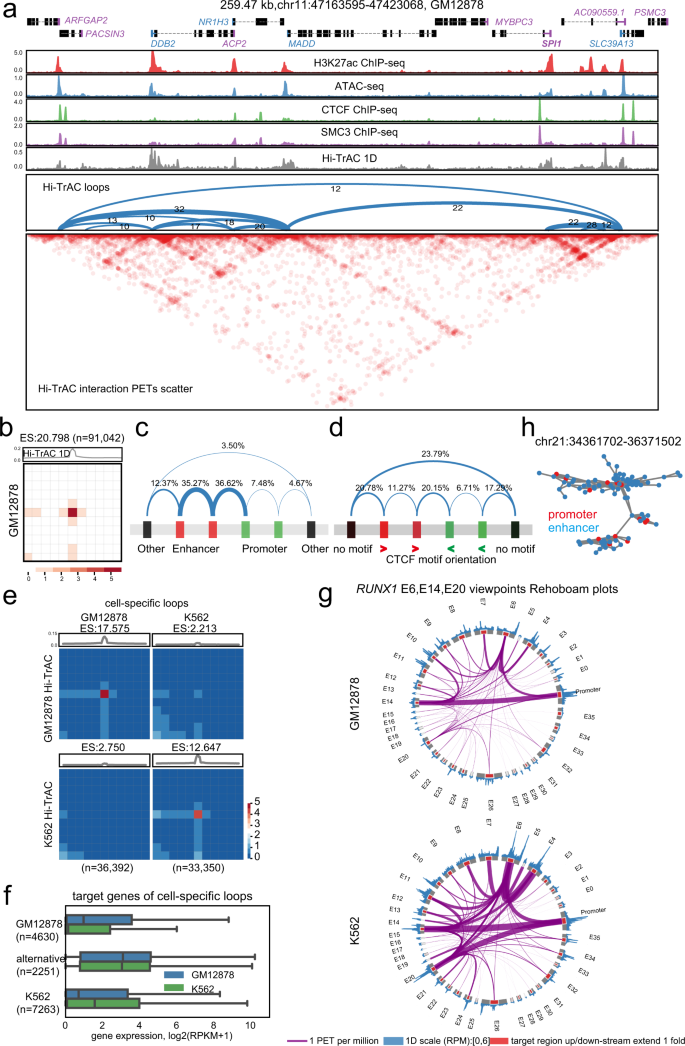
正如预期的那样,Hi-TrAC信号高度集中在可达区域(补充图。gydF4y2Ba5gydF4y2Ba).Hi-TrAC相互作用强度与不同基因组距离内位点的可及性相关性较低(补充图)。gydF4y2Ba5 bgydF4y2Ba),表明Hi-TrAC检测到的相互作用是特定的空间接触。为了探索单个顺式调控元件的细尺度染色质结构是如何组织的,我们进一步分析了Hi-TrAC数据显示的染色质环路。在GM12878单元中,以gydF4y2BaSPIgydF4y2Ba1克ydF4y2Ba该基因编码关键淋巴样细胞发育相关的ETS家族转录因子PU.1,我们观察到在远端和近端调控元件之间形成典型的点对点染色质环模式(图1)。gydF4y2Ba2gydF4y2Ba)gydF4y2Ba.在全球范围内,至少有10个pet支持一个循环,我们在GM12878细胞中称为91,042个高置信循环(图2)。gydF4y2Ba2 bgydF4y2Ba及补充资料gydF4y2Ba3.gydF4y2Ba),远比其他技术(补充图。gydF4y2Ba5度gydF4y2Ba).gydF4y2Ba
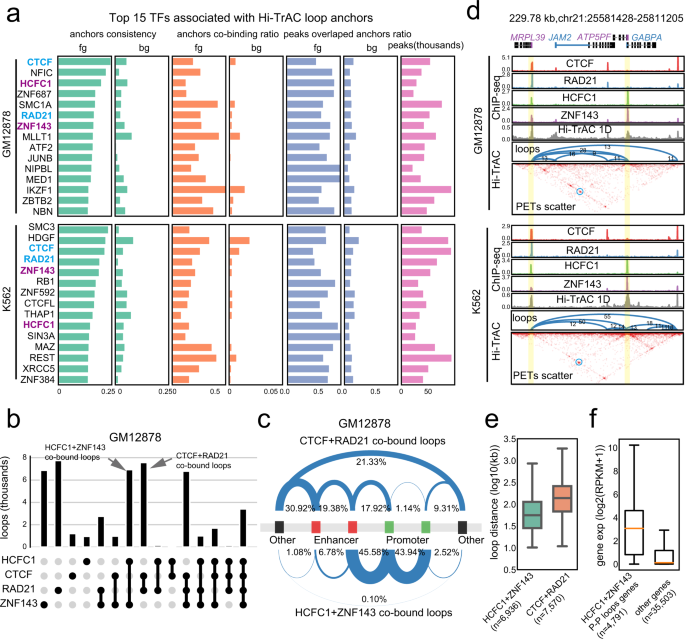
一个gydF4y2Ba基因组浏览器快照显示了Hi-TrAC检测到的染色质环gydF4y2BaSPIgydF4y2Ba1克ydF4y2Ba基因在GM12878细胞中的表达。循环显示为拱形,并且每个循环的pet数量也显示出来。循环由cLoops2 callLoops模块调用,需要至少10个支持的pet。交互矩阵显示在底部。gydF4y2BabgydF4y2BaGM12878中Hi-TrAC数据调用的91,042个环路的聚合分析(补充数据)gydF4y2Ba3.gydF4y2Ba).将循环中相互作用的pet及其附近区域(循环锚点的上游和下游的5倍)作为矩阵平均为聚合热图。ES为富集分数,表示相互作用信号相对于相邻区域的富集程度。使用cLoops2 agg模块进行分析。gydF4y2BacgydF4y2BaGM12878 Hi-TrAC环的类别概述,包括假定的顺式调控元件,包括增强子、启动子和其他。gydF4y2BadgydF4y2BaGM12878 Hi-TrAC环的类别概述,涉及到环的两个锚点的CTCF motif的方向。gydF4y2BaegydF4y2BaGM12878和K562中细胞特异性环的聚合分析(补充数据gydF4y2Ba4gydF4y2Ba).差异丰富的循环被cLoops2 callDiffLoops模块调用。gydF4y2BafgydF4y2Ba在GM12878和K562之间,与细胞特异性环相关的基因的表达水平分布,以及启动子与替代增强子成环的基因的表达水平分布。每个类别的基因数量都被注明。该方框从数据的第一个四分位数延伸到第三个四分位数,在中位数处有一条线。晶须从盒子延伸到四分位范围的1.5倍。胡须末端的飞行点没有显示出来。源数据作为源数据文件提供。gydF4y2BaggydF4y2Ba罗波安图显示在启动子-增强子相互作用的差异gydF4y2BaRUNX1gydF4y2BaGM12878和K562细胞的基因座。从增强子E6, E14和E20的角度展示了相互作用。源数据作为源数据文件提供。gydF4y2BahgydF4y2BaGM12878染色体21上由增强子和启动子组成的最长连接子网络的示例。增强子和启动子形成复杂的连接,形成自然无标度的调控网络。源数据作为源数据文件提供。gydF4y2Ba
我们将Hi-TrAC环的各种特征与其他方法检测到的特征进行了比较。Hi-TrAC环通常距离较短,环锚集中在启动子和增强子上(补充图)。gydF4y2Ba5 dgydF4y2Ba).其中大部分为增强子-启动子,以及增强子-增强子环(图2)。gydF4y2Ba2摄氏度gydF4y2Ba).与其他数据集相比,在Hi-TrAC环锚处的CTCF motif取向更加多样化(补充图)。gydF4y2Ba5 hgydF4y2Ba和无花果。gydF4y2Ba二维gydF4y2Ba),其中一小部分在收敛方向,这些环似乎更遥远(补充图。gydF4y2Ba5我gydF4y2Ba),表明这些环可能与畴的形成有关。许多Hi-TrAC环锚没有CTCF基序(图2)。gydF4y2Ba二维gydF4y2Ba和补充图。gydF4y2Ba5 hgydF4y2Ba),提出了这些循环的不同功能和组织机制,Hi-TrAC捕获了更多功能的循环。gydF4y2Ba
为了研究染色质环与细胞身份和活性之间的关系,我们进一步在另一种人类细胞系K562细胞中进行Hi-TrAC(补充数据)gydF4y2Ba1克ydF4y2Ba).在K562细胞中共鉴定出98,850个染色质环(补充数据gydF4y2Ba3.gydF4y2Ba).我们假设细胞特异性染色质环可能控制细胞特异性基因表达。为了验证这一点,我们比较了GM12878和K562细胞中的环路特征,并分别鉴定出36,392和33,350个细胞类型特异性环路(补充数据gydF4y2Ba4gydF4y2Ba).细胞特异性环路在相应细胞中显示了显著的相互作用信号富集(图2)。gydF4y2Ba2 egydF4y2Ba).我们鉴定了4630个与gm12878特异性启动子环相关的基因和7263个与k562特异性启动子环相关的基因。同时,我们还发现了2251个基因在GM12878和K562细胞之间显示了可选的启动子-增强子循环。GM12878特异性环相关基因在GM12878细胞中表达量高于K562细胞;反之亦然(图;gydF4y2Ba2 fgydF4y2Ba).然而,GM12878和K562细胞之间显示选择性环的基因表达无显著差异(图2)。gydF4y2Ba2 fgydF4y2Ba).通过聚合分析,我们使用其他方法的数据集验证了Hi-TrAC识别的细胞特异性环路,包括原位Hi-C, H3K27ac HiChIP和RAD21 ChIA-PET(补充图)。gydF4y2Ba6gydF4y2Ba),证明Hi-TrAC成功检测了与细胞活性相关的差异染色质环。gydF4y2Ba
综合调控相互作用网络以几个代表性基因位点为例。EBF1是B淋巴生成的关键转录因子,在GM12878细胞中特异性表达,而GATA1是红细胞生成的主转录因子,仅在K562细胞中表达。gydF4y2BaEBF1gydF4y2Ba而且gydF4y2BaGATA1gydF4y2Ba基因位点在相应细胞中表现出独特的相互作用模式,与它们的表达谱一致(补充图)。gydF4y2Ba7 a、bgydF4y2Ba).之前的一项优雅的CRISPRi筛选研究确定了多种调控元件gydF4y2BaGATA1gydF4y2Ba表达式gydF4y2Ba68gydF4y2Ba.有趣的是,拿gydF4y2BaGATA1gydF4y2Ba以启动子为观点,Hi-TrAC虚拟4c信号与CRISPRi评分相关性良好(补充图;gydF4y2Ba7 c, dgydF4y2Ba),表明Hi-TrAC检测功能相关调控相互作用的鲁棒性。gydF4y2Ba
以另一个k562特异性关键转录因子基因为例gydF4y2BaRUNX1gydF4y2Ba作为一个例子(补充图。gydF4y2Ba7 egydF4y2Ba).虽然综合调控网络可以用相互作用接触矩阵热图和环路圆弧图来表示,但它太复杂了,无法对每个个体进行直观检查gydF4y2Ba独联体gydF4y2Ba监管元素。因此,我们设计了一个罗波安图来可视化特定基因组区域的染色质环路,它清楚地揭示了连接不同顺式调控元件的单个环路:K562细胞中的启动子和增强子E6和E20之间的相互作用要比GM12878细胞中的强得多,因此这可能是其在K562细胞中的表达(图128)。gydF4y2Ba2 ggydF4y2Ba).gydF4y2Ba
我们整合了所有的增强子和启动子循环,并生成了一个全面的调控相互作用网络(图2)。gydF4y2Ba2 hgydF4y2Ba).网络中增强子和启动子的连接程度符合无标度网络幂律,揭示了调控网络的复杂性(图2)。gydF4y2Ba2 hgydF4y2Ba和补充图。gydF4y2Ba8gydF4y2Ba).增强子的连接程度高于启动子的连接程度(补充图。gydF4y2Ba8gydF4y2Ba),与增强子-增强子循环的高比例相关(图。gydF4y2Ba2摄氏度gydF4y2Ba).平均而言,一个增强子与一个启动子直接相互作用,一个启动子与几乎三个增强子直接接触,这表明顺式调控网络的鲁棒性设计是冗余的(补充图。gydF4y2Ba8 bgydF4y2Ba).gydF4y2Ba
为了测试直接和间接增强子环在相互作用网络中对目标启动子的功能贡献,我们选择了gydF4y2BaCEMIP2gydF4y2Ba基因位点作为模型,在K562细胞中具有多个潜在的增强子(注释为E1-E8)(补充图。gydF4y2Ba8 cgydF4y2Ba).E2直接与启动子相互作用,E3和E5都与E2相互作用强烈,但与启动子不相互作用。有趣的是,通过CRISPR/Case9删除这三种潜在增强子中的任何一种都会导致蛋白表达降低gydF4y2BaCEMIP2gydF4y2Ba(补充图。gydF4y2Ba8 dgydF4y2Ba).与此同时,gydF4y2BaCEMIP2gydF4y2Ba删除这些元素可以减少启动子的相互作用(补充图。gydF4y2Ba8 egydF4y2Ba).这些结果表明,删除相互作用网络中的一个节点可能会影响整个调控相互作用网络的稳定性,导致基因表达失调。gydF4y2Ba
HCFC1和ZNF143与启动子中心染色质环相关gydF4y2Ba
我们注意到很多环锚没有被CTCF或SMC3占据。gydF4y2Ba2gydF4y2Ba),特别是对于短距离的环,这表明其他因素可能在这些位点组织染色质环。为了识别这些潜在的因素,我们分析了GM12878中Hi-TrAC环锚点上162个转录因子(TFs)和K562细胞中360个转录因子(TFs)的富集gydF4y2Ba5gydF4y2Ba).两种细胞中富集最多的15种tf包括CTCF和RAD21(图2)。gydF4y2Ba3gydF4y2Ba).有趣的是,分析还显示,两个细胞都有两个高度富集的tf, HCFC1和ZNF143,这表明它们也可能广泛参与编排染色质环。HCF1和ZNF143是普遍表达的tf,在靶基因启动子中起作用,调节细胞代谢、增殖和分化gydF4y2Ba69gydF4y2Ba,gydF4y2Ba70gydF4y2Ba,gydF4y2Ba71gydF4y2Ba,gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba,gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba.HCFC1和ZNF143的调控异常与疾病(如癌症)的发病有关gydF4y2Ba77gydF4y2Ba,gydF4y2Ba78gydF4y2Ba,gydF4y2Ba79gydF4y2Ba,gydF4y2Ba80gydF4y2Ba.越来越多的证据表明HCFC1和ZNF143可能参与了染色质结构的组织gydF4y2Ba8gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba81gydF4y2Ba,gydF4y2Ba82gydF4y2Ba,gydF4y2Ba83gydF4y2Ba,gydF4y2Ba84gydF4y2Ba,gydF4y2Ba85gydF4y2Ba.在GM12878中,与HCFC1/ZNF143而非CTCF或RAD21共结合的环数与CTCF/RAD21而非HCFC1或ZNF143共结合的环数相似(图1)。gydF4y2Ba3 bgydF4y2Ba).此外,我们的数据表明,只有一小部分CTCF/RAD21共结合环与启动子相关,而惊人的是,近90%的启动子环由HCFC1/ZNF143结合锚定。gydF4y2Ba3 c, dgydF4y2Ba).HCFC1/ZNF143共结合环的基因组距离通常比CTCF/RAD21环短(图2)。gydF4y2Ba3 egydF4y2Ba).具有HCFC1/ZNF143启动子-启动子环的基因比其他基因表达水平更高(图2)。gydF4y2Ba3 fgydF4y2Ba).这些结果提示了不同结构蛋白的染色质环的“分工”模型。gydF4y2Ba
一个gydF4y2Ba在GM12878和K562细胞中,与Hi-TrAC环相关的前15个转录因子。162和360个转录因子的结合位点从ReMap 2020中编译gydF4y2Ba105gydF4y2BaGM12878和K562。与循环锚点最显著相关的前15个因素,按一致性排序,如面板左侧所示(补充数据gydF4y2Ba5gydF4y2Ba)gydF4y2Ba.GM12878和K562之间重叠的顶部因子用紫色和蓝色突出显示。Fg代表前台数据,即实际循环。Bg代表后台数据,这意味着实际循环锚点附近的区域,并用作控件。源数据作为源数据文件提供。gydF4y2BabgydF4y2Ba在GM12878的两个锚点上,HCFC1、CTCF、RAD21和ZNF143单独或组合结合的环的分布(上面板)。源数据作为源数据文件提供。gydF4y2BacgydF4y2Ba关于GM12878中HCFC1 + ZNF143共结合的环和CTCF + RAD21共结合的环的假定顺式调控元件的环类别的总结。gydF4y2BadgydF4y2Ba一个由HCFC1 + ZNF143而不是CTCF + RAD21共结合的启动子-启动子环的例子。gydF4y2BaegydF4y2BaGM12878中HCFC1 + ZNF143共结合环和CTCF + RAD21共结合环的锚点距离分布。该方框从数据的第一个四分位数延伸到第三个四分位数,在中位数处有一条线。晶须从盒子延伸到四分位范围的1.5倍。胡须末端的飞行点没有显示出来。N =循环数。源数据作为源数据文件提供。gydF4y2BafgydF4y2Ba在GM12878中HCFC1 + ZNF143等基因共结合的启动子-启动子环基因的表达水平。该方框从数据的第一个四分位数延伸到第三个四分位数,在中位数处有一条线。晶须从盒子延伸到四分位范围的1.5倍。胡须末端的飞行点没有显示出来。gydF4y2BangydF4y2Ba=基因的数量。源数据作为源数据文件提供。gydF4y2Ba
干扰CTCF、RAD21、ZNF143和HCFC1会导致环的明显扰动gydF4y2Ba
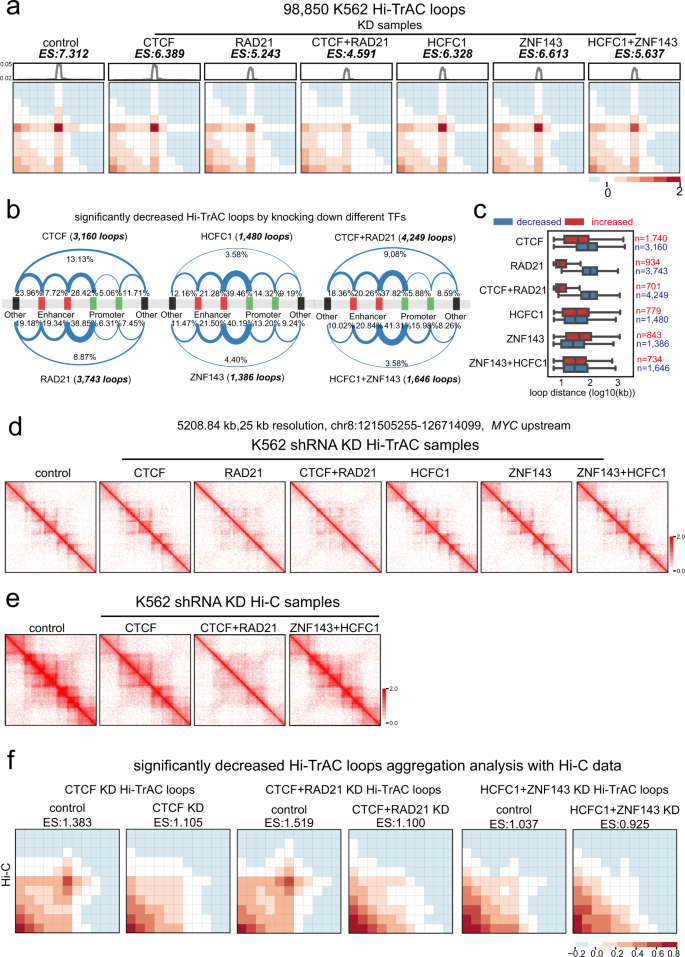
为了测试CTCF、RAD21、HCFC1和ZNF143在维持染色质循环中的作用,我们在K562细胞中单独或联合敲除了这些不同的因子(KD)。gydF4y2Ba9 a、bgydF4y2Ba).然后用Hi-TrAC(补充数据)分析得到的细胞gydF4y2Ba1克ydF4y2Ba).将所有环内的pet富集值与邻近区域进行比较,以比较敲除对染色质环的影响。同时敲除CTCF和RAD21或同时敲除HCFC1和ZNF143会导致染色质环更严重的下降(补充数据)gydF4y2Ba6gydF4y2Ba,无花果。gydF4y2Ba4gydF4y2Ba),这表明这些tf在一般情况下促进了循环,并且它们可能合作地调节循环。与这一观点一致,每次敲除分别显著减少3160 (CTCF KD)、3743 (RAD21 KD)、1480 (HCFC1 KD)和1386 (ZNF143 KD)环,而增强较小数量的环:1740 (CTCF KD)、934 (RAD21 KD)、779 (HCFC1 KD)和843 (ZNF143 KD)(补充图)。gydF4y2Ba9 cgydF4y2Ba).CTCF和RAD21同时敲除减少4249个回路,增强701个回路;HCFC1和ZNF143同时敲除减少1646个回路,增强734个回路。gydF4y2Ba9 cgydF4y2Ba).虽然在一小部分可达区域检测到可达性降低和相互作用减少,但染色质环强度的变化与全球水平的可达性变化没有很强的相关性(补充数据)gydF4y2Ba7gydF4y2Ba,补充图。gydF4y2Ba9 dgydF4y2Ba).以两个随机选取的区域为例(补充图。gydF4y2Ba9 e, fgydF4y2Ba),在敲除CTCF减少的3160个循环中,412个循环中锚点的可及性没有降低,这表明染色质相互作用和可及性都可能对Hi-TrAC信号起作用。我们发现,敲除CTCF和RAD21后降低的环锚分别有48.8%和35.5%为非增强子和启动子,而敲除HCFC1和ZNF143后降低的环锚较低部分(分别为24.93%和25.11%)属于这类可达区域(图2)。gydF4y2Ba4 bgydF4y2Ba).同样,同时敲除CTCF和RAD21降低环的锚点,非启动子和非增强子区域的比例(36.03%)也高于同时敲除HCFC1和ZNF143降低的比例(21.86%)。gydF4y2Ba4 bgydF4y2Ba).相比之下,无论是单独敲除还是同时敲除HCFC1和ZNF143,都会导致更高比例的增强子和启动子相关环路中断(图2)。gydF4y2Ba4 bgydF4y2Ba).敲除CTCF和/或RAD21后,环的中位尺寸减小了~ 100 kb,敲除HCFC1和/或ZNF143后,环的中位尺寸减小了~ 20-30 kb(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).这些结果表明,与CTCF和RAD21相比,HCFC1和ZNF143参与了不同组染色质环的组织。gydF4y2Ba
一个gydF4y2Ba聚合分析显示,敲除CTCF、RAD21、HCFC1和ZNF143后,K562细胞染色质环强度下降(补充数据)gydF4y2Ba6gydF4y2Ba).浓缩分数(ES)是每个循环中所有浓缩分数的平均值。KD,击倒。gydF4y2BabgydF4y2Ba关于TF(转录因子)敲除细胞中减少循环的假定顺式调节元件的循环类别的总结。gydF4y2BacgydF4y2BaTF敲除后改变环的基因组距离分布。该方框从数据的第一个四分位数延伸到第三个四分位数,在中位数处有一条线。晶须从盒子延伸到四分位范围的1.5倍。胡须末端的飞行点没有显示出来。N =循环数。源数据作为源数据文件提供。gydF4y2BadgydF4y2Ba的上游基因组区域结构域中断的一个例子gydF4y2BaMYCgydF4y2Ba用RAD21敲除RAD21或CTCF后Hi-TrAC检测基因。gydF4y2BaegydF4y2Ba原位Hi-C数据显示与面板相同区域的染色质结构域被破坏gydF4y2BadgydF4y2Ba拆除CTCF和RAD21(补充数据gydF4y2Ba8gydF4y2Ba).gydF4y2BafgydF4y2Ba利用与面板相同的Hi-C原位数据,对Hi-TrAC检测到的显著减少的环进行环聚集分析gydF4y2BaegydF4y2Ba.gydF4y2Ba
为了进一步验证Hi-TrAC检测到的染色质结构变化,我们对对照细胞和敲除细胞进行了原位Hi-C(补充数据)gydF4y2Ba8gydF4y2Ba).与我们在Hi-TrAC数据中观察到的结果一致(图。gydF4y2Ba4 dgydF4y2Ba),原位Hi-C数据也显示,通过敲除CTCF和RAD21,而不是单独敲除CTCF或同时敲除HCFC1和ZNF143,结构域结构也受到严重破坏(图2)。gydF4y2Ba4 egydF4y2Ba).Hi-TrAC鉴定出明显减少的环路也显示相应细胞的原位Hi-C数据减少(图2)。gydF4y2Ba4 fgydF4y2Ba).这些结果表明Hi-TrAC可以准确地检测染色质结构的变化。gydF4y2Ba
为了研究染色质环与基因表达之间的相关性,我们分析了对照细胞和敲除细胞中的基因表达谱(补充数据)gydF4y2Ba9gydF4y2Ba).敲除这些因子会导致上调和下调基因:CTCF为371和239,RAD21为926和925,HCFC1为379和237,ZNF143为255和466(补充图)。gydF4y2Ba10gydF4y2Ba).值得注意的是,HCFC1和ZNF143的同时敲低比单独敲低下调了更多的基因(646个)。虽然基因表达变化与染色质环之间一般没有显著相关性,但同时敲除HCFC1和ZNF143下调基因的Pearson相关系数(PCC)为0.219。gydF4y2Ba10 bgydF4y2Ba),这表明需要这两种因素的循环可能有助于这组基因的表达。然而,这些数据也表明染色质环的变化与基因表达之间存在复杂的关系,这表明HCFC1和znf143依赖的环可以介导基因激活或抑制。gydF4y2Ba
HCFC1和ZNF143分别与CTCF和RAD21协同工作gydF4y2Ba
为了确定染色质环的改变是否是耗尽这些tf的直接后果,我们在敲除细胞中用ChIP-seq检查了它们的染色质结合谱(补充数据)gydF4y2Ba10gydF4y2Ba).同时敲除CTCF和RAD21显著降低了RAD21的结合,而仅轻微降低CTCF的结合(补充图)。gydF4y2Ba11个gydF4y2Ba).有趣的是,HCFC1的结合也显著受损。双敲除HCFC1和ZNF143明显削弱了它们的结合,对CTCF和RAD21的结合也有轻微影响(补充图)。gydF4y2Ba11个gydF4y2Ba).HCFC1和ZNF143结合位点通过“ACTACANNTCCCA”ZNF143相关基序富集(补充图)。gydF4y2Ba11 bgydF4y2Ba).超过70%的HCFC1和ZNF143共结合位点位于启动子(补充图。gydF4y2Ba11 cgydF4y2Ba).在含有RAD21的CTCF和含有ZNF143双敲除细胞的HCFC1中,减少的环锚中最富集的基序包括“CTCF”和“GATA”基序(补充图)。gydF4y2Ba11 dgydF4y2Ba).尽管在HCFC1和ZNF143双敲低细胞中,只有8%的环锚点是HCFC1和ZNF143共结合峰(补充图。gydF4y2Ba11 egydF4y2Ba), HCFC1/ZNF143 motif仍然是富集最多的motif之一(补充图。gydF4y2Ba11 dgydF4y2Ba).此外,在HCFC1和ZNF143双敲除细胞中,那些不被HCFC1或ZNF143结合的减少的环锚被“CTCF”和“GATA”基序富集(补充图)。gydF4y2Ba11 fgydF4y2Ba).这些结合基序分析表明,HCFC1和ZNF143可能单独或与CTCF和RAD21一起起作用。gydF4y2Ba
HCFC1和ZNF143通过组织启动子环来协调基因表达gydF4y2Ba
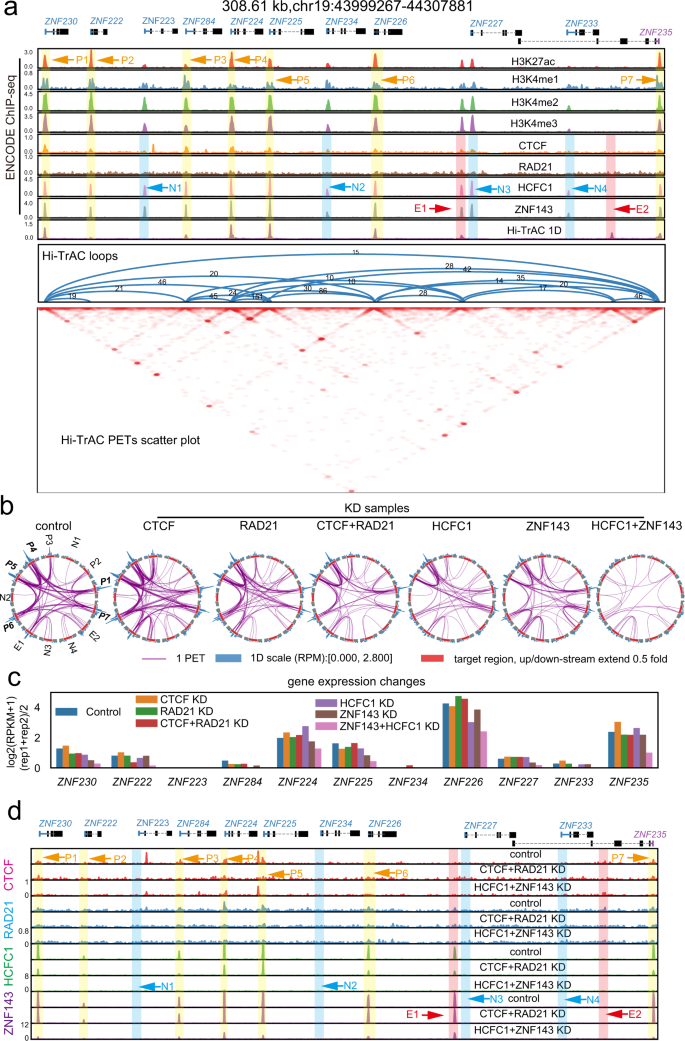
为了进一步研究HCFC1和ZNF143在组织染色质结构中的功能,我们检测了一个含有多个锌指(ZNF)基因的灵长类动物特异性基因组区域。该区域表现出强烈的启动子-启动子相互作用,在K562和GM12878细胞之间保守。在该位点的环锚处检测到多个HCFC1和ZNF143结合峰,而没有检测到强烈的CTCF和RAD21结合(图2)。gydF4y2Ba5gydF4y2Ba).同时敲除HCFC1和ZNF143严重破坏了该区域内的启动子-启动子环,如Rehoboam图所示。gydF4y2Ba5 bgydF4y2Ba),同时伴有靶基因表达减少(图。gydF4y2Ba5度gydF4y2Ba),表明HCFC1和ZNF143在调控环和这些基因表达方面起着关键作用。ChIP-seq证实,启动子-启动子环的减少与HCFC1和ZNF143的绑定受损相关(图。gydF4y2Ba5 dgydF4y2Ba).我们进一步验证了Hi-TrAC在HCFC1和ZNF143敲除细胞中检测到的启动子-启动子环的破坏,并使用3C-qPCR检测。结果表明,两者的gydF4y2BaZNF224gydF4y2Ba-gydF4y2BaZNF284gydF4y2Ba而且gydF4y2BaZNF225gydF4y2Ba-gydF4y2BaZNF235gydF4y2Ba敲除HCFC1和ZNF143后环明显减少(补充图。gydF4y2Ba12个一个gydF4y2Ba).另外两个在ZNF基因簇区域外随机选择的启动子-启动子环,gydF4y2BaMRPL24gydF4y2Ba-gydF4y2BaPRCCgydF4y2Ba而且gydF4y2BaNDC1gydF4y2Ba-gydF4y2BaTCEANC2gydF4y2Ba,敲除HCFC1和ZNF143也显著受损(补充图。gydF4y2Ba12 a, bgydF4y2Ba).gydF4y2Ba
一个gydF4y2BaHi-TrAC检测到K562细胞19号染色体上ZNF基因簇的染色质环。还显示了活跃组蛋白修饰的ENCODE ChIP-seq信号和4个tf (H3K4me1、2,3和H3K27ac、CTCF、RAD21、HCFC1和ZNF143)gydF4y2Ba115gydF4y2Ba).与HCFC1和ZNF143共结合且有环的启动子被标注为P1 ~ P7,与HCFC1和ZNF143共结合但未检测到环的启动子被标注为N1 ~ N4。与HCFC1和ZNF143共结合且有环的非启动子区域标注为E1,与其他区域有环但未与HCFC1和ZNF143结合的区域标注为E2。gydF4y2BabgydF4y2Ba对照细胞和敲除细胞ZNF簇区域染色质环的罗波安图如图所示gydF4y2Ba一个gydF4y2Ba.源数据作为源数据文件提供。KD,击倒。gydF4y2BacgydF4y2Ba通过RNA-seq检测敲除CTCF、RAD21、HCFC1和ZNF143在K562细胞中ZNF基因的表达变化。gydF4y2BadgydF4y2Ba用ChIP-seq检测对照、CTCF + RAD21双敲除或HCFC1 + ZNF143双敲除K562细胞中CTCF、RAD21、HCFC1和ZNF143在ZNF基因簇区域的结合谱。gydF4y2Ba
为了进一步测试HCFC1和znf143依赖的染色质环是否对这些基因的表达有积极或消极的贡献,我们使用CRISPR删除ZNF225或ZNF234启动子环锚定在ZNF基因簇区域:表达gydF4y2BaZNF225gydF4y2Ba启动子被HCFC1和ZNF143结合,并与该区域的其他启动子相互作用,而非表达gydF4y2BaZNF234gydF4y2Ba启动子与HCFC1和ZNF143的结合非常弱,不与其他启动子相互作用,因此作为阴性对照(图。gydF4y2Ba5 a - cgydF4y2Ba).删除gydF4y2BaZNF225gydF4y2Ba启动子环锚定导致在gydF4y2BaZNF225gydF4y2Ba启动子和减少子gydF4y2BaZNF225gydF4y2Ba基因表达符合预期(补充图。gydF4y2Ba12 c, dgydF4y2Ba).令人惊讶的是,删除gydF4y2BaZNF225gydF4y2Ba启动子锚定导致该区域其他启动子环的形成增加,也增加了这些基因的表达(补充图。gydF4y2Ba12 c, dgydF4y2Ba).这些结果表明,HCFC1和ZNF143结合锚有助于启动子环的形成,从而对附近基因的表达产生负面影响,可能是通过直接竞争有限的转录调控因子。gydF4y2Ba
讨论gydF4y2Ba
我们在这里提出Hi-TrAC作为一种敏感的技术,用于在可访问的基因组区域的转录和染色质调节元件之间映射染色质环路。该技术与track -loop的基本概念相同,后者利用Tn5的能力将DNA桥整合到可接近的染色质区域,以共价连接物理接触的染色质位点,从而避免了3c衍生技术所需的近距离连接gydF4y2Ba59gydF4y2Ba.track -loop需要5000 - 1亿个细胞和5-7天的台架工作。Hi-TrAC更加通用和高效,只需0.01万个细胞,不到两天的工作量就可以完成。此外,它仅通过3亿个测序读取,就能捕捉到详细的相互作用信息,分辨率高达200 bp,具有高灵敏度检测调控元件之间的相互作用。Hi-TrAC检测到的染色质相互作用与Micro-C、MCC甚至CRISPRi检测到的染色质相互作用高度相关,表明Hi-TrAC可以可靠地捕获调控元件的空间接触。此外,与Micro-C相比,Hi-TrAC检测到的染色质环多了4倍(300k vs 64k),而pet仅为10% (223 M vs 2396 M);Hi-TrAC检测到了MCC检测到的几乎所有潜在增强子与其目标启动子之间的相互作用。与其他基于3c的技术相比,Hi-TrAC在低测序量的高分辨率下检测更多染色质环方面也具有优势。因此,Hi-TrAC可以作为一种廉价、高灵敏度和鲁棒的替代方法,用于分析染色质和转录调控元件之间的相互作用。gydF4y2Ba
Hi-TrAC中使用的桥接连接器就像绘制基因组图谱的尺子。要由连接器桥接,两个相互作用的位点之间的空间距离应小于连接器的长度。我们测试了不同长度的连接子,发现在侧翼Tn5结合位点之间间隔30 bp的连接子表现最好。较长的连接蛋白捕获了太多的染色质间相互作用,而较短的连接蛋白失去了许多远端相互作用(补充数据)gydF4y2Ba11gydF4y2Ba).所选择的桥接连接体估计约为20纳米长,这表明Hi-TrAC捕获相互作用区域之间的空间物理距离在这一范围内。gydF4y2Ba
增强子-启动子相互作用环在控制基因时空表达中起着关键作用gydF4y2Ba31gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba43gydF4y2Ba.以β-珠蛋白基因座为例,胎儿和成人血红蛋白的表达转换是由基因座控制区(LCR)和相应基因的启动子在转录因子的作用下相互作用调控的gydF4y2Ba86gydF4y2Ba,gydF4y2Ba87gydF4y2Ba.为了在全基因组范围内获得基因调控机制的全面信息,识别增强子和启动子的调控相互作用网络是非常重要的。然而,在之前的研究中,即使是最全面的3D数据也只能提供有限的全基因组增强子-启动子环信息gydF4y2Ba8gydF4y2Ba,gydF4y2Ba61gydF4y2Ba.据我们所知,我们现在提供了GM12878、K562细胞和mESCs中可达染色质区域之间最全面的相互作用数据,共报告了约50万个染色质环。此外,我们的数据还揭示了共享的染色质环和细胞特异性环,有助于负责分化和细胞功能的基因的细胞特异性表达。即使是在两种细胞类型中表达水平相似的基因,调控元件的相互作用,特别是增强子的相互作用,也表现出不同的模式,这表明基因表达在不同的细胞中可能受到不同的调控。gydF4y2Ba
根据环锚点相对于染色质域组织的位置,染色质环可以大致分为三类:(1)在TAD边界;(2)亚tad边界;(3)在TADs和子TADs内。它们也可以根据循环锚的功能注释进行分类:(1)启动子;(2)增强剂;(3)其他。虽然连接增强子和启动子的环通常在TADs或子TADs中发现,但其他环被映射到这些染色质域的边界上。通常,从域边界开始的循环要比链接增强子和启动子(<100 kb)长得多(>100 kb)。已有数据证实CTCF和内聚蛋白复合物在维持TADs的染色质相互作用中起关键作用gydF4y2Ba48gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba88gydF4y2Ba,gydF4y2Ba89gydF4y2Ba,gydF4y2Ba90gydF4y2Ba通过挤压模型gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba.几种特定的转录因子已被发现有助于增强子和启动子之间的染色质循环gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba.YY1促进增强子和启动子之间的环路形成并调控基因表达,ZNF143被报道与CTCF共同作用促进染色质环路。然而,总的来说,调控增强子-启动子循环的机制需要进一步阐明。在本研究中,我们发现除了CTCF和RAD21外,ZNF143和HCFC1也是最富集的因子,并在GM12878和K562细胞中共享,表明这两个因子可能在染色质成环中发挥普遍作用。有趣的是,在GM12878细胞中,与ZNF143和HCFC1共结合的环锚中96%以上涉及增强子和启动子,而与CTCF和RAD21共结合的环锚中只有38%涉及增强子和启动子,这表明ZNF143和HCFC1在促进增强子-启动子环中发挥了普遍作用。敲除HCFC1和/或ZNF143中断的环的中位数大小为20 kb,与增强子和启动子相互作用的大小一致;敲除CTCF和/或RAD21中断的环的中位数大小约为100 kb,与结构域边界之间的距离一致。gydF4y2Ba
尽管之前有人认为启动子-启动子相互作用可以通过使目标启动子靠近来促进基因表达gydF4y2Ba29gydF4y2Ba,gydF4y2Ba91gydF4y2Ba,gydF4y2Ba92gydF4y2Ba,gydF4y2Ba93gydF4y2Ba,需要直接的数据来支持这一假设。在这里,我们发现HCFC1和ZNF143结合在ZNF基因簇位点启动子环的锚上;同时敲除HCFC1和ZNF143破坏了启动子-启动子环,降低了这些目标启动子的表达,从而为启动子-启动子环有助于基因表达提供了强有力的证据。然而,由于染色质可达性是Hi-TrAC检测染色质相互作用的必要条件,Hi-TrAC检测到的相互作用降低可能来自单独的相互作用降低,也可能来自可达性和相互作用的降低,这两者都可以由HCFC1和ZNF143调节。基于这些结果,我们提出不同的结构蛋白在组织染色质环方面存在分工:CTCF和RAD21负责构建染色质结构域的外部框架,而HCFC1和ZNF143通过组织调控元件之间的环来装饰内部结构。随着高分辨率基因组结构的开发和完成,以及更多的转录因子染色质结合数据集,将会发现更多的染色质环调控因子,包括通用调控因子和细胞类型特异性调控因子。我们关于可达染色质区域相互作用网络的全面数据为进一步探索基因组组织的复杂功能和机制提供了丰富的资源。gydF4y2Ba
方法gydF4y2Ba
细胞系gydF4y2Ba
GM12878细胞购自科瑞尔研究所;K562 (CCL-243)、293 T (CRL-3216)、mES-E14TG2a (CRL-1821)和mES-R1 (SCRC-1011)细胞购自ATCC。gydF4y2Ba
Hi-TrAC实验程序gydF4y2Ba
细胞用1%甲醛在室温培养基中固定10分钟。用1毫升冰冷的PBS清洗细胞两次,然后将细胞放在冰上。Tnp配合物由2 μL short adapter (50 μM)、2 μL bridging linker (25 μL)混合而成gydF4y2Ba11gydF4y2Ba)、2 μL甘油和4 μL Tn5 (100 μM),室温孵育20 min。用100 μL反应缓冲液(50 mM tri -acetate, pH 7.5, 150 mM acetate钾,10 mM acetate镁,4 mM亚精胺,0.5% NP-40)重悬细胞,室温孵育10 min。向渗透细胞中加入10 μL Tnp复合物,用移液法轻轻混合,在37℃恒温器上孵育4小时,间歇混合。加入EDTA(终浓度25 mM)和SDS(终浓度0.3%)停止反应。在反应混合物中加入2 μL蛋白酶K (20 mg/mL), 65℃孵育过夜反交联。用苯酚-氯仿萃取纯化DNA。在含有dNTPs的反应混合物中,用T4 DNA聚合酶在室温下修复DNA间隙30分钟。通过将大片段(>100 bp)选择性地结合到AMPure XP珠上,去除游离桥接子(68 bp)。用80 μL洗脱缓冲液从AMPure XP珠中洗脱DNA,用限制性内切酶2 μL MluCI (NEB, R0538L)和2 μL NlaIII (NEB, R0125L)在100 μL反应液中37℃消化30 min。加入100 μL 2x B&W缓冲液(10 mM Tris-HCl, pH 7.5, 1 mM EDTA, 2 M NaCl, 0.1% Tween-20)调整反应混合物为1倍B&W缓冲液,然后加入5 μL Streptavidin C1珠(Invitrogen, 65001)旋转混合30 min。 The beads were washed 5 times with 1 mL 1x B&W buffer. Biotin-labeled DNA fragments captured on beads were ligated to multiplexing adapters by adding 5 μL each adapter (50 μM) and 1 μL T7 DNA ligase (NEB, M0318L) in 100 μL ligation mixture and incubating at room temperature for 1 h with rotation. Before PCR amplification, the beads were washed 5 times with 1x B&W buffer. The Hi-TrAC libraries were then amplified with multiplexing indexed primers in the following reaction mixture: 20 μL Phusion HF PCR Master Mix (NEB, M0531S), 1 μL Illumina Multiplexing PCR primer 1.0 (10 μM), 1 μL Illumina Multiplexing PCR index primer (10 μM) and 18 μL H2 gydF4y2BaO为12个周期。将300 bp - 700 bp之间的DNA片段在Illumina平台上进行配对测序。gydF4y2Ba
击倒和western blottinggydF4y2Ba
通过shRNA慢病毒转导,在K562细胞中敲除CTCF、RAD21、HCFC1和ZNF143。shRNA模板克隆到pGreenPuro慢载体(System Biosciences, SI505A-1)。shRNA目标是:Control-shRNA-1: GCGCGATAGCGCTAATAATTT, Control-shRNA-2: CAACAAGATGAAGAGCACCAA;CTCF-shRNA-1: GGAGAAACGAAGAAGAGTA, CTCF-shRNA-2: GTAGAAGTCAGCAAATTAA;RAD21-shRNA-1: AGAGTTGGATAGCAAGACA, RAD21-shRNA-2: GGAAGCTAATTGTTGACAGTGTCAA;HCFC1-shRNA-1: GCAACCACCATCGGAAATAAA, HCFC1-shRNA-2: AGAACAACATTCCAAGGTACCTGAA;ZNF143-shRNA-1: GCTACAAGAGTAACTGCTAAA, ZNF143-shRNA-2: ggacgacgtttttctacacaagta。9 μg psPAX2 (Addgene, #12260)和3 μg pMD2包质粒共转染12 μg慢载体。G (Addgene, #12259)转染到293个T细胞,在100mm培养皿中培养。转染后12 h用12 mL新鲜培养基改变,转染后72 h收集含有病毒的培养基上清。将培养基加入到300万个K562细胞中开始转导。 Cells were harvested 72 h after infection. Protein expression was checked by western blotting. We used Invitrogen NuPAGE gel electrophoresis system following the owner’s manual, and proteins were transferred onto PVDF membrane. Primary antibodies used for detecting corresponding proteins are: anti-CTCF (Cell Signaling Technology, 3418 S, dilution 1:1000), anti-RAD21 (Abcam, ab217678, dilution 1:1000), anti-HCFC1 (Santa Cruz Biotechnology, sc-390950, dilution 1:1000) and anti-ZNF143 (Abnova Corporation, H00007702-M01, dilution 1:1000).
RNA-seq文库构建gydF4y2Ba
采用QIAzol裂解试剂(QIAGEN)和RNeasy mini kit (QIAGEN)对5000个细胞进行总RNA提取和纯化。RNA-seq文库由纯化RNA按照Smart-seq2协议构建gydF4y2Ba94gydF4y2Ba.gydF4y2Ba
生成循环锚删除单元格gydF4y2Ba
设计CRISPR靶向序列,克隆至pSpCas9(BB)−2A-Puro载体(Addgene #62988)。用Cas9和sgRNA表达质粒转染K562细胞24 h后,用2 μg/mL嘌呤霉素处理细胞48 h,杀死未转染的细胞。将存活的细胞按每孔1个细胞的密度分选到96孔板中,培养2 ~ 3周,用特异性PCR引物进行基因分型,鉴定环锚缺失克隆。所使用的靶向序列如下:CEMIP2-E2, 1-GATCGAGTTCTAGTTGACCC, 2-GTGCGTCTATGAATCTGCGC;Cemip2-e3, 1-gtaagcacatggcccgtcag, 2-tcgaacaggaacgtactatc;Cemip2-e5, 1-ctaacgcaatccacctagaa, 2-taaggctctctacttagcgg;znf225启动子,1-TGGCGCTTAACGACGAACCC, 2-TTTATGGGGCACGGCGACCA;znf234启动子,1-AAGGAGGATCCTATACGTGA, 2-TAAGCCGCAACGTGACTCTG。gydF4y2Ba
公开数据和预处理gydF4y2Ba
本研究使用的公共数据,包括Hi-C、HiChIP、ChIA-PET、capture Hi-C、MCC、Micro-C、RNA-seq、ATAC-seq、DNase-seq、ChIP-seq汇总于gydF4y2Ba补充信息gydF4y2Ba.同一因子的生物重复和技术重复被合并,后续分析只使用唯一的reads。gydF4y2Ba
公开基因组注释gydF4y2Ba
人类(GENCODE .v30.basic.annotation.gtf)和老鼠(GENCODE . vm21 .basic.annotation.gtf)基因注释来自GENCODEgydF4y2Ba95gydF4y2Ba用于任何基因相关分析。本研究使用人类基因组版本hg38和小鼠基因组版本mm10。如果人类或小鼠的数据是在其他基因组版本中生成的,它们总是被转换为hg38或mm10进行分析。gydF4y2Ba
假定的人类细胞增强子和启动子注释从NIH路线图表观基因组学联盟获得gydF4y2Ba96gydF4y2Ba处理过程如下:1)收集标注为“Enh”和“Tss”的所有区域;2)重叠区域采用BEDtools合并;3)在HOMER包中使用annotatePeaks.pl对合并区域进行进一步注释gydF4y2Ba97gydF4y2Ba基因注释文件,从GENCODE下载gydF4y2Ba95gydF4y2Ba;如果一个区域位于TSS上游或下游2 kb以内,则将其定义为启动子;否则,它被定义为增强器。4)将间隙<100 bp的相邻增强子或启动子用BEDtools merge再次合并。gydF4y2Ba
假定的小鼠胚胎干细胞顺式调控元件由ATAC-seq峰定义。如果一个峰值在TSS上游或下游2 kb以内,则将其定义为启动子;否则,它被定义为增强器。gydF4y2Ba
Hi-TrAC数据预处理gydF4y2Ba
FASTQ文件中的原始对端读取首先从两端修剪连接器序列CTGTCTCTTATACACATCT。只保留两端长度≥10 bp的成对端标签(成对端标签)。使用Bowtie2将修剪过的pet映射到hg38gydF4y2Ba98gydF4y2Ba-端-端-非常敏感参数。MAPQ≥10的映射pet转换为BEDPE文件。对距离小于1kb且两端无连接子序列的映射pet进行进一步筛选。如果两端位置相同,则过滤pet的PCR重复。下游分析主要使用唯一的染色体内pet (cis pet)作为BEDPE文件。所有描述的处理步骤在cLoops2包中总结为tracPre2.pygydF4y2Ba99gydF4y2Ba.tracPre2.py还生成了质量控制统计结果。使用BEDPE文件分析PET级别的属性,cLoops2前模块将它们处理到cLoops2数据目录中,用于其他分析,如域调用、循环调用和可视化。gydF4y2Ba
Hi-TrAC或Micro-C的虚拟4C信号gydF4y2Ba
虚拟4c信号仅通过保持pet的一端位于目标tss的上游或下游1kb以内来生成。然后这些pet被堆积起来作为1D信号。该方法在cLoops2绘图模块中实现,用于可视化,或在cLoops2转储模块中实现,用于数据提取gydF4y2Ba99gydF4y2Ba.gydF4y2Ba
mESC Hi-TrAC和Micro-C回路的比较gydF4y2Ba
Micro-C循环由Juicer包中的HiCCUPS调用(v1.22.01)gydF4y2BaOne hundred.gydF4y2Ba对于从GEO下载的26亿PETs HIC文件,参数设置为-cpu -ignore-sparsity -r 2500 -f 0.1 -k KR -p 4 -i 8 -d 2,根据原始论文,与其他分辨率相比,导致了最多的循环gydF4y2Ba60gydF4y2Ba.由于HiCCUPS算法的升级,从Micro-C数据中调用的循环比原来的论文更多gydF4y2Ba60gydF4y2Ba.在cLoops中描述的循环调用算法gydF4y2Ba101gydF4y2Ba被轻微改进和实现为cLoops2 callLoops模块gydF4y2Ba99gydF4y2Ba用于Hi-TrAC数据循环调用。mESC Hi-TrAC循环由cLoops2 callLoops模块调用,参数为-eps 200,500,1000,2000 -minPts 20 -p 30 -w -j -i -max_cut -cut 5000。对于重叠分析,循环锚点扩展到5 kb,使用BEDTools (v2.29.2)包中的pairtopair子命令获得唯一的重叠循环gydF4y2Ba102gydF4y2Ba使用-type notboth或-type both选项。gydF4y2Ba
从人体细胞的Hi-TrAC数据调用循环gydF4y2Ba
cLoops2 callLoops模块的关键参数设置为-eps 200,500,1000,2000 -minPts 10 -max_cut,用于调用GM12878 Hi-TrAC数据中的循环,需要至少10个PETs支持的循环。对于K562 Hi-TrAC数据,通过参数设置-eps 200,500,1000,2000 -minPts 10 -cut 5000调用循环,过滤距离小于5kb的pet,默认参数将自动过滤所有小于20kb的循环。gydF4y2Ba
环聚合分析gydF4y2Ba
为相互作用的pet回路构造了一个11 × 11的接触矩阵,以及它的五个相同大小的上游和下游窗口。一个循环的个体富集分数计算为11 × 11接触矩阵中心的pet数量除以所有其他值的平均值。整体浓缩分数是每个循环的所有浓缩分数的平均值。对11 × 11接触矩阵进一步归一化,得到矩阵中pet的总数和z-score归一化。热图绘制了所有归一化11 × 11矩阵的平均矩阵。分析在cLoops2 agg模块中实现,并带有-loops选项。除了特别提到的参数外,使用默认参数-loop_norm生成可视化结果。gydF4y2Ba
CTCF主题方向gydF4y2Ba
全基因组CTCT基序方向由FIMO标记gydF4y2Ba103gydF4y2BaCTCF基序记录在CIS-BP数据库中gydF4y2Ba104gydF4y2Ba.gydF4y2Ba
调用差分循环gydF4y2Ba
将不同条件下的样本循环进行组合,并在两种条件下进行量化。循环锚点附近的邻近区域,即cLoops中定义的用于估计循环统计检验的排列的邻近背景区域,也被量化。两种条件下的背景数据线性拟合。假设背景数据没有差异,使用拟合的线性模型将处理数据集中循环中的pet转换为控制集。虚假发现率(FDR)是在背景数据MA图中寻找平均和折叠变化的截止点所必需的参数。然后将截断应用于转换后的循环数据。最后将泊松p值分配给每个循环如下:gydF4y2Ba
其中fg表示处理与对照测试循环中PETs的较大值,bg表示比较的较小值,fgNearby为测试条件下背景数据PETs的个数,bgNearby为对照条件下背景数据PETs的个数。Pseudo是一个通用的噪声控制值,在所有时间设置为1。这里除伪数外的所有数字都是通过背景数据上方的线性拟合进行转换的。p值通过Bonferroni校正进行校正,默认情况下0.01作为显著性的截止值。gydF4y2Ba
该算法在cLoops2 callDiffLoops模块中实现gydF4y2Ba99gydF4y2Ba, GM12878和K562 Hi-TrAC数据的关键参数为-fdr 0.05,称为差异富集环路。gydF4y2Ba
与Hi-TrAC环相关的转录因子gydF4y2Ba
集成公共ChIP-seq数据和Hi-TrAC环旨在识别与染色质环相关的转录因子。我们从ReMap 2020中收集了GM12878中162个转录因子和K562中360个转录因子的结合位点gydF4y2Ba105gydF4y2Ba(remap2020_all_macs2_hg38_v1_0.bed)在2020-08-09。对于一个环,将其左锚点上游(基因组中较小的坐标)和右锚点下游(基因组中较大的坐标)的三个大小的区域链接起来作为背景数据(假环)进行比较,以计算两个锚点上TF结合位点的富集情况。任何与真正的循环锚重叠的背景区域都被删除。将具有转录因子结合位点的锚点与背景区域的重叠部分编译成左锚点矩阵和右锚点矩阵。行是锚点或背景,列是二进制矩阵中的因子。在二进制矩阵中,1表示锚被因子绑定,0表示不绑定。利用这两个二进制矩阵,计算并使用以下属性来寻找富集的转录因子:1)锚点一致性:对于一个TF,使用来自左锚点矩阵和来自右锚点矩阵的向量来计算Spearman相关系数,表示一个因子在两个锚点处的共结合一致性。2)锚点共绑定比:对于一个TF,锚点被TF绑定的比例。3) TF峰重叠率:一个TF峰与环锚区重叠的比例。为了过滤tf,使用以下截断值:1)与背景比较,一致性比>2; 2) anchors co-binding ratio >0.1; 3) comparing to the background, the ratio of co-binding ratio >2; 4) comparing to the background, the ratio of TF peaks overlap ratio ≥ 1. Except for using the anchor flanking regions as background, we also implemented the random shuffling value 1000 times as background to ensure the observed attributes are higher than permutation background and require FDR < 0.001. The remaining TFs were sorted by consistency in descending order, by which known looping associated factors such as CTCF and cohesin are among the top-ranked factors.
对照和TF敲除K562细胞Hi-TrAC数据分析gydF4y2Ba
通过tracPre2.py对原始Hi-TrAC数据进行处理,提取出唯一的顺式pet (Supplementary data .py)gydF4y2Ba1克ydF4y2Ba).对K562 Hi-TrAC数据中调用的所有循环进行聚合循环分析,以获得富集分数,并检查两个shrna和生物重复之间的一致性。来自相同TF击倒的所有独特顺式pet被汇集在一起,并对所有下游分析进行了7400万次抽样(汇集的对照样本的pet最少,为7437万)。全局富集分数也用于显示TF敲除样本中K562 Hi-TrAC环的全部或子集的全局变化。以-noPCorr -pcut 0.001作为关键参数调用从敲除样本到对照样本的差异富集环路(所有从K562 Hi-TrAC数据调用的环路均作为比较集),通过该关键参数进行上述未校正泊松检验gydF4y2BaPgydF4y2Ba-value 0.001用于选择显著变化的循环。gydF4y2Ba
用于交互变化可视化的罗波安图gydF4y2Ba
在罗波安图中,从Hi-TrAC环路或其他具有扩展附近区域的来源推断的每个假定的顺式调控元件被显示为圆的一部分,Hi-TrAC 1D剖面显示在圆外,Hi-TrAC相互作用密度显示为拱形的宽度,或每个Hi-TrAC PET显示为圆部分之间的拱形。只能设置视点以使pet面向某些元素。我们将可视化结果命名为罗波安图,因为它看起来像西部世界第三季中名为罗波安的人工智能系统的预测分歧。这种可视化方法在cLoops2蒙太奇模块中实现gydF4y2Ba99gydF4y2Ba.gydF4y2Ba
对照和TF敲除K562细胞Hi-C数据分析gydF4y2Ba
HiC-Pro (v2.11.1)将原始Hi-C数据读取处理到人类参考基因组hg38gydF4y2Ba106gydF4y2Ba.所有后续分析仅使用HiC-Pro输出的后缀为allValidPairs的文件中的染色体内pet。使用cLoops2图形模块生成可视化图形。结合Hi-C库的重复,用聚合分析验证Hi-TrAC检测到的减少环路,考虑到Hi-C相互作用PETs的稀疏性,只使用环路和附近超过20个PETs的区域进行分析。gydF4y2Ba
分析对照和TF敲除K562细胞的RNA-seq数据gydF4y2Ba
STAR将原始RNA-seq数据读取映射到人类参考基因组hg38 (v2.7.3a)gydF4y2Ba107gydF4y2Ba.基因注释文件(v30)从GENCODE下载gydF4y2Ba95gydF4y2Ba采用袖扣法定量基因表达水平(v2.2.1)gydF4y2Ba108gydF4y2Ba.进一步,Cufflinks package中的Cuffdiff (v2.2.1)调用显着差异表达基因(击倒样本vs对照),要求gydF4y2BaPgydF4y2Ba-value <0.001,折叠变化≥1。gydF4y2Ba
ChIP-seqgydF4y2Ba
对照组和TF敲除后的K562细胞用1%甲醛在室温下固定10分钟。用1毫升冰冷的PBS清洗细胞两次,然后将细胞放在冰上。10万个固定细胞用于CTCF和RAD21 ChIP-seq文库制备,50万个固定细胞用于HCFC1和ZNF143 ChIP-seq文库制备。用含有1mm PMSF和1x蛋白酶抑制剂鸡尾酒的1x TE缓冲液重悬细胞。染色质剪切在Diagenode Bioruptor Pico超声设备上进行,温度为4°C,循环6次,开30秒,关30秒,得到200-1000 bp的片段。将染色质溶液调整为1x RIPA缓冲液(1x TE, 0.1% SDS, 0.1%脱氧胆酸钠和1% Triton X-100)加上200 mM NaCl。1.5万×离心后收集染色质上清gydF4y2BaggydF4y2Ba在4°C微量离心机中浸泡10分钟。10%的染色质上清保存作为输入。将2 μg抗体与20 μL Dynabeads Protein A珠子(ThermoFisher Scientific, Cat. 10001D)混合,室温旋转1小时。1× PBS洗珠一次,加入染色质溶液,4℃旋转孵育过夜。用RIPA缓冲液洗珠两次,然后用RIPA缓冲液加300 mM NaCl洗两次,然后用LiCl缓冲液(1x TE, 250 mM LiCl, 0.5% NP-40, 0.5%脱氧胆酸钠)洗两次,最后用1x TE缓冲液洗两次。通过蛋白酶K消化洗脱DNA和反向交联,并在65°C孵育6小时。使用MinElute Reaction Cleanup Kit (QIAGEN, Cat. 28206)净化DNA。使用End-It DNA End-Repair Kit (Lucigen, Cat。ER0720),然后用Klenow Fragment (3 ' -> 5 ' exo-) (NEB, Cat。然后用T4 DNA连接酶(NEB, Cat。 M0202L). Amplify the library and add index by PCR. DNA fragments between 200 bp and 600 bp were purified and sequenced on Illumina platforms.
实验中使用了以下抗体:抗ctcf (Cell Signaling Technology, Cat. 3418 S),抗rad21 (Abcam, Cat. 3418 S)。ab217678),抗hcfc1 (Cell Signaling Technology, Cat. 69690 S),抗znf143 (Abnova, Cat。H00007702-M01)。gydF4y2Ba
ATAC-seqgydF4y2Ba
按照报道的方案,用50,000个细胞进行ATAC-seqgydF4y2Ba109gydF4y2Ba.gydF4y2Ba
3 c-qpcrgydF4y2Ba
用1ml冷冻裂解液(10mm Tris-HCl, pH 8.0, 10mm NaCl, 0.2% NP-40, 1 ×蛋白酶抑制剂)重悬100万个固定细胞,然后在冰上孵育20分钟。4℃离心收集细胞,用稀释的CutSmart缓冲液(346 μL H)重悬gydF4y2Ba2 gydF4y2BaO, 50 μL 10× CutSmart Buffer, 44 μL 1% SDS)。在65°C孵育10分钟。加入10% Triton X-100 50 μL,在37℃恒温器上摇1小时。加入100u所选限制性内切酶(用于gydF4y2BaMYCgydF4y2Ba启动子-增强子,使用SpeI;为gydF4y2BaZNF224gydF4y2Ba-gydF4y2BaZNF284gydF4y2Ba,使用BamHI;为gydF4y2BaZNF225gydF4y2Ba-gydF4y2BaZNF235gydF4y2Ba,使用印地二;为gydF4y2BaMRPL24gydF4y2Ba-gydF4y2BaPRCCgydF4y2Ba而且gydF4y2BaNDC1gydF4y2Ba-gydF4y2BaTCEANC2gydF4y2Ba,使用NcoI),然后在37°C摇晃过夜。离心收集消化后的细胞核,用100 μL灭活缓冲液(1 × PBS, 1% SDS)重悬,65℃孵育20 min。加入895 μL稀释T4结扎缓冲液(695 μL HgydF4y2Ba2 gydF4y2BaO, 100 μL 10 × T4 DNA连接酶反应缓冲液,100 μL 10% Triton X-100)混合均匀。加入100u T4 DNA连接酶,16℃孵育过夜。加入30 μL 10% SDS和100 μg蛋白酶K停止结扎反应,65℃孵育反交联。苯酚-氯仿萃取纯化3c文库。根据相互作用对的序列设计引物和探针,用qPCR定量相互作用频率。启动子区gydF4y2BaMYCgydF4y2Ba以基因作为输入对照。使用的引物和探针有:gydF4y2BaMYCgydF4y2Ba启动子输入,正向:CTCAGCAGCAGCTCCAAATA,探针:/56-FAM/AGAGTGCTG/ZEN/CTAGAGCAACAAGCA/3IABkFQ/,反向:GACCATGGAAGTTGCCTTCT;gydF4y2BaMYCgydF4y2Ba启动子-增强子,正向:TCATTTCAGGGAGCAAACAAATC,探针:/56-FAM/ACGCTTCGA/ZEN/CTTAGCTAGTTGCCC/3IABkFQ/,反向:TTACTCTGGAATAGGTTCCATGC;gydF4y2BaZNF224gydF4y2Ba-gydF4y2BaZNF284gydF4y2Ba,正向:GACTGGTGGTCTCTTCTTAGTG,探针:/56-FAM/ATTTCCCAC/ZEN/GAAGCCTGTCAGGTC/3IABkFQ/,反向:TCGATCACCAGTTCTTTGAGG;gydF4y2BaZNF225gydF4y2Ba-gydF4y2BaZNF235gydF4y2Ba,正向:GTAGCTGGATCTCCTAGACTCA,探针:/56-FAM/AGGAGTTTC/ZEN/CAAACAACAGGCGTCT/3IABkFQ/,反向:ATTAACATTGTATCAAATATTGCTCAACCA;gydF4y2BaMRPL24gydF4y2Ba-gydF4y2BaPRCCgydF4y2Ba,正向:GCCTGGCACATACTGAATACT,探针:/56-FAM/AAAGGATAG/ZEN/GCTCTTCCCGCACC/3IABkFQ/,反向:GCGGAAAGTGGAGGTGAG;gydF4y2BaNDC1gydF4y2Ba-gydF4y2BaTCEANC2gydF4y2Ba,正向:AGACCGAGTCAAATGCTTCAG,探针:/56-FAM/TAGTCTAGG/ZEN/GCGTACAGGAGACCG/3IABkFQ/,反向:GCCTTCCTGCCTTTGAACT。gydF4y2Ba
对照和TF敲除K562细胞的ChIP-seq和ATAC-seq数据分析gydF4y2Ba
原始ChIP-seq和ATAC-seq数据读取被Bowtie2映射到人类参考基因组hg38gydF4y2Ba98gydF4y2Ba关键参数为-local -非常敏感-no-unal -no-mixed - no-discord。MAPQ > =10映射的pet被deepTools转换为规范化信号(每百万读取)为bigWig文件gydF4y2Ba110gydF4y2Ba用于可视化或聚合分析。gydF4y2Ba
主题分析gydF4y2Ba
通过HOMER包中的findMotifsGenome.pl对Hi-TrAC环锚或ChIP-seq峰进行Motif分析gydF4y2Ba97gydF4y2Ba.只有排名靠前的重要已知图案被展示出来。gydF4y2Ba
GO术语富集分析gydF4y2Ba
利用HOMER包中的findGO.pl脚本对基因进行GO项富集分析gydF4y2Ba97gydF4y2Ba,术语中需要10个以上的重叠基因,而术语中只有不到1000个基因。只有最丰富的术语排序gydF4y2BaPgydF4y2Ba-values显示。gydF4y2Ba
数据可视化gydF4y2Ba
大部分一维剖面和热图可视化是由cLoops2绘图模块显示的。网络通过NetworkX进行可视化和分析gydF4y2Ba111gydF4y2Ba.其他图由matplotlib生成gydF4y2Ba112gydF4y2Ba和seaborngydF4y2Ba113gydF4y2Ba.gydF4y2Ba
统计和再现性gydF4y2Ba
Hi-TrAC文库的样本量未采用统计学方法预估。分析中不排除任何数据。实验不是随机的。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
本研究生成的Hi-TrAC、RNA-seq、ATAC-seq、Hi-C和ChIP-seq数据已以登录代码存入GEOgydF4y2BaGSE180175gydF4y2Ba和根据加入代码的SRAgydF4y2BaSRP328503gydF4y2Ba.gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
循环调用和特定于单元格或条件的循环调用的主要分析总结在cLoops2包中,并在GitHub上免费提供文档、测试数据和更新:gydF4y2Bahttps://github.com/YaqiangCao/cLoops2gydF4y2Ba.该套餐也可在Zendo购买:gydF4y2Bahttps://zenodo.org/record/7140320#.YzsjWy-B0UEgydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
Naumova, N.等人。有丝分裂染色体的组织。gydF4y2Ba科学gydF4y2Ba342gydF4y2Ba, 948-953(2013)。gydF4y2Ba
Bonev, B. & Cavalli, G. 3D基因组的组织和功能。gydF4y2BaNat. Rev. GenetgydF4y2Ba17gydF4y2Ba, 661-678(2016)。gydF4y2Ba
罗利,M. J.和Corces, V. G. 3D基因组架构的组织原则。gydF4y2BaNat. Rev. GenetgydF4y2Ba19gydF4y2Ba, 789-800(2018)。gydF4y2Ba
吉布斯,J. H. &戴克,J.三维基因组的层次结构。gydF4y2Ba摩尔。细胞gydF4y2Ba49gydF4y2Ba, 773-782(2013)。gydF4y2Ba
自组织基因组:基因组结构和功能的原理。gydF4y2Ba细胞gydF4y2Ba183gydF4y2Ba, 28-45(2020)。gydF4y2Ba
张,H.等。有丝分裂- g1相变过程中的染色质结构动态。gydF4y2Ba自然gydF4y2Ba576gydF4y2Ba, 158-162(2019)。gydF4y2Ba
Lieberman-Aiden, E.等人。对远距离相互作用的全面测绘揭示了人类基因组的折叠原理。gydF4y2Ba科学gydF4y2Ba326gydF4y2Ba, 289-293(2009)。gydF4y2Ba
饶,S. S.等。千碱基分辨率的人类基因组3D图揭示了染色质环的原理。gydF4y2Ba细胞gydF4y2Ba159gydF4y2Ba, 1665-1680(2014)。gydF4y2Ba
罗利,m.j.等。进化保守原理预测三维染色质组织。gydF4y2Ba摩尔。细胞gydF4y2Ba67gydF4y2Ba, 837 - 852。e837(2017).
塞克斯顿等人。果蝇基因组的三维折叠和功能组织原理。gydF4y2Ba细胞gydF4y2Ba148gydF4y2Ba, 458-472(2012)。gydF4y2Ba
诺拉,e.p.等人。x失活中心调控景观的空间划分。gydF4y2Ba自然gydF4y2Ba485gydF4y2Ba, 381-385(2012)。gydF4y2Ba
狄克逊,J. R.等。通过染色质相互作用分析确定的哺乳动物基因组的拓扑结构域。gydF4y2Ba自然gydF4y2Ba485gydF4y2Ba, 376-380(2012)。gydF4y2Ba
Phillips-Cremins, J. E.等。在谱系承诺过程中,结构蛋白亚类塑造了基因组的3D组织。gydF4y2Ba细胞gydF4y2Ba153gydF4y2Ba, 1281-1295(2013)。gydF4y2Ba
Wijchers, P. J.等人。将subTAD系于不同核隔室的原因和结果。gydF4y2Ba摩尔。细胞gydF4y2Ba61gydF4y2Ba, 461-473(2016)。gydF4y2Ba
帕雷罗,V.等人。内聚蛋白在哺乳动物染色体臂上与CTCF功能相关。gydF4y2Ba细胞gydF4y2Ba132gydF4y2Ba, 422-433(2008)。gydF4y2Ba
Wendt, K. S.等。内聚蛋白通过cctc结合因子介导转录隔离。gydF4y2Ba自然gydF4y2Ba451gydF4y2Ba, 796-801(2008)。gydF4y2Ba
Zuin, J.等。内聚蛋白和CTCF对人类细胞染色质结构和基因表达有不同的影响。gydF4y2Ba国家科学院学报美国gydF4y2Ba111gydF4y2Ba, 996-1001(2014)。gydF4y2Ba
桑伯恩,A. L.等。染色质挤压解释了野生型和工程基因组中环和结构域形成的关键特征。gydF4y2Ba国家科学院学报美国gydF4y2Ba112gydF4y2Ba, e6456-e6465(2015)。gydF4y2Ba
Fudenberg, G.等人。环状挤压形成染色体结构域。gydF4y2Ba细胞的代表。gydF4y2Ba15gydF4y2Ba, 2038-2049(2016)。gydF4y2Ba
戴维森,i.f.等。DNA环被人体内聚物挤压。gydF4y2Ba科学gydF4y2Ba366gydF4y2Ba, 1338-1345(2019)。gydF4y2Ba
甘吉,等人。凝结蛋白对DNA环挤压的实时成像。gydF4y2Ba科学gydF4y2Ba360gydF4y2Ba, 102-105(2018)。gydF4y2Ba
维安,L.等人。内聚力挤压的能量学和生理影响。gydF4y2Ba细胞gydF4y2Ba173gydF4y2Ba, 1165-1178(2018)。gydF4y2Ba
金勇,史志,张海华,芬克尔斯坦,于海华,人内聚物通过环挤压压缩DNA。gydF4y2Ba科学gydF4y2Ba366gydF4y2Ba, 1345-1349(2019)。gydF4y2Ba
张杨,等。染色质环挤压在V(D)J生理重组中的基本作用。gydF4y2Ba自然gydF4y2Ba573gydF4y2Ba, 600-604(2019)。gydF4y2Ba
张欣,等。染色质环挤压在抗体类转换中的基本作用。gydF4y2Ba自然gydF4y2Ba575gydF4y2Ba, 385-389(2019)。gydF4y2Ba
戴,H. Q.等。环挤压介导RAG扫描的生理性Igh位点收缩。gydF4y2Ba自然gydF4y2Ba590gydF4y2Ba, 338-343(2021)。gydF4y2Ba
富勒伍德,m.j.等人。雌激素受体结合的人类染色质相互作用组。gydF4y2Ba自然gydF4y2Ba462gydF4y2Ba, 58-64(2009)。gydF4y2Ba
Handoko, L.等。多能细胞中ctcf介导的功能性染色质相互作用组。gydF4y2BaNat麝猫。gydF4y2Ba43gydF4y2Ba, 630-638(2011)。gydF4y2Ba
Chepelev I., Wei G., Wangsa D., Tang Q., Zhao K.全基因组增强子-启动子相互作用的特征揭示了相互作用基因的共表达和高阶染色质组织模式。gydF4y2Ba细胞Res。gydF4y2Ba22gydF4y2Ba, 490-503(2012)。gydF4y2Ba
德马雷,L. E.等。内聚相关染色质相互作用的基因组景观。gydF4y2Ba基因组Res。gydF4y2Ba23gydF4y2Ba, 1224-1234(2013)。gydF4y2Ba
唐恩,J. M.等。细胞识别基因的控制发生在哺乳动物染色体的绝缘区。gydF4y2Ba细胞gydF4y2Ba159gydF4y2Ba, 374-387(2014)。gydF4y2Ba
唐哲,等。ctcf介导的人类3D基因组结构揭示了转录的染色质拓扑结构。gydF4y2Ba细胞gydF4y2Ba163gydF4y2Ba, 1611-1627(2015)。gydF4y2Ba
贾维尔,B. M.等。谱系特异性基因组结构将增强子和非编码疾病变体与目标基因启动子联系起来。gydF4y2Ba细胞gydF4y2Ba167gydF4y2Ba, 1369 - 1384。e1319(2016).
季欣,等。人多能性细胞的三维染色体调控图景。gydF4y2Ba细胞干细胞gydF4y2Ba18gydF4y2Ba, 262-275(2016)。gydF4y2Ba
Smith, E. M., Lajoie, B. R., Jain, G. & Dekker, J.不变TAD边界限制了启动子和CFTR位点周围远端元件之间的细胞类型特异性环路相互作用。gydF4y2Ba点。j .的嗡嗡声。麝猫gydF4y2Ba98gydF4y2Ba, 185-201(2016)。gydF4y2Ba
深谷,T.,林,B.和莱文,M.增强子控制转录爆发。gydF4y2Ba细胞gydF4y2Ba166gydF4y2Ba, 358-368(2016)。gydF4y2Ba
安德烈,G.等。对发育肢体中数百个调控位点的描述揭示了染色质折叠的两种机制。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 223-233(2017)。gydF4y2Ba
弗隆,E. E. M.莱文,M.发育增强子和染色体拓扑结构。gydF4y2Ba科学gydF4y2Ba361gydF4y2Ba, 1341-1345(2018)。gydF4y2Ba
Kojic, A.等。内聚体- sa1和内聚体- sa2在三维染色体组织中的不同作用。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba25gydF4y2Ba, 496-504(2018)。gydF4y2Ba
Schoenfelder, S. & Fraser, P.基因表达控制中的远程增强子-启动子接触。gydF4y2BaNat. Rev. GenetgydF4y2Ba20.gydF4y2Ba, 437-455(2019)。gydF4y2Ba
金,S. & Shendure, J.转录因子与3d基因组相互作用的机制。gydF4y2Ba摩尔。细胞gydF4y2Ba76gydF4y2Ba, 306-319(2019)。gydF4y2Ba
Benabdallah, n.s.等。增强子-启动子接近度降低伴随增强子激活。gydF4y2Ba摩尔。细胞gydF4y2Ba76gydF4y2Ba, 473-484 e477(2019)。gydF4y2Ba
哦,s等人。增强子释放和重定向激活疾病易感性基因。gydF4y2Ba自然gydF4y2Bahttps://doi.org/10.1038/s41586-021-03577-1gydF4y2Ba(2021)。gydF4y2Ba
罗布森,M. I.,林格尔,A. R.和芒德洛斯,S.监管景观:增强者-促进者沟通如何在3D中雕刻。gydF4y2Ba摩尔。细胞gydF4y2Ba74gydF4y2Ba, 1110-1122(2019)。gydF4y2Ba
van Arensbergen, J., van Steensel, B. & Bussemaker, H. J.在寻找增强子-启动子相互作用特异性的决定因素。gydF4y2Ba细胞生物学趋势。gydF4y2Ba24gydF4y2Ba, 695-702(2014)。gydF4y2Ba
Haberle, V. & Stark, A.真核核心启动子和转录起始的功能基础。gydF4y2Ba细胞生物学。gydF4y2Ba19gydF4y2Ba, 621-637(2018)。gydF4y2Ba
李,Y.等。内聚- ctcf锚定环的结构基础。gydF4y2Ba自然gydF4y2Ba578gydF4y2Ba, 472-476(2020)。gydF4y2Ba
诺拉,e.p.等人。CTCF的靶向降解使染色体区域的局部绝缘从基因组区室化中分离。gydF4y2Ba细胞gydF4y2Ba169gydF4y2Ba, 930 - 944。e922(2017).
哈赫伊斯,J. H. I.等。内聚蛋白释放因子WAPL限制染色质环的延伸。gydF4y2Ba细胞gydF4y2Ba169gydF4y2Ba, 693 - 707。e614(2017).
饶,s.s.p.等。内聚蛋白的丢失消除了所有环域。gydF4y2Ba细胞gydF4y2Ba171gydF4y2Ba, 305 - 320。e324(2017).
Heidari, N.等。人类基因组中调控相互作用的全基因组图谱。gydF4y2Ba基因组ResgydF4y2Ba24gydF4y2Ba, 1905-1917(2014)。gydF4y2Ba
贝利,S. D.等。ZNF143提供序列特异性以确保基因启动子上染色质相互作用。gydF4y2BaCommun Nat。gydF4y2Ba2 gydF4y2Ba, 6186(2015)。gydF4y2Ba
周,Q.等。ZNF143介导ctcf结合启动子-增强子环,需要小鼠造血干细胞和祖细胞功能。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 43(2021)。gydF4y2Ba
Weintraub, a.s.等人。YY1是增强子-启动子循环的结构调节因子。gydF4y2Ba细胞gydF4y2Ba171gydF4y2Ba, 1573 - 1588。e1528(2017).
Beagan, J. A.等。YY1和CTCF在早期神经谱系承诺期间协调一个3D染色质循环开关。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 1139-1152(2017)。gydF4y2Ba
谢,D.等。人体细胞中动态反式作用因子共定位。gydF4y2Ba细胞gydF4y2Ba155gydF4y2Ba, 713-724(2013)。gydF4y2Ba
Pan, x等。YY1控制Igkappa基因库和b细胞发育,并在Igkappa基因位点上与凝集蛋白一起定位。gydF4y2BaEMBO J。gydF4y2Ba32gydF4y2Ba, 1168-1182(2013)。gydF4y2Ba
李,L.等。YY1与鸟嘌呤四重复合物相互作用,调节DNA环和基因表达。gydF4y2BaNat,化学。医学杂志。gydF4y2Ba17gydF4y2Ba, 161-168(2021)。gydF4y2Ba
Lai, B.等。track -loop检测基因组结构和染色质可达性。gydF4y2BaNat方法。gydF4y2Ba15gydF4y2Ba, 741-747(2018)。gydF4y2Ba
谢,T. S.等。解析与转录相关的哺乳动物染色质折叠的3D景观。gydF4y2Ba摩尔。细胞gydF4y2Ba78gydF4y2Ba, 539-553 e538(2020)。gydF4y2Ba
克里滕斯坦等人。哺乳动物染色体结构的超微结构细节。gydF4y2Ba摩尔。细胞gydF4y2Ba78gydF4y2Ba, 554 - 565。e557(2020).
Hua, P.等。在碱基对分辨率下定义基因组结构。gydF4y2Ba自然gydF4y2Bahttps://doi.org/10.1038/s41586-021-03639-4gydF4y2Ba(2021)。gydF4y2Ba
Grubert, F.等人。人类基因组中内聚素介导的染色质环的景观。gydF4y2Ba自然gydF4y2Ba583gydF4y2Ba, 737-743(2020)。gydF4y2Ba
Mifsud, B.等人。用高分辨率捕获Hi-C绘制人类细胞中的远程启动子接触。gydF4y2BaNat麝猫。gydF4y2Ba47gydF4y2Ba, 598-606(2015)。gydF4y2Ba
Mumbach, m.r.等人。原代人类细胞中的增强子连接组可识别疾病相关DNA元件的靶基因。gydF4y2BaNat麝猫。gydF4y2Ba49gydF4y2Ba1602-1612(2017)。gydF4y2Ba
Mumbach, m.r.等人。HiChIP:高效、灵敏的蛋白质定向基因组结构分析。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 919-922(2016)。gydF4y2Ba
魏,X.等。HiCAR是一种稳健而灵敏的分析开放染色质相关基因组组织的方法。gydF4y2Ba摩尔。细胞gydF4y2Ba82gydF4y2Ba, 1225 - 1238。e1226(2022).
Fulco, c.p.等。用CRISPR干扰系统映射功能增强子-启动子连接。gydF4y2Ba科学gydF4y2Ba354gydF4y2Ba, 769-773(2016)。gydF4y2Ba
胡宁,L. & Kunkel, G. R.无处不在的转录蛋白ZNF143激活多种基因,同时协助组织染色质结构。gydF4y2Ba基因gydF4y2Ba769gydF4y2Ba, 145205(2021)。gydF4y2Ba
是的,B.等人。ZNF143在染色质环和基因调控中的作用。gydF4y2Ba麝猫面前gydF4y2Ba11gydF4y2Ba, 338(2020)。gydF4y2Ba
Myslinski, E, Gerard, M. A, Krol, A. & Carbon, P.人类细胞周期调控BUB1B基因的转录需要hStaf/ZNF143。gydF4y2Ba核酸测定。gydF4y2Ba35gydF4y2Ba, 3453-3464(2007)。gydF4y2Ba
Halbig, K. M., Lekven, a.c. & Kunkel, G. R.转录激活因子ZNF143对斑马鱼的正常发育至关重要。gydF4y2BaBMC Mol. Biol。gydF4y2Ba13gydF4y2Ba, 3(2012)。gydF4y2Ba
ngono - mbongo, R. P., Myslinski, E., Aster, J. C. & Carbon, P. ZNF143, Notch1和THAP11通过重叠结合位点调控基因表达。gydF4y2Ba核酸测定。gydF4y2Ba41gydF4y2Ba, 4000-4014(2013)。gydF4y2Ba
Parker, J. B., Yin, H., Vinckevicius, A. & Chakravarti, D.宿主细胞因子-1募集到e2f结合和细胞周期控制基因是由THAP11和ZNF143介导的。gydF4y2Ba细胞的代表。gydF4y2Ba9gydF4y2Ba, 967-982(2014)。gydF4y2Ba
汉考克,m.l.等。胰岛素受体与启动子结合并调节基因表达。gydF4y2Ba细胞gydF4y2Ba177gydF4y2Ba, 722-736 e722(2019)。gydF4y2Ba
陈,T.等。Hcfc1和Ronin突变可导致先天性钴胺素代谢错误和核糖体病。gydF4y2BaCommun Nat。gydF4y2Ba13gydF4y2Ba134(2022)。gydF4y2Ba
Izumi, H.等。ZNF143通过转录调控DNA复制和细胞周期相关基因在肿瘤生长中的作用gydF4y2Ba癌症科学。gydF4y2Ba101gydF4y2Ba, 2538-2545(2010)。gydF4y2Ba
Kawatsu, Y.等。ZNF143的强表达和高MIB-1标记指数的联合独立预测肺腺癌的疾病特异性生存时间较短。gydF4y2BaBr。j .癌症gydF4y2Ba110gydF4y2Ba, 2583-2592(2014)。gydF4y2Ba
白安瑞,蔡柏凯,洪,叶凯,杨海燕。锌指蛋白143的缺失与结肠癌中白细胞介素8- cxcr轴的关系。gydF4y2Ba细胞生物学。gydF4y2Ba23gydF4y2Ba, 4043-4053(2019)。gydF4y2Ba
张,L.等。在肝细胞癌中,znf143介导的H3K9三甲基化通过激活MDIG上调CDC6。gydF4y2Ba癌症Res。gydF4y2Ba80gydF4y2Ba, 2599-2611(2020)。gydF4y2Ba
Myslinski, E, Gerard, M. A, Krol, A. & Carbon, P.对人类Staf/ZNF143结合位点的基因组规模定位分析表明,人类Staf/ZNF143在哺乳动物启动子中具有广泛的作用。gydF4y2Ba生物。化学。gydF4y2Ba281gydF4y2Ba, 39953-39962(2006)。gydF4y2Ba
米肖,J.等人。HCFC1是活跃的人类cpg岛启动子的常见成分,与ZNF143、THAP11、YY1和GABP转录因子的占用相一致。gydF4y2Ba基因组Res。gydF4y2Ba23gydF4y2Ba, 907-916(2013)。gydF4y2Ba
Vinckevicius, A. Parker, J. B. & Chakravarti . THAP11/ZNF143/HCFC1复合物招募到染色质的基因组决定因素。gydF4y2Ba细胞生物学。gydF4y2Ba35gydF4y2Ba, 4135-4146(2015)。gydF4y2Ba
Whalen, S., Truty, R. M. & Pollard, K. S.增强子-启动子相互作用由循环染色质上的复杂基因组签名编码。gydF4y2BaNat麝猫。gydF4y2Ba48gydF4y2Ba, 488-496(2016)。gydF4y2Ba
Mourad, R. & Cuvier, O.结构蛋白和绝缘体的无tad分析。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, e27(2018)。gydF4y2Ba
Wilber, A. Nienhuis, A. W. & Persons, D. A.胎儿到成人血红蛋白转换的转录调节:新的治疗机会。gydF4y2Ba血gydF4y2Ba117gydF4y2Ba, 3945-3953(2011)。gydF4y2Ba
桑卡兰,V. G. & Orkin, S. H.从胎儿到成人血红蛋白的转换。gydF4y2Ba冷泉港。教谕。地中海。gydF4y2Ba3.gydF4y2Ba, a011643(2013)。gydF4y2Ba
伍茨,G.等人。拓扑相关结构域和染色质环依赖于内聚蛋白,并由CTCF、WAPL和PDS5蛋白调控。gydF4y2BaEMBO J。gydF4y2Ba36gydF4y2Ba, 3573-3599(2017)。gydF4y2Ba
施瓦泽,W.等人。两种独立的染色质组织模式显示内聚蛋白去除。gydF4y2Ba自然gydF4y2Ba551gydF4y2Ba, 51-56(2017)。gydF4y2Ba
Kubo, N.等。启动子-近端CTCF结合促进远端增强子依赖的基因激活。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba28gydF4y2Ba, 152-161(2021)。gydF4y2Ba
李,G.等。广泛的启动子中心染色质相互作用为转录调控提供了拓扑基础。gydF4y2Ba细胞gydF4y2Ba148gydF4y2Ba, 84-98(2012)。gydF4y2Ba
吴,Y.等。从表观基因组数据的遗传分析预测启动子锚定染色质相互作用。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 2061(2020)。gydF4y2Ba
Eun, B., Sampley, M. L., Good, a . L., Gebert, C. M. & Pfeifer, K.通过共享增强子的启动子互扰解释了Nctc1在Igf2/H19/Nctc1印迹位点的父系偏置表达。gydF4y2Ba核酸测定。gydF4y2Ba41gydF4y2Ba, 817-826(2013)。gydF4y2Ba
皮切利等人。使用Smart-seq2从单细胞获得全长RNA-seq。gydF4y2BaProtoc Nat。gydF4y2Ba9gydF4y2Ba, 171-181(2014)。gydF4y2Ba
弗兰克什,A.等人。人类和小鼠基因组的GENCODE参考注释。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, d766-d773(2019)。gydF4y2Ba
表观基因组学,C.等。111个参考人类表观基因组的综合分析。gydF4y2Ba自然gydF4y2Ba518gydF4y2Ba, 317-330(2015)。gydF4y2Ba
海因茨,S.等。谱系决定转录因子的简单组合是巨噬细胞和B细胞鉴定所需的主要顺式调节元件。gydF4y2Ba摩尔。细胞gydF4y2Ba38gydF4y2Ba, 576-589(2010)。gydF4y2Ba
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 357-u354(2012)。gydF4y2Ba
曹勇,刘胜,任国强,唐强,赵凯。cLoops2:染色质相互作用的全堆栈综合分析工具。gydF4y2Ba核酸测定。gydF4y2Ba50gydF4y2Ba, 57-71(2022)。gydF4y2Ba
杜兰德,n.c.等人。Juicer提供了一个一键式系统,用于分析循环分辨率hi-c实验。gydF4y2Ba细胞系统。gydF4y2Ba3.gydF4y2Ba, 95-98(2016)。gydF4y2Ba
曹毅,等。使用cLoops精确循环调用3D基因组数据。gydF4y2Ba生物信息学gydF4y2Ba36gydF4y2Ba, 666-675(2020)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
格兰特,C. E.贝利,T. L.和诺布尔,W. S. FIMO:扫描给定母题的出现。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 1017-1018(2011)。gydF4y2Ba
Weirauch, m.t.等人。真核转录因子序列特异性的测定与推断。gydF4y2Ba细胞gydF4y2Ba158gydF4y2Ba, 1431-1443(2014)。gydF4y2Ba
切尼比,J.等人。ReMap 2020:来自人类和拟南芥dna结合测序实验综合分析的调控区域数据库。gydF4y2Ba核酸测定。gydF4y2Ba48gydF4y2Ba, d180-d188(2020)。gydF4y2Ba
仆人,N.等。HiC-Pro:用于Hi-C数据处理的优化灵活管道。gydF4y2Ba基因组医学杂志。gydF4y2Ba16gydF4y2Ba, 259(2015)。gydF4y2Ba
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 15-21(2013)。gydF4y2Ba
特拉普内尔,C.等。TopHat和Cufflinks RNA-seq实验的差异基因和转录本表达分析。gydF4y2BaProtoc Nat。gydF4y2Ba7gydF4y2Ba, 562-578(2012)。gydF4y2Ba
Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y. & Greenleaf, W. J.天然染色质转座用于开放染色质,dna结合蛋白和核小体位置的快速和敏感表观基因组分析。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba, 1213-1218(2013)。gydF4y2Ba
Ramirez, F. et al. deepTools2:用于深度测序数据分析的下一代web服务器。gydF4y2Ba核酸测定。gydF4y2Ba44gydF4y2Ba, w160-w165(2016)。gydF4y2Ba
哈格伯格,A,斯瓦特,P. & S Chult, D.使用NetworkX探索网络结构、动态和功能。(洛斯阿拉莫斯国家实验室(LANL),洛斯阿拉莫斯,NM(美国),2008)。gydF4y2Ba
亨特,J. D. Matplotlib:一个2D图形环境。gydF4y2Ba第一版。科学。Eng。gydF4y2Ba9gydF4y2Ba, 90-95(2007)。gydF4y2Ba
M. L. Seaborn:统计数据可视化。gydF4y2BaJ.开源软件。gydF4y2Ba6gydF4y2Ba, 3021(2021)。gydF4y2Ba
Di Stefano, B.等。C/EBPalpha通过上调Klf4和增加Lsd1和Brd4的水平来创造iPSC重编程的精英细胞。gydF4y2Ba细胞生物学。gydF4y2Ba18gydF4y2Ba, 371-381(2016)。gydF4y2Ba
人类基因组中DNA元素的综合百科全书。gydF4y2Ba自然gydF4y2Ba489gydF4y2Ba, 57-74(2012)。gydF4y2Ba
确认gydF4y2Ba
我们感谢NHLBI DNA测序核心设施对文库进行测序;NHLBI流式细胞仪用于细胞分类。我们感谢沃伦·伦纳德博士对手稿的批判性阅读。这项工作得到了国家心脏、肺和血液研究所校内研究部和4DN变革合作项目奖(A-0066) (KZ)的支持。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
K.Z.构想了这个项目。S.L.设计并执行了实验。Y.C.分析了数据。K.C.参与了实验,Q.T.参与了实验设计。s.l., Y.C和K.Z.写了这篇论文。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然通讯gydF4y2Ba感谢匿名审稿人对本工作的同行评议所作的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
刘胜,曹勇,崔凯,刘志强。gydF4y2Baet al。gydF4y2BaHi-TrAC揭示了转录因子在组织染色质环中的分工。gydF4y2BaNat CommungydF4y2Ba13gydF4y2Ba, 6679(2022)。https://doi.org/10.1038/s41467-022-34276-8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467-022-34276-8gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba