摘要gydF4y2Ba
的gydF4y2Ba葱属植物gydF4y2Ba属植物作为蔬菜、调味品或药用植物在全球范围内种植,其特点是基因组大,刺激性强。然而,基因组进化和其独特风味形成的基因组基础仍然知之甚少。在此,我们报道了一个11.27 gb的染色体尺度的洋葱基因组组装。gydF4y2Ba答:fistulosumgydF4y2Ba)。长末端重复序列的不均匀爆发促进了基因组成分的多样性,分散的重复事件在很大程度上解释了基因扩展gydF4y2Ba葱属植物gydF4y2Ba基因组。蒜氨酸酶和催泪因子合成酶的广泛复制和分化是植物风味形成过程中的重要进化事件gydF4y2Ba葱属植物gydF4y2Ba作物。此外,风味相关基因的不同选择偏好可能导致洋葱中异蒜素含量的变化。此外,我们揭示了中国是洋葱束的起源和驯化中心。我们的发现提供了一些见解gydF4y2Ba葱属植物gydF4y2Ba基因组进化,风味形成和驯化历史,并使未来基因组辅助育种这些作物的重要性状。gydF4y2Ba
介绍gydF4y2Ba
的gydF4y2Ba葱属植物gydF4y2Ba属在地理上自然分布于北半球,多样性的主要中心从地中海盆地到中亚gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba。对季节性干旱和恶劣环境具有较强的适应性。意识到…的潜力gydF4y2Ba葱属植物gydF4y2Ba促进人类健康的蔬菜正在推动对这些功能作物的需求不断增加。此外,gydF4y2Ba葱属植物gydF4y2Ba属以一组独特的从半胱氨酸中提取的亚砜而闻名gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,这有助于其独特的风味,药用特性,抗病和驱虫gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba。与糖、有机酸、生物碱和萜烯衍生代谢物等与质量相关的成分相比,人们对其生物合成和调控的了解较少gydF4y2Ba葱属植物gydF4y2Ba-特异性烷基半胱氨酸亚砜(acso)gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba如蒜素、异蒜素和蛋氨酸。在主要的acso中gydF4y2Ba葱属植物gydF4y2Ba异蒜素可以在蒜氨酸酶(ALL)和催泪因子合成酶(LFS)的作用下水解成丙硫醚s -氧化物(俗称催泪因子)。gydF4y2Ba3.gydF4y2Ba而在组织损伤时,蒜素可以自发催化成蒜素gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba11gydF4y2Ba。然而,这种独特风味形成的基因组基础在gydF4y2Ba葱属植物gydF4y2Ba农作物目前还不清楚。gydF4y2Ba
的gydF4y2Ba葱属植物gydF4y2Ba属拥有一些主要的经济作物,包括束洋葱、威尔士洋葱或层洋葱(gydF4y2Ba答:fistulosumgydF4y2Ba)、洋葱(gydF4y2Ba答:cepagydF4y2Ba)、大蒜(gydF4y2Ba答:一种gydF4y2Ba)和青葱(gydF4y2Ba答:ascalonicumgydF4y2Ba)。由于它们形态上的相似性、较弱的生殖隔离和相似的染色体核型,它们之间的遗传关系gydF4y2Ba葱属植物gydF4y2Ba物种没有很好的定义gydF4y2Ba2gydF4y2Ba,gydF4y2Ba12gydF4y2Ba。传统的gydF4y2Ba葱属植物gydF4y2Ba分类学主要基于植物形态gydF4y2Ba9gydF4y2Ba,gydF4y2Ba13gydF4y2Ba。例如,gydF4y2Ba答:fistulosumgydF4y2Ba一般根据其分蘖特征和假茎形态分类。葱(gydF4y2Ba答:ascalonicumgydF4y2Ba因为它们的分蘖特征相似,所以经常被误认为是簇洋葱。中国红洋葱(gydF4y2Ba答:cepagydF4y2Bavar。gydF4y2BaproliferumgydF4y2Ba)常与洋葱(gydF4y2Ba答:fistulosumgydF4y2Bal . vargydF4y2Ba。viviparumgydF4y2Ba),因为它们的球茎相似。这些模棱两可gydF4y2Ba葱属植物gydF4y2Ba分类学经常导致对该属的进化和驯化的误解,这需要在基因组水平上加以澄清。gydF4y2Ba
自然或人工种间杂交是普遍存在的gydF4y2Ba葱属植物gydF4y2Ba属,已被经验和随机利用来引入抗病和品质性状gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba。基因组信息的种间杂交可能是一种有效的扩展策略gydF4y2Ba葱属植物gydF4y2Ba理想农艺性状的遗传多样性。然而,gydF4y2Ba葱属植物gydF4y2Ba属因其具有高比例重复序列的大基因组而臭名昭着,这导致该属缺乏高质量的基因组组装gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba。因此,这些重要作物的基因组辅助育种落后于基因组相对较小的作物。因此,基因组信息是非常需要和可取的gydF4y2Ba葱属植物gydF4y2Ba研究和繁殖群落。gydF4y2Ba
在这里,我们报告了染色体尺度的参考基因组组装gydF4y2Ba答:fistulosumgydF4y2Ba通过结合PacBio、Bionano、HiC和Illumina测序技术工具包获得。比较基因组学和基因组共线性分析有力地阐明了基因组进化和亲缘关系gydF4y2Ba葱属植物gydF4y2Ba作物。此外,我们研究了参与ACSOs生物合成和水解的基因的进化,以阐明特殊风味形成的基因组机制gydF4y2Ba葱属植物gydF4y2Ba作物。此外,135年gydF4y2Ba葱属植物gydF4y2Ba重新排序的加入,以澄清系统基因组进化和迁移路线gydF4y2Ba葱属植物gydF4y2Ba作物。此外,转录组学和代谢分析进行了探索的形成含硫香精gydF4y2Ba葱属植物gydF4y2Ba作物。本研究提供了基因组进化和扩展的见解gydF4y2Ba葱属植物gydF4y2Ba物种和将使基因组辅助育种。gydF4y2Ba
结果gydF4y2Ba
染色体尺度的大基因组组装和基因注释gydF4y2Ba
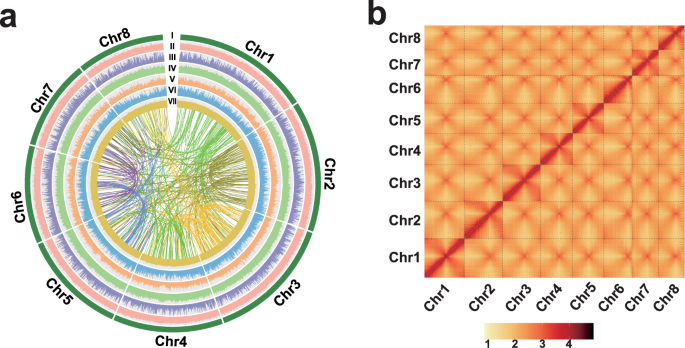
我们组装了洋葱的染色体水平基因组(gydF4y2Ba答:fistulosumgydF4y2Ba),对洋葱地方品种进行从头开始的基因组测序。首先,gydF4y2Bak-mergydF4y2Ba分析(gydF4y2BakgydF4y2Ba= 17)显示,估计基因组大小为11.97千兆碱基对(Gb),杂合率为0.64%,重复序列占整个基因组的89.89%(补充图17)。gydF4y2Ba1gydF4y2Ba及补充表gydF4y2Ba2gydF4y2Ba)。生成PacBio序列(1,649.82 Gb, 138.1 ×基因组覆盖率),将基因组组装成contigs,得到27,972个contigs的草图,总长度为15.36 Gb, contig N50为4.72兆碱基对(Mb, Supplementary Table)gydF4y2Ba1gydF4y2Ba)。经过抛光和质量改进,得到了改进后的组件(支架N50, 8.98 Mb)(表1)gydF4y2Ba1gydF4y2Ba及补充表gydF4y2Ba3.gydF4y2Ba)。最后,利用Hi-C互作数据集(1136.4 Gb, 95.1 ×基因组覆盖率)将基因组构建成超级支架,98.71%的组装contigs被锚定在8条染色体上(图2)。gydF4y2Ba1 bgydF4y2Ba及补充表gydF4y2Ba1gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba- - - - - -gydF4y2Ba5gydF4y2Ba)。最终基因组组装量为11.27 Gb,序列N50为7.34 Mb,超级支架N50为1.34 Gb(图2)。gydF4y2Ba1gydF4y2Ba、表gydF4y2Ba1gydF4y2Ba,及补充表格gydF4y2Ba3.gydF4y2Ba-gydF4y2Ba4gydF4y2Ba),表示当前发布的数据之间的最佳邻接gydF4y2Ba葱属植物gydF4y2Ba基因组装配。gydF4y2Ba
多个基因组评估支持高质量的gydF4y2Ba答:fistulosumgydF4y2Ba基因组组装。首先,Benchmarking Universal Single-Copy Orthologue (BUSCO,占1614个核心真核基因的91.0%)和core eukaryotic Gene Mapping Approach (CEGMA,占94.35%的保守基因)分析表明,组装的基因组具有很高的完整性(Supplementary Tables)gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba)。同时,对一致性质量值(QV)和完整性进行了分析gydF4y2Ba答:fistulosumgydF4y2Ba分别为37.06%和93.61%,表明我们的装配精度较高gydF4y2Ba20.gydF4y2Ba。通过将Illumina短读数定位到基因组组装上,我们获得了99.65%的定位率,99.34%的基因组覆盖率和0.0063%的SNP率,证明了组装基因组的高质量(补充表)gydF4y2Ba8gydF4y2Ba)。此外,我们观察到以前的遗传图谱之间存在良好的共线关系gydF4y2Ba21gydF4y2Ba和组装的染色体(补充图2)。gydF4y2Ba2gydF4y2Ba),这进一步支持了我们高质量的基因组组装。尽管基因组大小较大,但高的长端重复反转录转座子组装指数(LAI, 24.81)表明该基因具有黄金连续性gydF4y2Ba答:fistulosumgydF4y2Ba基因组组装gydF4y2Ba22gydF4y2Ba。gydF4y2Ba
使用RepeatMasker、TRF和RepeatProteinMask(补充表)对7885 Mb(占总基因组的69.81%)重复序列进行注释gydF4y2Ba9gydF4y2Ba)。大多数重复序列是转座元件(te),而长端重复(LTR)逆转录转座子是最丰富的tegydF4y2Ba答:fistulosumgydF4y2Ba基因组(占总基因组的62.18%;补充图。gydF4y2Ba3gydF4y2Ba、补充表gydF4y2Ba10gydF4y2Ba)。使用从头基因预测、基于蛋白质的同源性搜索和基于转录组的预测的互补方法已用于注释蛋白质编码序列。共预测基因62255个,平均基因长度为5000.13碱基对(bp),平均基因编码序列长度为820.11 bp,平均每个基因有3.93个外显子,平均外显子长度为208.47 bp,基因注释率为98.30%(补充表)gydF4y2Ba11gydF4y2Ba- - - - - -gydF4y2Ba13gydF4y2Ba)。此外,在62,259个预测基因中,约有四分之一(14,862个)被ltr插入或与ltr重叠。gydF4y2Ba3 bgydF4y2Ba)。据报道,LTR在基因的转导、复制和重组以及选择性剪接和表观遗传控制中具有潜在的作用gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba。因此,我们假设基因复制或移动gydF4y2Ba答:fistulosumgydF4y2Ba可能随着ltr的增殖和转位而发生。我们还鉴定了1361个miRNA, 4365个tRNA, 2832个rRNA和3444个snrnagydF4y2Ba答:fistulosumgydF4y2Ba使用tRNAscan-SE管道进行基因组测序(补充表)gydF4y2Ba14gydF4y2Ba)。gydF4y2Ba
葱属植物gydF4y2Ba基因组进化与染色体同源性gydF4y2Ba
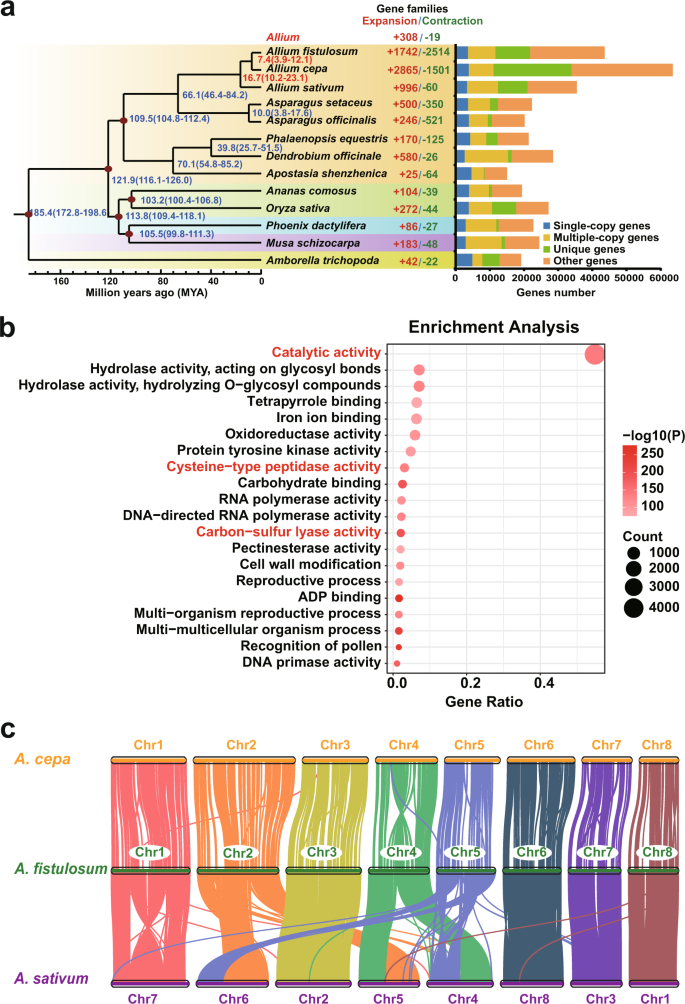
探索…的基因组进化gydF4y2Ba葱属植物gydF4y2Ba,从中选取了13种gydF4y2BaAsparagalegydF4y2Ba,gydF4y2Ba棕榈目gydF4y2Ba,gydF4y2Ba禾本目gydF4y2Ba,gydF4y2BaAmborellaceaegydF4y2Ba,gydF4y2BaScitamineaegydF4y2Ba用于进一步的比较基因组分析。在这些植物中,我们发现了三种gydF4y2Ba葱属植物gydF4y2Ba作物的总基因家族和独特基因家族数量最多(图2)。gydF4y2Ba2gydF4y2Ba)。共选取492个单拷贝基因家族进行最大似然树重构,结果表明gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba大约740万年前(MYA)分化,而共同的祖先是gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba背离gydF4y2Ba答:一种gydF4y2Ba约16.7亿年前gydF4y2Ba11gydF4y2Ba,gydF4y2Ba25gydF4y2Ba(无花果。gydF4y2Ba2gydF4y2Ba)。在选定的基因组中gydF4y2Ba葱属植物gydF4y2Ba作物表现出大量扩增和收缩基因,共扩增308个基因家族gydF4y2Ba葱属植物gydF4y2Ba作物。重要的是,我们发现这些基因家族主要富集于基因本体(GO)途径,如“催化活性”、“碳-硫裂解酶活性”和“半胱氨酸型肽酶活性”,这意味着高硫代谢活性gydF4y2Ba葱属植物gydF4y2Ba作物负责与风味形成相关的硫化合物的生物合成(图2)。gydF4y2Ba2 a, bgydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba13种植物的系统发育树及其基因家族的进化。每个节点旁边的数值表示每个节点的估计发散时间。点上的红点表示存在支持估计时间的化石证据。中间的数字表示扩大和缩小的基因家族。右图显示了单拷贝、多拷贝、唯一和其他同源物的基因数目。gydF4y2BabgydF4y2Ba共享扩增基因的前20个富集基因本体术语有3个gydF4y2Ba葱属植物gydF4y2Ba作物。采用卡方检验计算gydF4y2BaPgydF4y2Ba当所有期望频率都大于5时,基因项的-值,否则,我们使用Fisher精确检验来计算gydF4y2BaPgydF4y2Ba值。gydF4y2BacgydF4y2Ba三个基因组共线性gydF4y2Ba葱属植物gydF4y2Ba作物。源数据作为源数据文件提供。gydF4y2Ba
的分布gydF4y2BaKsgydF4y2Ba的同区同源对的值gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:一种gydF4y2Ba是否与…相似gydF4y2Ba答:一种gydF4y2Ba和gydF4y2Ba芦笋officinalisgydF4y2Ba11gydF4y2Ba(补充图。gydF4y2Ba4gydF4y2Ba),这表明,与大蒜类似,gydF4y2Ba答:fistulosumgydF4y2Ba也经历了三次WGD事件。此外,大多数基因的三个主要gydF4y2Ba葱属植物gydF4y2Ba在共线性分析中,作物对应于单个块。因此,这三个基因组在分化后没有发生独特的WGD事件(补充图2)。gydF4y2Ba5gydF4y2Ba)。此前,二者之间的宏观协同程度很高gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba是否有报道使用103个锚点标记gydF4y2Ba21gydF4y2Ba。在这里,我们观察到在染色体水平上几乎是一对一的同型关系,除了Chr4,它在两者之间经历了明显的反转gydF4y2Ba葱属植物gydF4y2Ba作物(图。gydF4y2Ba2摄氏度gydF4y2Ba,补充图gydF4y2Ba6gydF4y2Ba)。此外,四gydF4y2Ba答:fistulosumgydF4y2Ba染色体(Chr3, Chr6, Chr7,和Chr8)与基因的染色体表现出几乎一对一的同质关系gydF4y2Ba答:一种gydF4y2Ba(Chr2, Chr8, Chr3, and Chr1;无花果。gydF4y2Ba2摄氏度gydF4y2Ba,补充图gydF4y2Ba6gydF4y2Ba)。剩下的四条染色体表现出几次重排。的Chr1之间有明显的反转gydF4y2Ba答:fistulosumgydF4y2Ba和Chr7gydF4y2Ba答:一种gydF4y2Ba, Chr2gydF4y2Ba答:fistulosumgydF4y2Ba与?的Chr5和Chr6部分共合gydF4y2Ba答:一种gydF4y2Ba, ch4gydF4y2Ba答:fistulosumgydF4y2Ba与Chr4和Chr5呈部分合型关系gydF4y2Ba答:一种gydF4y2Ba和的Chr5gydF4y2Ba答:fistulosumgydF4y2Ba由?的Chr4和Chr6部分重排gydF4y2Ba答:一种gydF4y2Ba(无花果。gydF4y2Ba2摄氏度gydF4y2Ba,补充图gydF4y2Ba6gydF4y2Ba)。尽管他们的基因组大小有很大的差距,但在三者之间观察到高度的染色体亲缘性gydF4y2Ba葱属植物gydF4y2Ba基因组,这表明他们在进化上彼此接近。gydF4y2Ba
对基因组扩展和基因复制的见解gydF4y2Ba
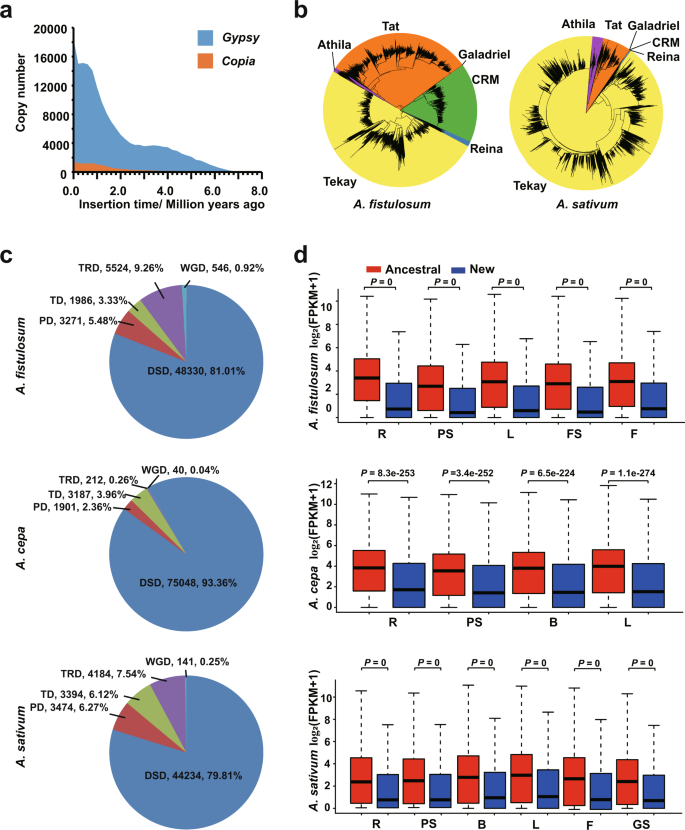
葱属植物gydF4y2Ba长期以来,植物一直以其庞大的基因组而闻名,其中蔬菜作物的基因组最大gydF4y2Ba26gydF4y2Ba。因此,我们分析了可能与之相关的关键因素gydF4y2Ba葱属植物gydF4y2Ba基因组的扩张。我们确定ltr是主要的基因组成分gydF4y2Ba答:fistulosumgydF4y2Ba(补充图。gydF4y2Ba3gydF4y2Ba及补充表gydF4y2Ba10gydF4y2Ba),gydF4y2Ba答:一种gydF4y2Ba基因组gydF4y2Ba10gydF4y2Ba。gydF4y2Ba吉普赛gydF4y2Ba是主要的LTR,主要在最近200万年爆发(图2)。gydF4y2Ba3gydF4y2Ba及补充图gydF4y2Ba7gydF4y2Ba)。在gydF4y2Ba吉普赛gydF4y2Ba其中,Tekey和Tat演化支的ltr最丰富gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:一种gydF4y2Ba基因组。CRM分支对LTR成分的贡献显著(10.38%)gydF4y2Ba答:fistulosumgydF4y2Ba(无花果。gydF4y2Ba3 bgydF4y2Ba)。虽然gydF4y2BaCopiagydF4y2Ba在两种作物中,LTRs只占重复序列的一小部分,其组成在两种作物之间存在显著差异gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:一种gydF4y2Ba基因组(图。gydF4y2Ba3gydF4y2Ba,附图gydF4y2Ba7gydF4y2Ba和gydF4y2Ba8gydF4y2Ba)。因此,ltr的积累,尤其是gydF4y2Ba吉普赛gydF4y2Ba型,负责基因扩增gydF4y2Ba葱属植物gydF4y2Ba而不同LTR支系的不均匀扩张导致了植物的多样性gydF4y2Ba葱属植物gydF4y2Ba基因组成分。gydF4y2Ba
一个gydF4y2Ba的插入次数分布gydF4y2Ba吉普赛gydF4y2Ba和gydF4y2BaCopiagydF4y2Ba反转位子活动在gydF4y2Ba答:fistulosumgydF4y2Ba。gydF4y2BabgydF4y2Ba的系统发育关系gydF4y2Ba吉普赛gydF4y2Baltr -反转录转座子在gydF4y2Ba答:fistulosumgydF4y2Ba(左)和gydF4y2Ba答:一种gydF4y2Ba(右)的基因组。gydF4y2BacgydF4y2Ba重复基因的分布在三个gydF4y2Ba葱属植物gydF4y2Ba基因组。DSD分散复制、PD近端复制、TD串联复制、TRD转座复制、WGD全基因组复制。gydF4y2BadgydF4y2Ba比较了3个品种的DSD原基因与新基因的表达水平gydF4y2Ba葱属植物gydF4y2Ba基因组(基因数目:gydF4y2BangydF4y2Ba= 13676;gydF4y2Ba答:fistulosumgydF4y2Ba,gydF4y2BangydF4y2Ba= 8424;gydF4y2Ba答:cepagydF4y2Ba,gydF4y2BangydF4y2Ba= 17817gydF4y2Ba答:一种gydF4y2Ba)。在箱形图中,箱形图的最小值、最大值和中心边界分别显示25%、75%四分位数值和平均值,而须形图则显示表达水平的最大值和最小值。gydF4y2BaP -gydF4y2Ba数值采用双尾Wilcoxon检验计算。R根,PS假茎,B球茎,L叶,FS花茎,F花,GS蒜芽。源数据作为源数据文件提供。gydF4y2Ba
这三个gydF4y2Ba葱属植物gydF4y2Ba在13个选择的基因组中,作物的总基因家族和独特基因家族数量最多(图2)。gydF4y2Ba2gydF4y2Ba)。为此,我们研究了16种基因组大小在0.2 ~ 28 Gb之间的物种基因复制的全球格局。大基因组的离散重复(DSD)事件发生频率较高,说明DSD事件在大基因组的基因扩增中起着重要作用(图2)。gydF4y2Ba3 cgydF4y2Ba,补充图gydF4y2Ba9gydF4y2Ba)。根据同源性将重复的DSD基因分为祖先基因和新基因gydF4y2Ba27gydF4y2Ba分别鉴定出13676、17817和8424对基因对(祖先/新基因对)gydF4y2Ba答:fistulosumgydF4y2Ba,gydF4y2Ba答:一种gydF4y2Ba,gydF4y2Ba答:cepagydF4y2Ba,分别。祖先DSD基因的表达水平明显高于新基因(图2)。gydF4y2Ba3 dgydF4y2Ba)。遗传基因和新基因之间的表达偏倚可能是由于它们的环境反应不同或功能冗余,这也意味着基因复制后可能出现假基因化、亚功能化或新功能化。我们认为普遍存在的DSD事件是基因扩增和分化的原因gydF4y2Ba葱属植物gydF4y2Ba基因组。gydF4y2Ba
风味相关基因的扩增与分化gydF4y2Ba
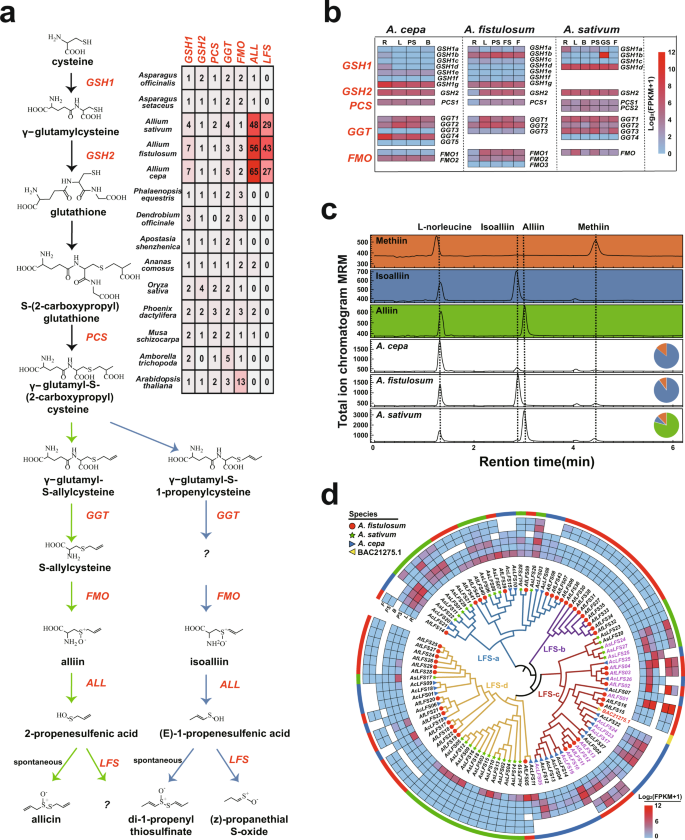
探索涉及的假定基因的进化事件gydF4y2Ba葱属植物gydF4y2Ba我们在14个植物基因组中鉴定了所有与acso相关的基因(图2)。gydF4y2Ba4gydF4y2Ba)。γ-谷氨酰半胱氨酸合成酶(gydF4y2BaGSH1gydF4y2Ba)同源物,一个谷胱甘肽合成酶(gydF4y2BaGSH2gydF4y2Ba)同源物,一个植物螯合素合成酶(gydF4y2Ba个人电脑gydF4y2Ba)同源,三gydF4y2Baγ-gydF4y2Ba谷氨酰转肽酶(gydF4y2BaGGTgydF4y2Ba)同源物和三种含黄素的单加氧酶(gydF4y2Ba弗兰克-蒙塔吉尼gydF4y2Ba)同源物在gydF4y2Ba答:fistulosumgydF4y2Ba基因组(图。gydF4y2Ba4gydF4y2Ba)。在这些基因中,gydF4y2BaAfGSH1b, AfGSH1g, AfGSH2, AfPSC1, AfGGT1, AfGGT2, AfFMO1gydF4y2Ba,gydF4y2BaAfFMO2gydF4y2Ba在根、假茎、叶、花茎和花中均有组成性表达,表明这些基因可能在acso的生物合成中发挥作用gydF4y2Ba葱属植物fistulosumgydF4y2Ba(无花果。gydF4y2Ba4 bgydF4y2Ba)。编码ACSOs水解关键酶的ALL基因在聚束洋葱中广泛扩增(56)gydF4y2BaAfALLgydF4y2BaS),洋葱球茎(65)gydF4y2Ba驱使着gydF4y2Ba5),大蒜(48)gydF4y2Ba资金gydF4y2Bas)基因组gydF4y2Ba3.gydF4y2Ba与其他选择的基因组相比(图2)。gydF4y2Ba4gydF4y2Ba,补充图gydF4y2Ba10gydF4y2Ba)。重要的是43 27 29gydF4y2BaLFSgydF4y2Ba在基因组中鉴定出同源物gydF4y2Ba答:fistulosumgydF4y2Ba,gydF4y2Ba答:cepagydF4y2Ba,gydF4y2Ba答:一种gydF4y2Ba,而不是gydF4y2BaLFSgydF4y2Ba在其他物种中发现了基因gydF4y2Ba葱属植物gydF4y2Ba所选基因组(图2)gydF4y2Ba4 dgydF4y2Ba)。gydF4y2Ba
一个gydF4y2BaACSOs的生物合成和水解途径以及14个基因组中各风味相关基因的数量。蓝色的途径与异蒜素的生物合成有关,异蒜素在鳞茎洋葱和束状洋葱中占主导地位。绿色的途径与大蒜中占优势的蒜素的生物合成有关。gydF4y2BabgydF4y2Ba研究了三种ACSOs生物合成基因的表达模式gydF4y2Ba葱属植物gydF4y2Ba作物。R根,L叶,PS假茎,B球茎,FS花茎,F花,GS蒜芽。基因的详细信息在补充数据中列出gydF4y2Ba1gydF4y2Ba- - - - - -gydF4y2Ba3.gydF4y2Ba。gydF4y2BacgydF4y2Ba三种acso的量化gydF4y2Ba葱属植物gydF4y2Ba作物采用超高效液相色谱法。饼状图显示了三种主要acso (alliin, isoalliin,和methiin)的百分比。gydF4y2BadgydF4y2BaLFS基因家族的系统发育树和表达模式gydF4y2Ba答:fistulosumgydF4y2Ba,gydF4y2Ba答:cepagydF4y2Ba,gydF4y2Ba答:一种gydF4y2Ba基因组。外环突出了来自gydF4y2Ba答:fistulosumgydF4y2Ba(红色),gydF4y2Ba答:cepagydF4y2Ba(蓝色),和gydF4y2Ba答:一种gydF4y2Ba(绿色)。热图显示了基因在不同组织中的表达情况:R根、L叶、PS假茎、B球茎、FS花茎、F花。源数据作为源数据文件提供。gydF4y2Ba
根据我们ACSOs的内容分析和相关报告gydF4y2Ba4gydF4y2Ba其中,异蒜素是主要的含硫生物活性物质gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba而在这两种作物中均未检测到蒜素。然而,alliin是主要的亚砜gydF4y2Ba答:一种gydF4y2Ba检测到少量异蒜素(图2)。gydF4y2Ba4摄氏度gydF4y2Ba)。LFS是将水解的异蒜素分解为体内特有的丙硫s -氧化物(催泪因子)的关键酶gydF4y2Ba葱属植物gydF4y2Ba作物。根据LFS基因家族的系统发育结构,我们将LFS基因家族分为LFS-a、LFS-b、LFS-c和LFS-d四组gydF4y2Ba葱属植物gydF4y2Ba作物(图。gydF4y2Ba4 dgydF4y2Ba)。此外,gydF4y2BaBAC21275.1gydF4y2Ba在鳞茎洋葱中被证实是功能性的LFSgydF4y2Ba28gydF4y2Ba并被聚为LFS-c家族(图2)。gydF4y2Ba4 dgydF4y2Ba)。gydF4y2BaAfLFS01gydF4y2Ba,最好的打击gydF4y2BaBAC21275.1gydF4y2Ba在洋葱丛中,除根外,大部分组织的转录水平最高gydF4y2BaAfLFSgydF4y2Ba基因检测(图2)gydF4y2Ba4 dgydF4y2Ba)。重要的是,八gydF4y2BaAfLFSgydF4y2Ba,七gydF4y2BaAcLFSgydF4y2Ba和三个gydF4y2BaAsLFSgydF4y2Ba该组基因表达模式相似(图2)。gydF4y2Ba4 dgydF4y2Ba)。基于密切的系统发育关系gydF4y2BaBAC21275.1gydF4y2Ba和相似的表达模式,我们推测LFS-c组可能在ACSOs水解中起同步作用gydF4y2Ba葱属植物gydF4y2Ba作物。然而,作为在系统发育上最接近的复制,gydF4y2BaAfLFS15gydF4y2Ba和gydF4y2BaAfLFS16gydF4y2Ba对表现出不同的表达模式gydF4y2BaAfLFS01gydF4y2Ba(无花果。gydF4y2Ba4 dgydF4y2Ba),提示这些同源基因可能存在功能分化。我们还观察到LFS组在这些作物中的不同表达偏倚。gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba倾向于广泛表达LFS-c组成员,而gydF4y2Ba答:一种gydF4y2Ba主要表达LFS-a组成员(图2)gydF4y2Ba4 dgydF4y2Ba)。同样,三个近端重复的基因(gydF4y2BaAfALL47gydF4y2Ba,gydF4y2BaAfALL48gydF4y2Ba,gydF4y2BaAfALL49gydF4y2Ba), 4个AcALL基因(gydF4y2BaAcALL05gydF4y2Ba,gydF4y2BaAcALL08gydF4y2Ba,gydF4y2BaAcALL09gydF4y2Ba,gydF4y2BaAcALL47gydF4y2Ba)和四个串联复制的AsALL基因(gydF4y2BaAsALL31gydF4y2Ba,gydF4y2BaAsALL32gydF4y2Ba,gydF4y2BaAsALL33gydF4y2Ba,gydF4y2BaAsALL34gydF4y2Ba)与已知的ALL基因(gydF4y2BaAAD26853.1gydF4y2Ba,gydF4y2BaAAA32639.1gydF4y2Ba,gydF4y2BaAAB32477.1gydF4y2Ba,gydF4y2BaACN78838.1gydF4y2Ba;补充图。gydF4y2Ba10gydF4y2Ba),在大多数组织中均表现出高表达水平。这些发现表明该基因簇可能是这三种作物的功能性ALL基因。gydF4y2Ba
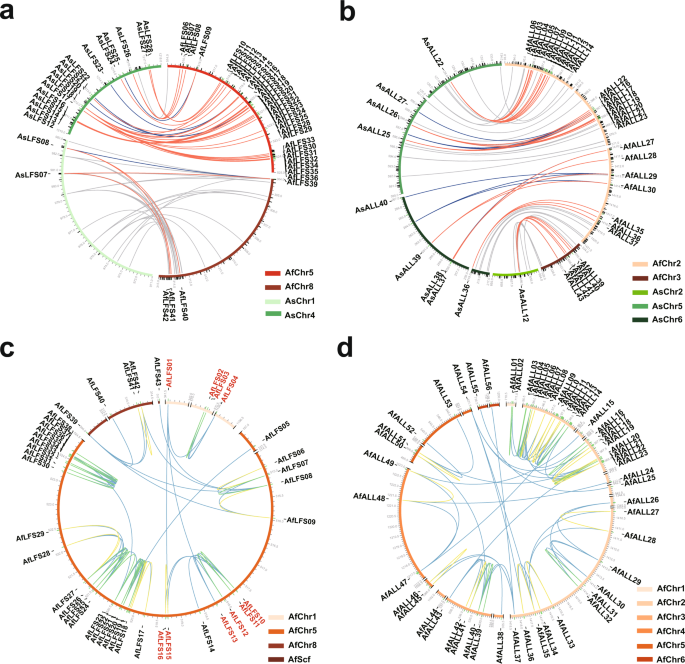
LFS-b组由9个AfLFS基因组成,在所有组织中均表现出极低的基因表达(图2)。gydF4y2Ba4 dgydF4y2Ba)。相反,在LFS-d组中,12个AsLFS基因聚集在一个独立的分支中,主要在根中表达(图2)。gydF4y2Ba4 dgydF4y2Ba)。此外,微共线性分析表明,几种gydF4y2BaLFSgydF4y2Ba和gydF4y2Ba所有gydF4y2Ba洋葱和大蒜的基因表现出一对多的互惠关系(图2)。gydF4y2Ba5 a、bgydF4y2Ba)。因此,我们推断的独立展开和微分gydF4y2BaLFSgydF4y2Ba和gydF4y2Ba所有gydF4y2Ba洋葱和大蒜从它们共同的祖先分化出来后的基因家族可能与它们的味道多样性有关(图2)。gydF4y2Ba4gydF4y2Bad,gydF4y2Ba5 a、bgydF4y2Ba及补充图gydF4y2Ba10gydF4y2Ba)。值得注意的是,分散复制,串联复制和近端复制事件gydF4y2BaLFSgydF4y2Ba和gydF4y2Ba所有gydF4y2Ba基因家族归因于基因重复(图2)。gydF4y2Ba5 c, dgydF4y2Ba、补充数据gydF4y2Ba1gydF4y2Ba)。特别是,33gydF4y2BaAfLFSsgydF4y2Ba在43个重复序列中,在Chr5上一个10 Mb的区域内以串联重复序列的形式聚集,LTRs和LFS基因在该区域交织共存,表明LTRs可能有助于LFS基因家族的增殖(图2)。gydF4y2Ba5度gydF4y2Ba,补充图gydF4y2Ba11gydF4y2Ba)。我们认为,硫代谢相关基因的独立扩增和分化在植物中普遍存在gydF4y2Ba葱属植物gydF4y2Ba基因组,这可能与特征辛辣味道的进化有关gydF4y2Ba葱属植物gydF4y2Ba作物。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba的微共线性关系gydF4y2BaLFSgydF4y2Ba(gydF4y2Ba一个gydF4y2Ba),gydF4y2Ba所有gydF4y2Ba(gydF4y2BabgydF4y2Ba)gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:一种gydF4y2Ba。红线表示基因在gydF4y2Ba答:fistulosumgydF4y2Ba,蓝线表示基因在gydF4y2Ba答:一种gydF4y2Ba,而灰线则表示有共线性关系的其他基因。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba的基因复制关系gydF4y2BaLFSgydF4y2Ba(gydF4y2BacgydF4y2Ba),gydF4y2Ba所有gydF4y2Ba(gydF4y2BadgydF4y2Ba)gydF4y2Ba答:fistulosumgydF4y2Ba。蓝线表示分散复制关系,绿线表示串联复制关系,黄线表示近端复制关系。基因的详细信息在补充数据中列出gydF4y2Ba1gydF4y2Ba- - - - - -gydF4y2Ba3.gydF4y2Ba。gydF4y2Ba
人口结构与迁徙路线gydF4y2Ba答:fistulosumgydF4y2Ba
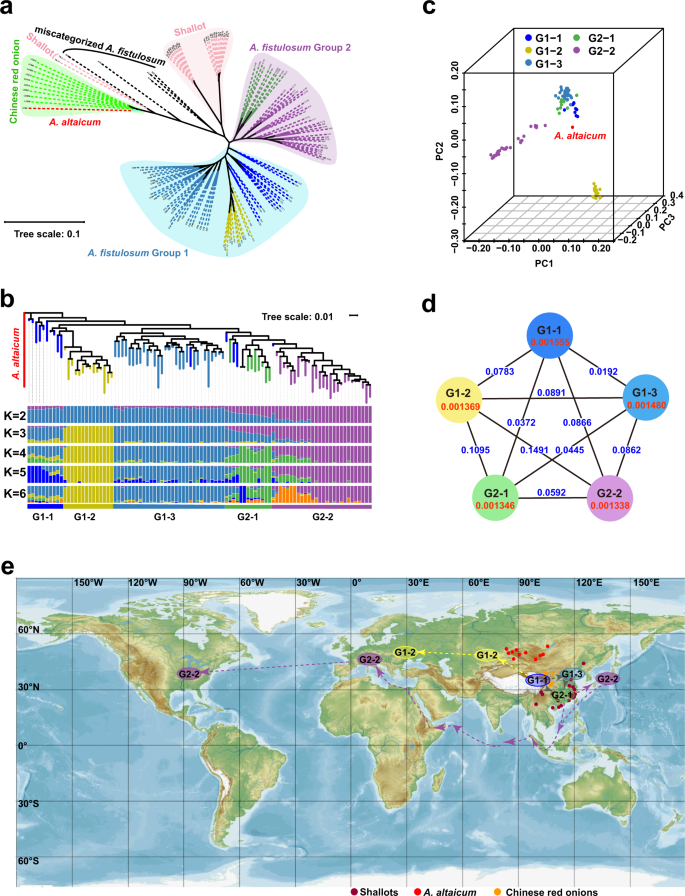
我们重新测序了135种不同的物种gydF4y2Ba葱属植物gydF4y2Ba的系统发育关系gydF4y2Ba答:fistulosumgydF4y2Ba作物及其近缘种(附图)gydF4y2Ba12gydF4y2Ba- - - - - -gydF4y2Ba13gydF4y2Ba及补充数据gydF4y2Ba4gydF4y2Ba)。我们得到了16453.99 Gb的Illumina短读,平均深度为9.49 ×基因组覆盖。总共鉴定出48,218,339个snp和18,182,723个插入和删除(InDels),平均为4.08个snp和1.54个InDels / kb。135的最大似然树gydF4y2Ba葱属植物gydF4y2Ba数据显示,以中国红洋葱(gydF4y2Ba答:cepagydF4y2Bavar。gydF4y2BaproliferumgydF4y2Ba)、青葱(gydF4y2Ba答:ascalonicumgydF4y2Ba),gydF4y2Ba答:fistulosumgydF4y2Ba第一组,以及gydF4y2Ba答:fistulosumgydF4y2Ba第二组(图。gydF4y2Ba6gydF4y2Ba)。九种中国红洋葱和gydF4y2Ba答:altaicumgydF4y2Ba加入紧密聚集成一组。22个葱聚集在一起,显示出两个不同的亚群,而剩下的两个葱接近中国红洋葱群(图2)。gydF4y2Ba6gydF4y2Ba)。中国红洋葱和青葱簇拥在一起gydF4y2Ba答:fistulosumgydF4y2Ba,与我们的参考基因组相对较低的基因组作图率(79-89%)相吻合。因此,我们得出结论,中国红洋葱和大葱是近亲或兄弟种gydF4y2Ba答:fistulosumgydF4y2Ba。九十六年gydF4y2Ba答:fistulosumgydF4y2Ba材料被分成两组;剩下的五个gydF4y2Ba答:fistulosumgydF4y2Ba在叶状图中,加入了大葱和中国红洋葱,这表明在叶状图中存在歧义或分配错误gydF4y2Ba葱属植物gydF4y2Ba分类(图。gydF4y2Ba6gydF4y2Ba及补充数据gydF4y2Ba4gydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba对135的最大似然树进行了重新排序gydF4y2Ba葱属植物gydF4y2Ba登记入册。gydF4y2BabgydF4y2Ba对96个群体的最大似然树和群体结构进行了重新排序gydF4y2Ba答:fistulosumgydF4y2Ba登记入册gydF4y2BacgydF4y2Ba, 96个重测序的主成分分析gydF4y2Ba答:fistulosumsgydF4y2Ba登记入册。gydF4y2BadgydF4y2Ba核苷酸多样性(π)和种群差异(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba)。红色字体的数字是π值,蓝色字体的数字是gydF4y2Ba置gydF4y2Ba值。gydF4y2BaegydF4y2Ba群集洋葱可能的迁移路线(地图从Mapswire下载)gydF4y2Bahttps://mapswire.com/world/physical-maps/gydF4y2Ba)。这些点表示近亲的栖息地gydF4y2Ba答:fistulosumgydF4y2Ba。gydF4y2Ba
澄清的演变和迁移gydF4y2Ba答:fistulosumgydF4y2Ba对96个确定的品种进行了系统亲缘关系和种群结构分析gydF4y2Ba答:fistulosumgydF4y2Ba登记入册,使用gydF4y2Ba答:altaicumgydF4y2Ba作为外群体。人口结构gydF4y2Ba答:fistulosumgydF4y2Ba的交叉验证(CV)误差被最小化gydF4y2BaKgydF4y2Ba= 5(补充图)gydF4y2Ba14gydF4y2Ba)。96年gydF4y2Ba答:fistulosumgydF4y2Ba条目可分为五个亚组,并分为两组(图2)。gydF4y2Ba6 a、bgydF4y2Ba)。类群1 (G1)根据种群结构可分为3个亚类群,各亚类群的地理分布各有特色。G1-1组主要分布在中国西部,G1-2组主要分布在中亚和俄罗斯联邦,G1-3组主要分布在中国北部和东北地区。类群2 (G2)也表现出两个可区分的地理亚群,中国东南部的植物聚集在G2-1亚群中。分布在日本、美国和欧洲的材料具有较高的基因组相似性,聚类为G2-2(图2)。gydF4y2Ba6 b, egydF4y2Ba)。gydF4y2Ba
此外,主成分分析(PCA)显示,G1-2和G2-2是单独聚集的,而三个中国起源群体(G1-1、G1-3和G2-1)则表现出更密切的关系(图2)。gydF4y2Ba6摄氏度gydF4y2Ba)。G1-3的连锁不平衡(LD)衰减最小,表明华北和东北地区可能是群落多样性中心gydF4y2Ba答:fistulosumgydF4y2Ba(补充图。gydF4y2Ba15gydF4y2Ba)。相比之下,G1-2表现出独特的结构模式(当gydF4y2BaKgydF4y2Ba= 3、4、5和6),核苷酸多样性(π)相对较低,LD衰减较高(图2)。gydF4y2Ba6 b, dgydF4y2Ba及补充图gydF4y2Ba15gydF4y2Ba)。此外,固定指标(gydF4y2BaFgydF4y2Ba圣gydF4y2Ba), G1-2/G1-1(0.0783)、G1-2/G1-3(0.0891)、G1-2/G2-1(0.1095)、G1-2/G2-2(0.1491)均高于其他亚组对(图2)。gydF4y2Ba6 dgydF4y2Ba)。G1-2的长前导分支(图2)gydF4y2Ba6 bgydF4y2Ba)表明G1-2类群可能存在较早的分化和独立驯化,这也部分解释了G1-2相对独立的PCA聚类(图2)。gydF4y2Ba6 b, cgydF4y2Ba)。此外,来自中国西部的4个物种(G1-1)与中亚的物种(G1-2,图1)聚集在一起。gydF4y2Ba6gydF4y2Ba), G1-1组向G1-2组有明显的基因流动(补充图2)。gydF4y2Ba16gydF4y2Ba),表明中亚人可能是从中国西部迁移过来的。G2的遗传结构呈现由G2-1向G2-2逐渐变化的趋势。当gydF4y2BaKgydF4y2Ba= 2和3,G2-1同时具有G1-3/G1-1和G2-2的遗传结构,说明G2-1与G1的亲缘关系比G2-2与G1的亲缘关系更密切(图2)。gydF4y2Ba6 bgydF4y2Ba)。此外,gydF4y2BaFgydF4y2Ba圣gydF4y2BaG1和G2-1之间的值低于G1和G2-2之间的值(图2)。gydF4y2Ba6 dgydF4y2Ba)。结合它们的地理位置,我们推测日本、美国和欧洲的物种(G2-2)是从中国东南部迁移或引进的(G2-1,图2)。gydF4y2Ba6 egydF4y2Ba)。gydF4y2Ba
异蒜素变异的基因组信号gydF4y2Ba答:fistulosumgydF4y2Ba
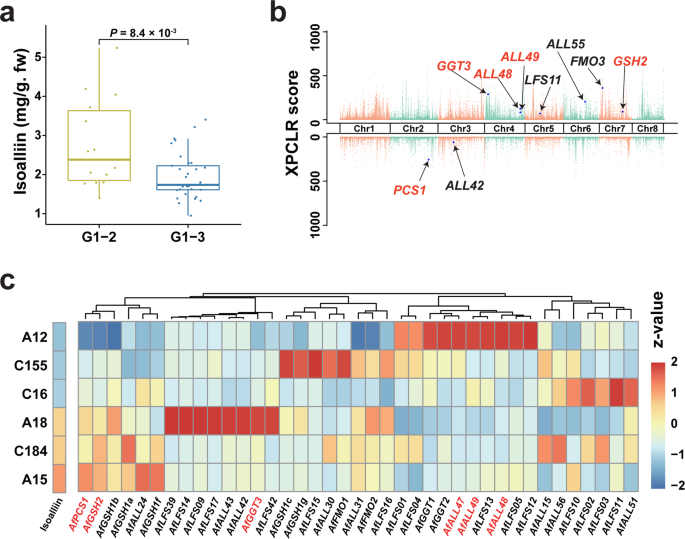
我们对91株的异蒜素含量进行了定量分析gydF4y2Ba答:fistulosumgydF4y2Ba确定异蒜素变异的基因组基础。G1-2品种的异蒜素含量显著高于G1-3品种(图2)。gydF4y2Ba7一个gydF4y2Ba)。通过比较这两组的选择性扫描,我们分别发现2718个和2133个基因位于G1-2和G1-3的选择性区域。几种与硫味形成有关的基因,如gydF4y2BaAfGSH2gydF4y2Ba,gydF4y2BaAfFMO3gydF4y2Ba,gydF4y2BaAfGGT3gydF4y2Ba,gydF4y2BaAfALL48gydF4y2Ba,gydF4y2BaAfALL49gydF4y2Ba,gydF4y2BaAfALL55gydF4y2Ba,gydF4y2BaAfLFS11gydF4y2Ba在G1-2阶段进行筛选。gydF4y2BaAfPCS1gydF4y2Ba和gydF4y2BaAfALL42gydF4y2Ba在G1-3中被确定为处于选择状态(图2)。gydF4y2Ba7 bgydF4y2Ba)。此外,6个材料叶片中几个acso生物合成或水解基因的表达量与内源异蒜素含量呈显著正相关,其中异蒜素含量差异显著gydF4y2BaAfGSH2gydF4y2Ba和gydF4y2BaAfPCS1gydF4y2Ba表现出最高的相关性(图2)。gydF4y2Ba7 cgydF4y2Ba)。在低异蒜素品种中高表达的25个风味形成相关基因(A12、C155和C16)中,19个ACSOs水解相关基因(gydF4y2Ba所有gydF4y2Ba和gydF4y2BaLFSgydF4y2Ba表明弱生物合成和强水解都可能导致它们较低的异蒜素积累。在这19个ACSOs水解相关基因中,差异较大gydF4y2BaLFSgydF4y2Ba或gydF4y2Ba所有gydF4y2Ba观察了6个材料的基因表达模式(图2)。gydF4y2Ba7 cgydF4y2Ba),这意味着ACSOs水解相关基因的广泛扩增可能为聚束洋葱异蒜素含量的变异提供了更多的进化机会。gydF4y2Ba
一个gydF4y2BaG1-2叶片中异蒜素含量(gydF4y2BangydF4y2Ba= 14)及G1-3 (gydF4y2BangydF4y2Ba= 33)。gydF4y2BaP -gydF4y2Ba数值采用双尾Wilcoxon检验计算。在箱形图中,箱形图的最小值、最大值和中心边界分别显示25%、75%四分位数值和平均值,而须形图则显示异蒜素含量的最大值和最小值。gydF4y2BabgydF4y2Ba上图:使用XP-CLR(跨群体复合似然比检验)值比较G1-2与G1-3的选择性扫描的全基因组分布。下图:G1-3与G1-2中选择性扫描的全基因组分布。gydF4y2BacgydF4y2Ba6个材料叶片中异蒜素浓度及含硫风味相关基因(平均FPKM < 0.5的基因过滤)的基因表达谱。色阶表示由R软件归一化的平均异蒜素含量或FPKM z值。源数据作为源数据文件提供。gydF4y2Ba
讨论gydF4y2Ba
在本研究中,我们生成了染色体水平的大基因组gydF4y2Ba答:fistulosumgydF4y2Ba,基因组大小为11.27 Gb, contig N50为7.34 Mb(表2)gydF4y2Ba1gydF4y2Ba)。我们的参考基因组在主要组装指标和几项评估(BUSCO, CEGMA, LAI和mercury;补充表gydF4y2Ba6gydF4y2Ba和gydF4y2Ba7gydF4y2Ba)。毫无疑问,这一基因组组装的成就将加速我们对主要基因的进化和相关性的理解gydF4y2Ba葱属植物gydF4y2Ba作物。洋葱和大蒜的基因组扩增都是由基因突变引起的gydF4y2Ba吉普赛gydF4y2Ba型LTR,但它们表现出LTR亚支的不均匀扩张。同时,DSD事件对基因扩增的贡献占主导地位gydF4y2Ba葱属植物gydF4y2Ba作物(图。gydF4y2Ba3.gydF4y2Ba)。基于ssr标记的洋葱遗传图谱显示,束洋葱和鳞茎洋葱具有高度保守的基因组同源性gydF4y2Ba21gydF4y2Ba在染色体水平上,鳞茎洋葱、葱束洋葱和大蒜的基因组共线性较高。洋葱的染色体(gydF4y2Ba答:fistulosumgydF4y2Ba)及洋葱(gydF4y2Ba答:cepagydF4y2Ba)几乎呈一一共线性(图2)。gydF4y2Ba2摄氏度gydF4y2Ba,补充图gydF4y2Ba6gydF4y2Ba),表明两者之间有很高的血缘关系gydF4y2Ba葱属植物gydF4y2Ba作物。因此,聚束洋葱的每条染色体的估计序列可以作为染色体结构具有高度共性的洋葱的模板。同时,这一基因组证据很好地解释了种间杂交的普遍存在和弱生殖隔离gydF4y2Ba葱属植物gydF4y2Ba作物gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,为指导今后的基因组辅助育种提供了有价值的信息gydF4y2Ba葱属植物gydF4y2Ba作物。gydF4y2Ba
的gydF4y2Ba葱属植物gydF4y2Ba由于其特定的半胱氨酸衍生的硫化合物,属具有独特的风味和药用特性gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba。我们观察到氧化石墨烯中硫代谢相关基因在“碳-硫裂解酶活性”和“半胱氨酸型肽酶活性”方面得到了特异性扩展gydF4y2Ba葱属植物gydF4y2Ba所检查的作物(图2)gydF4y2Ba2 bgydF4y2Ba)。重要的是,广泛的扩张gydF4y2Ba所有gydF4y2Ba和gydF4y2BaLFSgydF4y2Ba基因家族是一个显著的特征gydF4y2Ba葱属植物gydF4y2Ba作物。在完整的组织中,acso储存在叶肉细胞的细胞质中。在组织损伤时,液泡ALL酶接触并水解acso产生亚磺酸,亚磺酸通过自发反应进一步转化为各种含硫生物活性化合物gydF4y2Ba5gydF4y2Ba因此,我们推测ACSOs水解相关基因的大量扩增可能导致相关转录本和相关酶的过度积累,这可能有助于对外界刺激的快速反应。其中,LFS基因家族仅在gydF4y2Ba葱属植物gydF4y2Ba14个选定基因组中的作物(图2)。gydF4y2Ba4gydF4y2Ba)。这些结果有力地支持了gydF4y2Ba葱属植物gydF4y2Ba农作物进化出巨大而特殊的ALL和LFS基因家族,可能是为了利用其挥发性和刺激性的最终产物来抵抗疾病和昆虫gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba。gydF4y2Ba
的物种特异性聚类gydF4y2Ba所有gydF4y2Ba和gydF4y2BaLFSgydF4y2Ba基因家族在三者中表现出各自独立的扩展gydF4y2Ba葱属植物gydF4y2Ba作物(图。gydF4y2Ba4gydF4y2Ba,补充图gydF4y2Ba10gydF4y2Ba),这意味着它们的辛辣味道可能是趋同进化的结果。我们知道,即使只替换一个氨基酸,LFS的活性也会降低到1/100甚至更低gydF4y2Ba29gydF4y2Ba。因此,我们推测LFS基因家族的广泛复制和分化可能导致了蛋白质活性或功能多样性的差异。具有高活性和高表达水平的LFS基因可能参与了玉米撕裂特性和特色风味的产生gydF4y2Ba葱属植物gydF4y2Ba作物。那些没有或忽略LFS活性的成员可能在其他次级代谢途径中起作用。据报道,不同的萜烯合成酶(TPS)亚群参与不同的生物活性萜烯生物合成gydF4y2Ba30.gydF4y2Ba。同样的,gydF4y2Ba答:fistulosumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba,倾向于广泛表达可能在异蒜素水解中起作用的LFS-c基团成员。然而,gydF4y2Ba答:一种gydF4y2Ba主要表达LFS-a和LFS-c成员(图2)。gydF4y2Ba4 c, dgydF4y2Ba),可能对蒜素和异蒜素的分解起作用。acso可以分解成各种含硫组分,包括二烯丙基和甲基烯丙基gydF4y2Ba31gydF4y2Ba因此,我们推测LFS和ALL基因家族的不同分支可能在其他含硫生物活性化合物的形成中发挥作用,这需要在未来的遗传和生化研究中进一步验证。gydF4y2Ba
此外,ALL和LFS基因家族的扩增可能与LTRs的增殖是同步的。gydF4y2Ba11gydF4y2Ba)gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba。此外,gydF4y2Ba弗兰克-蒙塔吉尼gydF4y2Ba参与acso和硫代葡萄糖苷生物合成的基因仅在gydF4y2Ba拟南芥gydF4y2Ba在选定的14个基因组中(图2)。gydF4y2Ba4gydF4y2Ba),这可能与含硫硫代葡萄糖苷的积累有关gydF4y2Ba十字花科gydF4y2Ba作物gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba。然而,缺乏几个与硫代葡萄糖苷相关的基因gydF4y2Ba葱属植物gydF4y2Ba与植物相比,植物可能会选择不同的进化方向来合成含硫生物活性化合物gydF4y2Ba十字花科gydF4y2Ba植物(图。gydF4y2Ba4gydF4y2Ba及补充图gydF4y2Ba17gydF4y2Ba)。gydF4y2Ba
传统的gydF4y2Ba葱属植物gydF4y2Ba分类在进化研究和农业用途开发中造成混乱,主要是因为缺乏基因组信息。在此之前,gydF4y2Ba答:altaicumgydF4y2Ba通常被认为是gydF4y2Ba答:fistulosumgydF4y2Ba基于表型相似性和非编码叶绿体DNA相似性gydF4y2Ba12gydF4y2Ba,gydF4y2Ba34gydF4y2Ba(补充图。gydF4y2Ba12gydF4y2Ba)。在这里,我们发现它没有聚类gydF4y2Ba答:fistulosumgydF4y2Ba在基于snp的系统发育或PCA分析中,但在系统发育上更接近中国红洋葱(图2)。gydF4y2Ba6gydF4y2Ba及补充数据gydF4y2Ba4gydF4y2Ba)。因此,我们假设gydF4y2Ba答:altaicumgydF4y2Ba与…密切相关gydF4y2Ba答:fistulosumgydF4y2Ba而不是它的祖先。与早期的形态学分类相反,三层洋葱(gydF4y2Ba答:fistulosumgydF4y2BavargydF4y2Ba。viviparumgydF4y2Ba)和中国红洋葱(gydF4y2Ba答:cepagydF4y2BavargydF4y2Ba。proliferumgydF4y2Ba),通过气生球茎繁殖,分成两个不同的组(图2)。gydF4y2Ba6gydF4y2Ba,补充图gydF4y2BaS18gydF4y2Ba及补充数据gydF4y2Ba4gydF4y2Ba)。它们的空中鳞茎可以用于无性繁殖,这可能是趋同进化的结果。gydF4y2Ba
G1-1的地理位置靠近主要的多样性中心(从地中海盆地到中亚)gydF4y2Ba葱属植物gydF4y2Ba属gydF4y2Ba2gydF4y2Ba,在该亚群中观察到最高的核苷酸多样性(图2)。gydF4y2Ba6 dgydF4y2Ba)。这些发现与中国西北地区是其起源中心的历史记载相一致gydF4y2Ba答:fistulosumgydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba。此外,对文学的培养gydF4y2Ba答:fistulosumgydF4y2Ba中国的传统文化可以追溯到公元前三世纪。日本gydF4y2Ba答:fistulosumgydF4y2Ba本品被认为是在大约公元720年从中国引进的。gydF4y2Ba答:fistulosumgydF4y2Ba是在中世纪末期(公元1000-1400年)从亚洲传到西欧的吗gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba。这些记录支持了我们的系统发育结果,即G2-2(来自日本和欧美国家)可能是从中国南方迁移过来的。gydF4y2Ba
此外,在gydF4y2Ba答:fistulosumgydF4y2Ba在系统基因组分析中,不同的物种表现出不同的地理聚类而不是形态分类(图2)。gydF4y2Ba6 bgydF4y2Ba)。分布在西伯利亚及其邻近地区的中亚物种(G1-2)可能是从中国西部迁移而来(G1-1),而日本、美国和欧洲物种(G2-2)可能是从中国东南部迁移或引入的(G2-1)。gydF4y2Ba6 egydF4y2Ba)。综上所述,我们验证了中国西北地区(G1-1)是gydF4y2Ba答:fistulosumgydF4y2Ba。gydF4y2Ba
综上所述,高质量的染色体水平基因组gydF4y2Ba答:fistulosumgydF4y2Ba极大地促进了我们对基因组多样性和扩展的理解gydF4y2Ba葱属植物gydF4y2Ba作物。中国原产品种的高度多样化和近亲的存在表明中国是主要的原产地和驯化中心gydF4y2Ba答:fistulosumgydF4y2Ba作物。蒜氨酸酶和催泪因子合成酶基因家族的扩增和分化可能与此有关gydF4y2Ba葱属植物gydF4y2Ba-特定的风味形成。我们目前的研究将为进一步的功能基因组研究和重要农艺性状的基因组选择提供基础gydF4y2Ba葱属植物gydF4y2Ba作物。gydF4y2Ba
方法gydF4y2Ba
植物材料和测序文库准备gydF4y2Ba
对生长在中国浙江省杭州的洋葱串属“SXSJC”进行了基因组组装测序。关于135的详细信息gydF4y2Ba葱属植物gydF4y2Ba用于重测序的数据在补充数据中提供gydF4y2Ba4gydF4y2Ba。一共101个gydF4y2Ba答:fistulosumgydF4y2Ba加入者分布在世界各地,包括中国、日本和欧洲、美洲和中亚国家。青葱和中国红洋葱是从中国收集的,而野生的gydF4y2Ba答:altaicumgydF4y2Ba它被认为是“栽培”的近亲和潜在祖先gydF4y2Ba答:fistulosumgydF4y2Ba13gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,采自阿尔泰山脉(89.44E, 47.05 N;中国新疆)。gydF4y2Ba
基因组组装使用DNAsecure Plant Kit (Tiangen Biotech, Beijing, China)从洋葱幼叶中提取基因组DNA,并通过琼脂糖凝胶电泳检测DNA质量。单分子实时(SMRT) PacBio测序文库按照Pacific Biosciences的标准方案构建。简单地说,将高质量的基因组DNA剪切至~20 kb,将受损的末端修复并用钝端接头连接,然后使用PacBio Sequel平台对所得到的文库进行测序。gydF4y2Ba
对于Bionano测序,使用建议的试剂盒(Bionano Prep Plant DNA Isolation Kit)提取DNA,并标记整个基因组中的特定序列。标记的DNA被转移到一个墨盒中进行扫描,图像被转化为分子。gydF4y2Ba
根据建议的程序构建了Hi-C库。简单地说,在提取染色质之前,用甲醛溶液固定叶片样品,并用400U的DPNII限制性内切酶在37℃下消化染色质。DNA末端用生物素标记,用T4 DNA连接酶(NEB)进行DNA连接。结扎后加入蛋白酶K进行反交联。然后将DNA片段纯化并溶解。纯化后的DNA片段化至300-500 bp, DNA末端得到修复。生物素标记的DNA片段使用dynabead®M-280 Streptavidin (Life Technologies)进行分离。对Hi-C文库进行质量控制,并在Illumina HiSeq X 10测序仪上进行测序。gydF4y2Ba
对于Illumina测序,根据先前描述的程序(Illumina)构建DNA文库。简单地说,使用Covaris超声波破碎机将DNA随机破碎成片段,修复DNA末端,并添加poly (a)接头。使用纯化的DNA进行PCR,评估后在Illumina平台上对最终文库进行测序。gydF4y2Ba
基因组组装和质量评估gydF4y2Ba
测定基因组大小、杂合率和重复序列率gydF4y2Bak-mergydF4y2Ba39gydF4y2Ba分布分析(k = 17)。PacBio reads使用CANU组装contig水平的基因组gydF4y2Ba40gydF4y2Ba(v1.9,参数:genomeSize = 12g, corOutCoverge = 40g)。使用NextPolish (v1.2.4;Rerun = 3,gydF4y2Bahttps://github.com/Nextomics/NextPolishgydF4y2Ba)和Illumina短读使用Pilon (v1.2.2)gydF4y2Ba41gydF4y2Ba。随后,使用purge_dup (v1.2.3)删除冗余序列gydF4y2Ba42gydF4y2Ba。使用Bionano solution (v3.5.1, DeNovo assembly: - 5 - f1 -W 1 -c 1, Hybrid Scaffold: - b2 - n2)进行辅助装配,以提高装配精度gydF4y2Ba43gydF4y2Ba。利用Hi-C数据,利用allic (v0.9.8,gydF4y2Bahttps://github.com/tangerzhang/ALLHiCgydF4y2Ba)软件。gydF4y2Ba
采用互补方法评价基因组组装的质量。首先,基于BUSCO (v3.0.2)和CEGMA (v2.5)数据库中的植物保守基因进行基因组完整性评估。其次,使用BWA (v0.7.8)将Illumina短读段定位到组装的基因组上。gydF4y2Ba44gydF4y2Ba评估覆盖率和平均深度。LTR_retriever (v1.0.7)gydF4y2Ba22gydF4y2Ba采用包装法评价基因组质量。使用Mequery (v1.3)来评估基因组组装的一致性质量(QV)值和完整性gydF4y2Ba20.gydF4y2Ba。基因图谱的标记gydF4y2Ba21gydF4y2Ba利用Chromonomer (v1.07,gydF4y2Bahttps://github.com/jleluyer/chromonomer_workflowgydF4y2Ba),并使用Python脚本显示共线关系。gydF4y2Ba
基因组注释gydF4y2Ba
RepeatMasker和RepeatProteinMask (v4.07)gydF4y2Ba45gydF4y2Ba用于通过与重复库(Repbase v15.02)比对来识别te,而使用RepeatModeler (v1.05,gydF4y2Bahttp://www.repeatmasker.org/RepeatModeler/gydF4y2Ba(版本1.0.5)、RepeatScout和LTR_FINDER。使用串联重复序列查找器(TRF, v4.09)分析基因组中的串联重复序列。gydF4y2Ba
使用五个从头开始的基因预测程序进行从头预测:gydF4y2Ba46gydF4y2Ba、Geneid (v1.4);gydF4y2Bahttps://genome.crg.cat/software/geneid/index.htmlgydF4y2Ba), Genescan (v1.0,gydF4y2Bahttp://genes.mit.edu/GENSCAN.htmlgydF4y2Ba),微光hmm (v3.0.2,gydF4y2Bahttps://ccb.jhu.edu/software/glimmerhmm/gydF4y2Ba)和SNAP (v2013.11.29,gydF4y2Bahttps://snap.stanford.edu/gydF4y2Ba)。12个物种的蛋白质序列(gydF4y2Ba答:一种gydF4y2Ba11gydF4y2BaA. cepagydF4y2Ba25gydF4y2Ba,gydF4y2Ba文竹gydF4y2Ba47gydF4y2Ba芦笋gydF4y2Ba48gydF4y2Ba铁皮石斛gydF4y2Ba49gydF4y2Ba,深圳旅社gydF4y2Ba50gydF4y2Ba蝴蝶兰gydF4y2Ba51gydF4y2Ba,gydF4y2Ba凤凰dactyliferagydF4y2Ba52gydF4y2BaOryza sativagydF4y2Ba53gydF4y2Ba,gydF4y2Ba菠萝comosusgydF4y2Ba54gydF4y2Ba;三头蛇gydF4y2Ba55gydF4y2Ba,gydF4y2Ba和裂果菇gydF4y2Ba56gydF4y2Ba)与gydF4y2Ba答:fistulosumgydF4y2Ba利用TBLASTN (v2.2.26, e值<10e-5)对基因模型进行预测。大礼帽(v2.0.13gydF4y2Bahttp://ccb.jhu.edu/software/tophat/index.shtmlgydF4y2Ba)和袖扣(v2.1.1,gydF4y2Bahttp://cufflinks.cbcb.umd.edu/gydF4y2Ba)管道用于将RNA-seq数据映射到gydF4y2Ba答:fistulosumgydF4y2Ba基因组用于基因预测。Trinity (v2.1.1)和PASA (v2.2.0)gydF4y2Ba57gydF4y2Ba进行基因结构分析。使用这些方法预测的所有基因模型都使用加权和非冗余基因集1.1.1 (EVM, v1.1.1,gydF4y2Bahttp://evidencemodeler.github.io/gydF4y2Ba)。gydF4y2Ba
通过敲开SwissProt、TrEMBL和NCBI非冗余蛋白数据库进行基因注释,选择最佳位点进行注释。使用InterProScan (v4.7)检索InterPro (v29.0)数据库对motif和domain进行标注。使用最佳匹配分类获得每个基因的GO和KEGG通路。使用tRNAscan-SE (v1.4)预测非编码rna。gydF4y2Ba58gydF4y2Ba和INFERNAL (v1.1.2)gydF4y2Ba59gydF4y2Ba软件gydF4y2Ba
WGD事件分析gydF4y2Ba
FASTKs (gydF4y2Bahttps://github.com/mrmckain/FASTKsgydF4y2Ba)来计算同义替换(gydF4y2BaKsgydF4y2Ba中相邻块的值gydF4y2Ba答:fistulosumgydF4y2Ba。的密度估计gydF4y2BaKsgydF4y2Ba数值分布基于有限正态混合模型,使用MCLUST (gydF4y2Bahttps://sites.stat.washington.edu/mclust/gydF4y2Ba),并利用k值的分布来确定WGD事件。gydF4y2Ba
比较基因组学和基因组进化分析gydF4y2Ba
简单地说,同源基因家族(单拷贝和多拷贝家族)gydF4y2Ba答:fistulosumgydF4y2Ba其他基因组(gydF4y2Ba答:一种gydF4y2Ba11gydF4y2Ba,gydF4y2Ba答:cepagydF4y2Ba25gydF4y2Ba,gydF4y2Ba文竹gydF4y2Ba47gydF4y2Ba,gydF4y2Ba芦笋officinalisgydF4y2Ba48gydF4y2Ba,gydF4y2Ba石斛兰officinalegydF4y2Ba49gydF4y2Ba,gydF4y2BaApostasia shenzhenicagydF4y2Ba50gydF4y2Ba,gydF4y2Ba蝴蝶兰属equestrisgydF4y2Ba51gydF4y2Ba,gydF4y2Ba凤凰dactyliferagydF4y2Ba52gydF4y2Ba,gydF4y2Ba栽培稻gydF4y2Ba53gydF4y2Ba,gydF4y2Ba菠萝comosusgydF4y2Ba54gydF4y2Ba,gydF4y2BaAmborella trichopodagydF4y2Ba55gydF4y2Ba和gydF4y2Ba穆萨schizocarpagydF4y2Ba56gydF4y2Ba),使用OrthoMCL (v1.4,gydF4y2Bahttp://orthomcl.org/orthomcl/gydF4y2Ba)。利用MUSCLE (v3.8.31,gydF4y2Bahttp://www.drive5.com/muscle/gydF4y2Ba),随后使用RaxML (ML tree, Model GTRGAMMA, v8.2.12;gydF4y2Bahttp://sco.h-its.org/exelixis/web/software/raxml/index.htmlgydF4y2Ba)。Mcmctree (burn-in = 10,000,样本数= 100,000,采样频率= 2;gydF4y2Bahttp://abacus.gene.ucl.ac.uk/software/paml.htmlgydF4y2Ba),基于TimeTree数据库(gydF4y2Bahttp://www.timetree.org/gydF4y2Ba)及先前的报告gydF4y2Ba55gydF4y2Ba。使用CAFÉ (v4.2)检测基因家族的扩增和收缩gydF4y2Ba60gydF4y2Ba软件使用MCScanx流水线分析大蒜和洋葱的基因组共线性gydF4y2Ba61gydF4y2Ba。gydF4y2Ba
LTRs的插入时间及系统发育分析gydF4y2Ba
利用MUSCLE (v3.8.31, (gydF4y2Bahttp://www.drive5.com/muscle/gydF4y2Ba), ltr的插入时间(T)用公式T = K/2r计算。K为遗传距离,计算公式为K =−0.75ln (1-4λ/3)。保留两个ltr之间小于0.75的核苷酸发散率(λ)以供进一步分析。核苷酸取代率“r”被设定为每年每个位点有1.3e-8个取代。利用未校正的两两距离,利用TreeBest (v1.9.2,gydF4y2Bahttp://treesoft.sourceforge.net/treebest.shtmlgydF4y2Ba)和建议的参数。我们使用LTRdigest (v1.07)将ltr分类为Copia、Gypsy和其他超家族。gydF4y2Ba62gydF4y2Ba,而次级ltr则根据它们的系统发育特性来确定。gydF4y2Ba
祖先和新DSD基因的鉴定gydF4y2Ba
使用DupGen_finder将基因复制事件分为分散复制、近端复制、WGD复制、串联复制和转置复制。gydF4y2Bahttps://github.com/DXXDR/DupGen_findergydF4y2Ba)管道。我们研究了13个基因组和几个大基因组的基因复制的全球格局,其中包括三个大基因组gydF4y2Ba银杏叶gydF4y2Ba63gydF4y2Ba、黑麦gydF4y2Ba64gydF4y2Ba和油松gydF4y2Ba65gydF4y2Ba。基于JCVI分析染色体共线性,确定基因是否位于共线性区域gydF4y2Ba61gydF4y2Ba。这两个gydF4y2Ba答:一种gydF4y2Ba和gydF4y2Ba答:cepagydF4y2Ba使用gydF4y2Ba答:fistulosumgydF4y2Ba作为参考基因组,而gydF4y2Ba答:一种gydF4y2Ba作为参考基因组gydF4y2Ba答:fistulosumgydF4y2Ba。共线区域内的基因被认为是祖先DSD基因,而共线区域外的基因被认为是新DSD基因。我们选取分散的基因对,其中一个为祖先基因,对应的基因为新基因,分析其表达差异。gydF4y2Ba
风味相关基因的鉴定和进化分析gydF4y2Ba
为了鉴定参与硫代谢生物合成途径的基因,BLASTP (v 2.2.26)的关键参数为e值<1e-5,身份>为50%。利用HMMER (v3.1b1)软件鉴定候选基因的蛋白结构域,过滤不同结构域的基因。球茎洋葱LFS基因的蛋白序列(GenBank登录号:AB089203)和大蒜ALL基因(GenBank登录号:使用NCBI数据库中的AAB32477.1、ACN78838.1、AAD26853.1和AAA32639.1)对候选同源物进行识别。从文献综述中获得了硫代葡萄糖苷相关基因的查询序列gydF4y2Ba33gydF4y2Ba。利用MUSCLE (v3.8.31)软件比对LFS和ALL基因的全长氨基酸序列,利用Treebest (v1.9.2,gydF4y2Bahttp://treesoft.sourceforge.net/treebest.shtmlgydF4y2Ba)及ITOLs (gydF4y2Bahttps://itol.embl.de/gydF4y2Ba)。gydF4y2Ba
亚砜相关基因的空间表达gydF4y2Ba
对束状洋葱的根、假茎、叶、花茎、花和鳞茎洋葱的根、球茎、叶、假茎进行Illumina RNA-seq测序,并进行4次生物重复。基因表达数据集使用Tophat (gydF4y2Bahttp://ccb.jhu.edu/software/tophat/index.shtmlgydF4y2Ba)及袖扣(gydF4y2Bahttp://cufflinks.cbcb.umd.edu/gydF4y2Ba)管道。目的基因的表达模式利用热图文库(gydF4y2Bahttps://CRAN.R-project.org/package=pheatmapgydF4y2Ba)在R软件中使用loggydF4y2Ba2gydF4y2Ba(FPKM + 1)值。gydF4y2Ba
重测序和群体基因组学分析gydF4y2Ba
使用Illumina HIseq2500平台,从135份文献中生成了总计350 bp的对端读取。的映射率gydF4y2Ba答:ascalonicumgydF4y2Ba和gydF4y2Ba答:cepagydF4y2BavargydF4y2Ba。proliferumgydF4y2Ba小于90,4 ×基因组覆盖率小于60%。我们用了96gydF4y2Ba答:fistulosumgydF4y2Ba以过滤snp。snp的质量控制步骤如下:(1)去除Illumina文库构建适配器,(2)去除含有10%以上未知碱基的reads (N个碱基),(3)去除含有50%以上低质量碱基的低质量reads(测序质量值≤5)。BWA (v0.7.8)gydF4y2Ba44gydF4y2Ba用于将clean reads与参考基因组进行比对,Samtools (gydF4y2Bahttps://github.com/samtools/samtoolsgydF4y2Ba)使用以下参数(-q 1 -C 50 -m 2 -F 0.002 -d 1000)调用snp / indel。对映射质量<20、变异位置深度<3、缺失率< 0.05、SNP频率<5%的原始SNP/InDel集进行过滤。经过筛选,我们从135个样本中选择了高质量的snpgydF4y2Ba葱属植物gydF4y2Ba后续分析的附件。ANNOVAR (v20191024gydF4y2Bahttps://annovar.openbioinformatics.org/en/latest/gydF4y2Ba)用于变体的功能注释。使用SNPhylo (v20180901)构建系统发育树。gydF4y2Ba66gydF4y2Ba。利用admix (v1.23)软件对各种质资源的种群结构进行了研究。此外,使用VCFtools (v0.1.12b,gydF4y2Bahttps://vcftools.github.io/index.htmlgydF4y2Ba)。使用PopLDdecay (v03.41)计算链接不平衡分析(LD衰减)gydF4y2Ba67gydF4y2Ba。XP-CLR评分使用XP-CLR (v1.0)计算,滑动窗口大小为40 kb,步长为20 kbgydF4y2Ba68gydF4y2Ba选取前5%区域作为候选选择区域。gydF4y2Ba
中acso的测定gydF4y2Ba葱属植物gydF4y2Ba作物gydF4y2Ba
为了防止ACSO的降解,用O-(羧甲基)半盐酸羟胺(OCMHA)抑制蒜氨酸酶的活性gydF4y2Ba69gydF4y2Ba。取0.15 ~ 0.20 g肉重样品,取2 mL含5 mm钢球的离心管,加入990 μL OCMHA溶液(1.1 g/L甲醇/去离子水4:1,v/v)和10 μL去甲亮素溶液(1 mg/mL水)。用组织研磨机揉搓后,匀浆1 min。将得到的浆液离心(10分钟,室温,12000转/分),然后将上清稀释10000倍gydF4y2BaggydF4y2Ba。用乙腈和去离子水的混合物(4:1,v/v)。最终提取液保存在- 20°C的小瓶中直到分析。gydF4y2Ba
通过与正品标准(alliin, Beijing SoIarbio Life Science Company, Beijing, China;异蒜素,成都草源康生物技术公司,中国成都;和甲硫胺,来自上海源业生物技术公司,上海,中国)。在0.01 ~ 500 ng/mL浓度范围内采用混合校准曲线进行acso定量。ACSO含量测定采用Agilent 1290超高效液相色谱仪耦合6470三重四极杆光谱仪(Agilent Technologies, Waldbronn, Germany)。色谱柱为Waters BEH酰胺柱(100 × 2.1 mm, 1.7 μm),温度为60℃,流速为0.6 mL/min。流动相为(A)含0.5%甲酸的水和(B)含0.5%甲酸的乙腈。梯度洗脱:0-4 min, 10-15% A;4-8分钟,15-60% A;8-15分钟,10% A; injection volume 1 μL. Ionization was conducted in positive ion electrospray mode, and the operating parameters were as follows: capillary voltage, 3000 V; drying gas temperature, 325 °C; drying gas flow, 5 L/min; nebulizer pressure, 45 psi; sheath gas temperature, 350 °C; and sheath gas flow, 11 L/min. In addition, three biological replicates were used for all the accessions.
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然投资组合报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
本研究产生的所有原始测序数据已存入CNSA (gydF4y2Bahttps://db.cngb.org/cnsa/gydF4y2Ba根据加入gydF4y2BaCNP0002276gydF4y2Ba。的表达式数据gydF4y2Ba答:一种gydF4y2Ba从NCBI GEO数据库下载gydF4y2BaGSE145455gydF4y2Ba。gydF4y2Ba源数据gydF4y2Ba都提供了这篇论文。gydF4y2Ba
参考文献gydF4y2Ba
Friesen, N., Fritsch, R. M. & Blattner, F. R.gydF4y2Ba葱属植物gydF4y2Ba(gydF4y2BaAlliaceaegydF4y2Ba)基于核糖体DNA及其序列。gydF4y2BaAlisogydF4y2Ba22gydF4y2Ba; 372-395(2006)。gydF4y2Ba
李麒麒等。植物的系统发育与生物地理学gydF4y2Ba葱属植物gydF4y2Ba(Amaryllidaceae: Allieae)基于核糖体内转录间隔序列和叶绿体rps16序列,重点纳入中国特有种。gydF4y2Ba安。机器人。gydF4y2Ba106gydF4y2Ba, 709-733(2010)。gydF4y2Ba
艾迪,c.c.等。沉默洋葱催泪因子合成酶可引起硫次生代谢物谱的显著变化。gydF4y2Ba植物杂志。gydF4y2Ba147gydF4y2Ba, 2096-2106(2008)。gydF4y2Ba
Fritsch, R. & Keusgen, M.半胱氨酸硫化物在该属中的发生及其分类意义gydF4y2Ba葱属植物gydF4y2Bal . (Alliaceae)。gydF4y2Ba植物化学gydF4y2Ba67gydF4y2Ba(2006)。gydF4y2Ba
Yoshimoto, N. & Saito, K. s . alk (en)酰基半胱氨酸亚砜属gydF4y2Ba葱属植物gydF4y2Ba建议的生物合成、化学转化和生物活性。gydF4y2Ba[au:]gydF4y2Ba70gydF4y2Ba中文信息学报,4123-4137(2019)。gydF4y2Ba
付晓燕,李春春,周晓燕,刘生,吴峰gydF4y2Ba诉dahliaegydF4y2Ba番茄/马铃薯/洋葱伴生的番茄植株gydF4y2Ba科学。代表。gydF4y2Ba6gydF4y2Ba, 36445(2016)。gydF4y2Ba
Yan, Z.等。41种芳香植物对黄龙病相关菌媒介柑桔木虱的驱避作用。gydF4y2Ba生态。另一个星球。gydF4y2Ba10gydF4y2Ba科学通报,12940-12948(2020)。gydF4y2Ba
Kang, M.等。一个chromosome-levelgydF4y2BaCamptotheca acuminatagydF4y2Ba基因组组装为喜树碱生物合成的进化起源提供了见解。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 3531(2021)。gydF4y2Ba
夏,E.等。茶树参考基因组和81个品种的重测序为茶树基因组的进化和适应提供了新的思路。gydF4y2Ba摩尔。植物gydF4y2Ba13gydF4y2Ba中文信息学报,1013-1026(2020)。gydF4y2Ba
郭,S.等。对414份栽培和野生西瓜材料进行重测序,确定了果实品质性状的选择。gydF4y2BaNat,麝猫。gydF4y2Ba51gydF4y2Ba, 1616-1623(2019)。gydF4y2Ba
Sun, X.等。大蒜染色体水平的基因组组装(gydF4y2Ba大蒜gydF4y2Ba)提供了基因组进化和大蒜素生物合成的见解。gydF4y2Ba摩尔。植物gydF4y2Ba13gydF4y2Ba中文信息学报,1328-1339(2020)。gydF4y2Ba
Yusupov, Z.等。大蒜科葱属植物的系统基因组学研究为洋葱驯化研究提供了新的思路。gydF4y2Ba植物潜水员gydF4y2Ba43gydF4y2Ba, 102-110(2021)。gydF4y2Ba
佩夫利,e。b。进来gydF4y2Ba高科技与微繁殖3gydF4y2Ba(Bajaj编,Y.P.S.) 244-261(施普林格柏林海德堡,1992年)。gydF4y2Ba
Budylin, M. V., Kan, L. Y., Romanov, V. S. & Khrustaleva, L. GISH种间杂交后代的研究gydF4y2Ba洋葱gydF4y2Bal .,gydF4y2Ba葱属植物fistulosumgydF4y2BaL.对霜霉病有相对抗性。gydF4y2Ba拉斯。J.植物生理学。gydF4y2Ba50gydF4y2Ba, 387-394(2014)。gydF4y2Ba
Benke, a.p., Mahajan, V., Manjunathagowda, d.c.和Mokat, d.n.种间杂交gydF4y2Ba葱属植物gydF4y2Ba作物:现状及说明书。gydF4y2Ba麝猫。Resour。作物的另一个星球gydF4y2Ba。gydF4y2Ba69gydF4y2Ba, 1-9(2022)。gydF4y2Ba
Umehara, M.等。种间杂交gydF4y2Ba葱属植物fistulosumgydF4y2Ba和gydF4y2Ba葱属植物schoenoprasumgydF4y2Ba揭示富含胡萝卜素的表型。gydF4y2BaEuphyticagydF4y2Ba148gydF4y2Ba, 295(2006)。gydF4y2Ba
斯蒂斯,s.p.等人。硫代亚硫酸盐耐受性是一种洋葱非典型病原菌的毒力策略。gydF4y2Ba咕咕叫。医学杂志。gydF4y2Ba30.gydF4y2Ba, 3130 - 3140。e6(2020)。gydF4y2Ba
Han, T.等。多倍体促进物种多样化gydF4y2Ba葱属植物gydF4y2Ba通过生态转变。gydF4y2Ba叶绿醇。gydF4y2Ba225gydF4y2Ba, 571-583(2020)。gydF4y2Ba
Ricroch, A, Yockteng, R, Brown, S. C.和Nadot, S.几种栽培葱属植物基因组大小的进化。gydF4y2Ba基因组gydF4y2Ba48gydF4y2Ba, 511-520(2005)。gydF4y2Ba
rihie, A., Walenz, B. P., Koren, S. & Phillippy, A. M. mercury:基因组组装的无参考质量,完整性和分阶段评估。gydF4y2Ba基因组医学杂志。gydF4y2Ba21gydF4y2Ba, 245(2020)。gydF4y2Ba
Tsukazaki, H.等。基于ssr的洋葱染色体图谱的构建gydF4y2Ba葱属植物fistulosumgydF4y2Ba)。gydF4y2Ba定理。达成。麝猫。gydF4y2Ba117gydF4y2Ba, 1213-1223(2008)。gydF4y2Ba
陈欧,年代,j . &江:评估使用LTR组装基因组装配质量指数(LAI)。gydF4y2Ba核酸类。gydF4y2Ba46gydF4y2Ba, e126(2018)。gydF4y2Ba
Galindo-González, L., Mhiri, C., Deyholos, M. K. & Grandbastien, M. A. ltr -植物中的逆转录转座子:进化的引擎。gydF4y2Ba基因gydF4y2Ba626gydF4y2Ba, 14-25(2017)。gydF4y2Ba
肖辉,蒋宁,Schaffner, E. Stockinger, E. J. & van der Knaap, E.逆转录转座子介导的基因复制在番茄果实形态变异中的作用。gydF4y2Ba科学gydF4y2Ba319gydF4y2Ba, 1527-1530(2008)。gydF4y2Ba
芬克斯,R.等。洋葱(洋葱)首次基因组组装的启示gydF4y2Ba洋葱gydF4y2Ba)。gydF4y2BaG3(贝塞斯达)gydF4y2Ba11gydF4y2Ba, jkab243(2021)。gydF4y2Ba
舒克拉等人。洋葱基因组资源:一个基因组学和生物信息学驱动的洋葱育种资源。gydF4y2Ba植物基因gydF4y2Ba8gydF4y2Ba, 9-15(2016)。gydF4y2Ba
李希曼,高登,G. T.和Buell, C. R.化学多样性进化中的基因和基因组复制:来自Lamiaceae研究的观点。gydF4y2Ba咕咕叫。当今。植物医学杂志。gydF4y2Ba55gydF4y2Ba, 74-83(2020)。gydF4y2Ba
Imai, S.等。一种让眼睛流泪的洋葱酶。gydF4y2Ba自然gydF4y2Ba419gydF4y2Ba, 685-685(2002)。gydF4y2Ba
Arakawa, T.等。裂解因子合酶对短寿命亚磺酸立体控制转化的研究。gydF4y2BaACS Catal。gydF4y2Ba10gydF4y2Ba, 9-19(2019)。gydF4y2Ba
沈旸,等。染色体水平和单倍型解析基因组提供了广藿香四倍体杂交起源的见解。gydF4y2BaCommun Nat。gydF4y2Ba13gydF4y2Ba, 3511(2022)。gydF4y2Ba
Rose, P., Whiteman, M., Moore, P. K. &朱永忠。葱属植物中生物活性S-alk(en)yl半胱氨酸亚胺代谢物:潜在治疗药物的化学作用。gydF4y2Ba众议员纳特。gydF4y2Ba22gydF4y2Ba; 351-368(2005)。gydF4y2Ba
石田,M.,原,M.,福野,N., Kakizaki, T. &森光,Y.芥子油苷代谢、功能和改良油菜科蔬菜的育种。gydF4y2Ba品种。科学。gydF4y2Ba64gydF4y2Ba, 48-59(2014)。gydF4y2Ba
Sønderby, I. E, Geu-Flores, F. & Halkier, B. A.硫代葡萄糖苷基因的生物合成及其进展。gydF4y2Ba植物科学动态。gydF4y2Ba15gydF4y2Ba, 283-290(2010)。gydF4y2Ba
Friesen, N, Pollner, S, Bachmann, K. & Blattner, F. R. rapd和非编码叶绿体DNA揭示了栽培的单一起源gydF4y2Ba葱属植物fistulosumgydF4y2Ba从gydF4y2Ba答:altaicumgydF4y2Ba(Alliaceae)。gydF4y2Ba点。j .机器人。gydF4y2Ba86gydF4y2Ba, 554-562(1999)。gydF4y2Ba
辛格,B.和罗摩克里希纳,Y.威尔士洋葱(gydF4y2Ba葱属植物fistulosumgydF4y2Ba米佐拉姆邦一种很有前途的调味烹饪草本植物。gydF4y2Ba印第安·j·希尔gydF4y2Ba。gydF4y2Ba农业gydF4y2Ba30.gydF4y2Ba, 201-208(2017)。gydF4y2Ba
Ford-Lloyd, B.V. & Armstrong, S.J. ingydF4y2Ba蔬菜作物的遗传改良gydF4y2Ba(eds。Kalloo, G. & Bergh, B.O.) 51-58 (Pergamon,阿姆斯特丹,1993)。gydF4y2Ba
卡亚特,F.,穆罕默德,A.和易卜拉欣,A. ingydF4y2Ba植物育种策略的进展:蔬菜作物:第10卷:叶子,花头,绿豆荚,蘑菇和松露gydF4y2Ba(eds。Al-Khayri, j.m., Jain, S.M. & Johnson, D.V.) 135-182(施普林格International Publishing, Cham, 2021)。gydF4y2Ba
福特-劳埃德,b.v.和阿姆斯特朗,s.j. ingydF4y2Ba蔬菜作物的遗传改良gydF4y2Ba(eds。Kalloo, G. & Bergh, B.O.) 51-58 (Pergamon,阿姆斯特丹,1993)。gydF4y2Ba
Marcais, G. & Kingsford, C.一种快速、无锁的方法,用于有效并行计数的发生次数gydF4y2BakgydF4y2Ba即。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 764-770(2011)。gydF4y2Ba
korean, S.等。Canu:可扩展和准确的长读组件通过自适应k-mer加权和重复分离。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 722-736(2017)。gydF4y2Ba
沃克,b.j.等。Pilon:综合微生物变异检测和基因组组装改进的集成工具。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Ba, e112963(2014)。gydF4y2Ba
Guan, D.等。鉴定和去除原始基因组组合中的单倍型重复。gydF4y2Ba生物信息学gydF4y2Ba36gydF4y2Ba中文信息学报,2896-2898(2020)。gydF4y2Ba
Lam, e.t.等人。基于纳米通道阵列的基因组图谱用于结构变异分析和序列组装。gydF4y2Ba生物科技Nat。》。gydF4y2Ba30.gydF4y2Ba, 771-776(2012)。gydF4y2Ba
Li, H. & Durbin, R.基于Burrows-Wheeler变换的快速准确短读对齐。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 1754-1760(2009)。gydF4y2Ba
Tarailo‐Graovac, M. & Chen, N.使用RepeatMasker识别基因组序列中的重复元素。gydF4y2Ba咕咕叫。Protoc。Bioinforma。gydF4y2Ba25gydF4y2Ba, 4.10.1-4.10.14(2009)。gydF4y2Ba
斯坦克,M.等。从头开始预测替代转录本。gydF4y2Ba核酸类。gydF4y2Ba34gydF4y2Ba, w435-w439(2006)。gydF4y2Ba
李,s,等。观赏植物染色体水平基因组组装、注释和进化分析gydF4y2Ba文竹gydF4y2Ba。gydF4y2Ba长的矮。Res。gydF4y2Ba7gydF4y2Ba, 48(2020)。gydF4y2Ba
Harkess, A.等。芦笋基因组揭示了年轻Y染色体的起源和进化。gydF4y2BaCommun Nat。gydF4y2Ba8gydF4y2Ba, 1279(2017)。gydF4y2Ba
Yan, L.等。的基因组gydF4y2Ba石斛兰officinalegydF4y2Ba阐明了重要的中国传统兰科植物的生物学。gydF4y2Ba摩尔。植物gydF4y2Ba8gydF4y2Ba中文信息学报,922-934(2015)。gydF4y2Ba
张国强等。的gydF4y2BaApostasiagydF4y2Ba基因组与兰花的进化。gydF4y2Ba自然gydF4y2Ba549gydF4y2Ba, 379-383(2017)。gydF4y2Ba
蔡,J.等。兰花的基因组序列gydF4y2Ba蝴蝶兰属equestrisgydF4y2Ba。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba, 65-72(2015)。gydF4y2Ba
al - mssallem, i.s.等。枣椰树基因组序列gydF4y2Ba凤凰dactyliferagydF4y2BalgydF4y2BaCommun Nat。gydF4y2Ba4gydF4y2Ba, 2274(2013)。gydF4y2Ba
高,Z.-Y。et al。通过重组自交系重测序和改良亲本基因组序列剖析超级杂交稻产量相关位点。gydF4y2Ba自然科学进展。美国gydF4y2Ba110gydF4y2Ba, 14492-14497(2013)。gydF4y2Ba
Ming, R.等。菠萝基因组与CAM光合作用的进化。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba, 1435-1442(2015)。gydF4y2Ba
阿尔伯特,V. A.等。的gydF4y2BaAmborellagydF4y2Ba基因组与开花植物的进化。gydF4y2Ba科学gydF4y2Ba342gydF4y2Ba, 1467(2013)。gydF4y2Ba
贝尔瑟,C.等。利用纳米孔长读数和光学图的植物基因组的染色体尺度组装。gydF4y2BaNat。植物gydF4y2Ba4gydF4y2Ba中文信息学报,879-887(2018)。gydF4y2Ba
B. J.哈斯gydF4y2Ba拟南芥gydF4y2Ba使用最大转录物比对组装的基因组注释。gydF4y2Ba核酸类。gydF4y2Ba31gydF4y2Ba, 5654-5666(2003)。gydF4y2Ba
tRNAscan-SE:在基因组序列中改进转移RNA基因检测的程序。gydF4y2Ba核酸类。gydF4y2Ba25gydF4y2Ba, 955-964(1997)。gydF4y2Ba
naawrocki, e.p., Kolbe, d.l. & Eddy, s.r. Infernal 1.0: RNA比对的推断。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba科学通报,1335-1337(2009)。gydF4y2Ba
De Bie, T., Cristianini, N., Demuth, J. P. & Hahn, M. W. CAFE:基因家族进化研究的计算工具。gydF4y2Ba生物信息学gydF4y2Ba22gydF4y2Ba, 1269-1271(2006)。gydF4y2Ba
Tang, H.等。植物基因组的共线性与共线性。gydF4y2Ba科学gydF4y2Ba320gydF4y2Ba, 486-488(2008)。gydF4y2Ba
Steinbiss, S, Willhoeft, U, Gremme, G. & Kurtz, S.精细粒度注释和分类gydF4y2Ba新创gydF4y2Ba预测LTR逆转录转座子。gydF4y2Ba核酸类。gydF4y2Ba37gydF4y2Ba, 702 - 7013(2009)。gydF4y2Ba
Liu, H.等。银杏近乎完整的基因组揭示了裸子植物的进化。gydF4y2BaNat。植物gydF4y2Ba7gydF4y2Ba, 748-756(2021)。gydF4y2Ba
李,G.等。一个高质量的基因组组装突出黑麦基因组特征和重要的农艺学基因。gydF4y2BaNat,麝猫。gydF4y2Ba53gydF4y2Ba, 574-584(2021)。gydF4y2Ba
牛,S.等。油松基因组和甲基组揭示了针叶树进化的关键特征。gydF4y2Ba细胞gydF4y2Ba185gydF4y2Ba, 204 - 217。e14(2022).
Lee, T., Guo, H., Wang, X., Kim, C. & Paterson, a. H. SNPhylo:从大量SNP数据构建系统发育树的管道。gydF4y2BaBMC基因组学gydF4y2Ba15gydF4y2Ba, 162(2014)。gydF4y2Ba
张超,董生,徐军,何伟,杨涛,杨涛。PopLDdecay:一种快速有效的基于可变调用格式文件的链接不平衡衰减分析工具。gydF4y2Ba生物信息学gydF4y2Ba35gydF4y2Ba, 1786-1788(2018)。gydF4y2Ba
Chen, H., Patterson, N.和Reich, D.作为选择性扫描测试的种群分化。gydF4y2Ba基因组Res。gydF4y2Ba20.gydF4y2Ba, 393-402(2010)。gydF4y2Ba
Rektorisova, M., Hrbek, V., Jiru, M., Ovesna, J.和Hajslova, J.用液相色谱-串联质谱法测定大蒜中S-Alk(en)yl- l-半胱氨酸亚胺在7个月内的变异性。gydF4y2Ba植物性食物。减轻。gydF4y2Ba75gydF4y2Ba, 376-382(2020)。gydF4y2Ba
致谢gydF4y2Ba
我们感谢美国国家植物种质系统提供的gydF4y2Ba答:fistulosumgydF4y2Ba登记入册。我们感谢浙江大学农业生物与环境科学分析中心吴晓丹女士和方梅女士为ACSOs定量提供的技术支持,以及阜阳区农业技术推广中心张全芳女士为ACSOs定量提供的技术支持gydF4y2Ba答:fistulosumgydF4y2BavargydF4y2Ba。viviparumgydF4y2Ba加入“HZC”。本研究得到了中国农业部农业科学研究系统(CARS-24-B-01, Y-H.Z.)和中央高校基本科研业务费专项基金(+226−2022-00100,M-F.Z.)的资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及单位gydF4y2Ba
贡献gydF4y2Ba
J-Q.Y。,M-F.Z., and Y-H.Z. conceived of and designed the experiments. N.L., Z.H., X.H., Y.Z., L.K., and Y.W. performed genome assembly and assessment, comparative genome analysis, and other bioinformatic analyses; J-H.Y., X.L., Y-M.Z., G.D., Y-M.M., K.Z., Y-Y.M., M.A., and Y.X. conducted transcriptome assays and re-sequencing; J.M. and H.F. provided most of the答:fistulosumgydF4y2Ba登记入册;j.m.、H.Y和M.Z.进行了田间栽培和表型调查;N.L.和z.h写了初稿;J-Q.Y。,M-F.Z., and Y-H.Z. revised the manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益冲突。gydF4y2Ba
同行评审gydF4y2Ba
同行评议信息gydF4y2Ba
自然通讯gydF4y2Ba感谢匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Bab施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接从版权所有者处获得许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
廖宁,胡忠,缪杰,gydF4y2Baet al。gydF4y2Ba洋葱的染色体水平基因组组装揭示了洋葱的基因组进化和风味形成gydF4y2Ba葱属植物gydF4y2Ba作物。gydF4y2BaNat CommungydF4y2Ba13gydF4y2Ba, 6690(2022)。https://doi.org/10.1038/s41467-022-34491-3gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467-022-34491-3gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba