摘要
原肠胚形成控制早期胚胎中细胞多样性和轴型的出现。在哺乳动物中,这种转化是由胚胎和胚胎外组织界面上的动态信号中心协调的1,2,3..阐明体内轴形成的分子框架是我们理解人类发展的基础4,5,6推进基于干细胞的再生方法7.在这里,我们使用空间转录组学和基于干细胞的胚胎模型阐明了子宫内绒猴胚胎的早期原肠胚形成。基于高斯过程回归的3D转录组描述了前内脏内胚层的出现,其特点是保守(HHEX,LEFTY2,LHX1)和灵长类特有的(POSTN,SDC4,FZD5)因素。WNT信号在空间上协调胚胎盘中原始条纹的形成,并被SFRP1而且SFRP2维持前区多能性。羊膜规格出现在胚胎盘的边界穿过ID1,ID2下而且ID3对BMP信号的响应,为灵长类多能干细胞(PSCs)的羊膜分化提供了发育基础。空间特征图谱显示,启动的绒猴PSCs表现出与胚胎前盘最高的相似性,而未启动的PSCs则类似于着床前的外胚层。我们的3D转录组模型揭示了灵长类胚胎中谱系规范的分子密码,并为破译人类发育提供了体内参考。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
人类生殖系干细胞
信号转导与靶向治疗开放获取10月2日
德国与麻省理工学院:德国与安文东
BIOspektrum开放获取8月30日
哺乳动物胚胎发生的六种转录组图谱描述了三种不同植入模式的代谢调节
自然通讯开放获取2022年6月16日
访问选项
访问《自然》和其他54种《自然》杂志
获取Nature+,我们最超值的在线订阅
每月29.99美元
随时取消
订阅这本杂志
收到51个印刷问题和在线访问
199.00美元一年
每期仅需3.90美元
租或购买这篇文章
只要这篇文章,只要你需要它
39.95美元
价格可能受当地税收的影响,在结账时计算

数据可用性
从合子到原肠胚的三维虚拟截面的绒猴转录组可在线获取(http://131.111.33.80/marmoset3D/).请在开始时允许5-10秒加载转录组数据。STEP和单细胞RNA-seq数据可在ArrayExpress和Gene Expression Omnibus存储库获得e - mtab - 9367(灵长类动物原肠发育的空间胚胎分析),e - mtab - 9349(启动和幼稚绒猴多能干细胞作为灵长类动物发育的模型),e - mtab - 9388(http://www.human-gastrula.net)9,GSE136447(ref。12),GSE74767(ref。25),GSE76267(ref。39),e - mtab - 6819(ref。43),GSE134571(ref。16).3D胚胎重建可通过Morphosource获得,登录号为:CS5(000419264),早期CS6(000419284),晚期CS6(000419272)和CS7(000419278)。源数据提供了这篇论文。
代码的可用性
所使用的代码可在GitHub (https://github.com/Boroviak-Lab/SpatialModelling).
参考文献
Tam, P. P. & Loebel, D. A.小鼠胚胎发生中的基因功能:为原肠胚形成做好准备。Nat. Rev. Genet。8, 368-381(2007)。
Rossant, J. & Tam, P. P. L. L.囊胚谱系形成,早期胚胎不对称和轴模式在小鼠。发展136, 701-713(2009)。
阿诺德,S. J. &罗伯逊,E. J.做出承诺:早期小鼠胚胎中的细胞谱系分配和轴图。细胞生物学。10, 91-103(2009)。
罗桑特,J.和塔姆,P. P. L.探索早期人类胚胎发育。科学360, 1075-1076(2018)。
Boroviak, T. & Nichols, J.灵长类胚胎发生预测人类naïve多能性的特征。发展144, 175-186(2017)。
Simunovic, M. & Brivanlou, A. H.胚状体,类器官和类原肠胚:理解胚胎发生的新方法。发展144, 976-985(2017)。
Tabar, V. & Studer, L.再生医学中的多能干细胞:挑战和最新进展。Nat. Rev. Genet。15, 82-92(2014)。
罗斯,C. & Boroviak, T. E.卵黄囊在灵长类胚胎发生中的起源和功能。Commun Nat。11, 3760(2020)。
泰勒,r.c.v.等。人类原肠发育胚胎的单细胞转录组特征。自然600, 285-289(2021)。
degllincerti, A.等人。体外附着人胚胎的自组织。自然533, 251-254(2016)。
Shahbazi, M. N.等。胚胎在缺少母体组织时人类胚胎的自我组织细胞生物学。18, 700-708(2016)。
向,L.等。3d培养的人类原肠胚发育图景。自然577537-542(2019)。
Ma, H.等人。食蟹猴胚胎早期原肠形成后的体外培养。科学366, eaax7890(2019)。
牛毅等。利用长期体外胚胎培养解剖灵长类动物早期着床后发育。科学366, eaaw5754(2019)。
邵,Y.等。人多能干细胞在仿生植入样生态位的自组织羊膜发生。Nat。板牙。16, 419-425(2017)。
郑毅,等。利用干细胞控制人类外胚层和羊膜发育的模型。自然573, 421-425(2019)。
邵,Y.等。基于多能干细胞的植入后人羊膜囊发育模型。Commun Nat。8, 208(2017)。
温弗拉斯,A., Sorre, B., Etoc, F., Siggia, E. D. & Brivanlou, A. H.一种重现人类胚胎干细胞早期胚胎空间模式的方法。Nat方法。11, 847-854(2014)。
Martyn, I., Siggia, E. D. & Brivanlou, a . H.绘制细胞迁移和命运在一个原肠样模型到人类原始条纹。发展146, dev179564(2019)。
佐藤,K.等。使用高效基因组编辑生成严重联合免疫缺陷的非人灵长类动物模型。细胞干细胞19, 127-138(2016)。
Okano, H., Hikishima, K., Iriki, a . & Sasaki, E.普通绒猴作为生物医学和神经科学研究应用的新型动物模型系统。Semin。胎儿新生儿医学。17, 336-340(2012)。
佐佐木,等。培育具有生殖系传播的转基因非人灵长类动物。自然459, 523-527(2009)。
皮切利等人。使用Smart-seq2从单细胞获得全长RNA-seq。Protoc Nat。9, 171-181(2014)。
Boroviak, T.等。人类、绒猴和小鼠胚胎的单细胞转录组分析揭示了植入前发育的共同和不同特征。发展145, dev167833(2018)。
中村,T.等。小鼠、猴子和人类多能性的发育坐标。自然537, 57-62(2016)。
拉斯穆森,c.e. &威廉姆斯,c.k.i.机器学习的高斯过程第二章(麻省理工学院出版社,2006)。
马塞隆,G.克里格格,矩形帕萨潘诺périphérie。注意Geostatistique28(1960)。
Svensson, V., Teichmann, s.a. & Stegle, O. SpatialDE:空间变异基因的鉴定。Nat方法。15, 343-346(2018)。
彭,G.等。中原肠胚小鼠谱系命运和细胞身份分子注释的空间转录组。Dev细胞。36, 681-697(2016)。
彭,G.等。小鼠早期胚胎谱系分配和组织结构的分子结构。自然572, 528-532(2019)。
Rivera-Pérez, J. A. & Magnuson, T.在小鼠中形成原始条纹之前,Brachyury和Wnt3的局部激活。Dev,杂志。288, 363-371(2005)。
Perea-Gomez, A.等人。前内脏内胚层的节点拮抗剂可防止多个原始条纹的形成。Dev细胞。3., 745-756(2002)。
Molè, m.a.等。人类胚胎发生的单细胞特征确定多能性转变和假定的前低母细胞中心。Commun Nat。12, 3679(2021)。
Wamaitha, S. E.等。igf1介导的人胚胎干细胞自我更新概括了胚胎生态位。Commun Nat。11, 764(2020)。
佐赞,我,等等。转录调节因子ZNF398在人PSCs中介导tgf - β的多能性和上皮特性下游Commun Nat。11, 2364(2020)。
Nakaki, F. & Saitou, M. PRDM14:多能性和表观遗传重编程的独特调节器。学生物化学的发展趋势。科学。39, 289-298(2014)。
Irie, N.等。SOX17是人类原始生殖细胞命运的关键指标。细胞160, 253-268(2015)。
小林,T.和苏拉尼,m.a.关于人类生殖细胞的起源。发展145, dev150433(2018)。
Sasaki, K.等。食蟹猴的生殖细胞命运在新生羊膜中被指定。Dev细胞。39, 169-185(2016)。
Tewary, M.等。反应-扩散和位置信息的逐步模型支配着自组织的人类类原肠周模式。发展https://doi.org/10.1242/dev.149658(2017)。
杨,R.等。羊膜信号对灵长类动物中胚层的形成至关重要。Commun Nat。12, 5126(2021)。
霍勒纳格尔,厄尔曼,V.,海默,J., Rüther, U. &诺德海姆,A.。Id基因是胚胎干细胞诱导骨形态发生蛋白的直接靶点。生物。化学。274, 19838-19845(1999)。
Messmer, T.等人。在单细胞分辨率下,幼稚和启动的人类多能干细胞的转录异质性。细胞的代表。26, 815-824(2019)。
郭,G.等。人类幼稚外胚层细胞具有不受限制的谱系潜力。细胞干细胞https://doi.org/10.1016/j.stem.2021.02.025(2021)。
董,C.等。滋养层干细胞来源于naïve人类多能干细胞。eLife9, e52504(2020)。
Linneberg-Agerholm等人。Naïve人类多能干细胞响应Wnt, Nodal和LIF信号,产生可扩展的naïve胚胎外内胚层。发展146, dev180620(2019)。
O 'Rahilly, R. & Müller, F.人类胚胎发育阶段:修订和新的测量方法。细胞、组织、器官192, 73-84(2010)。
哈洛,C. R.,赫恩,J. P.和霍奇斯,J. K.绒猴的排卵:内分泌学,预测和检测。j .性。103, 17-24(1984)。
Clark, S. J.等人scNMT-seq能够在单细胞中对染色质可达性、DNA甲基化和转录进行联合分析。Commun Nat。9, 781(2018)。
麦考利,i.c.,庞廷,c.p. & Voet, T.单细胞多重组学:从单细胞进行多重测量。趋势麝猫。33, 155-168(2017)。
克鲁格,f·特里姆·加洛尔。Babraham生物信息学大量的http://www.bioinformatics.babraham.ac.uk/projects/trim /(2016)。
Dobin, A.等人。STAR:超快通用RNA-seq对准器。生物信息学29, 15-21(2013)。
Liao, Y., Smyth, G. K. & Shi, W. featuremets:一种高效的通用程序,用于分配序列读取基因组特征。生物信息学30., 923-930(2014)。
中村,T.等。食蟹猴早期胚胎和培养胚胎干细胞的单细胞转录组。科学。数据4, 170067(2017)。
费尔南德斯等人。WNT受体FZD7是维持人类胚胎干细胞多能状态所必需的。国家科学院学报美国111, 1409-1414(2014)。
好的,h。人类滋养层干细胞的来源。细胞干细胞22, 50-63 e6(2018)。
McQuin, C.等。CellProfiler 3.0:生物学的下一代图像处理。公共科学图书馆杂志。16, e2005970(2018)。
O 'Hagan, A.预测的曲线拟合和优化设计。J. R.统计社爵士。Bhttps://doi.org/10.1111/j.2517-6161.1978.tb01643.x(1978)。
Rasmussen, C. E. & Nickisch, H.机器学习(GPML)工具箱的高斯过程。j·马赫。学习。Res。11, 3011-3015(2010)。
Kalaitzis, A. A. & Lawrence, N. D.一种通过高斯过程回归对差异表达基因表达时间过程进行排序的简单方法。BMC Bioinform。12, 180(2011)。
一些用概率论处理的显著性检验。数学。剑桥菲洛斯教授。Soc。https://doi.org/10.1017/S030500410001330X(1935)。
卡斯,R. E. & Raftery, A. E.贝叶斯因子。j。统计协会。https://doi.org/10.1080/01621459.1995.10476572(1995)。
Adewumi, O.等。国际干细胞计划对人类胚胎干细胞系的鉴定。生物科技Nat。》。25, 803-816(2007)。2007 257.
Tewary, M.等。高通量微模式平台揭示了原肠成形期周围相关与神经形成前相关命运模式的节点依赖等分。公共科学图书馆杂志。17, e3000081(2019)。
维克斯,A.等。在微图案衬底上镀人iPSC细胞系揭示了ITGB1 nsSNV在内胚层形成中的作用。干细胞代表16, 2628-2641(2021)。
施耐德,C. A.,拉斯班德,W. S.和Eliceiri, K. W. NIH图像到ImageJ: 25年的图像分析。Nat方法。9, 671-675(2012)。
汤姆逊,J. A.卡利什曼,J. &赫恩,J. P.普通绒猴非手术子宫期植入前胚胎收集。【医学】灵长类动物。23, 333-336(1994)。
Boroviak, T.等。谱系特异性分析描述了哺乳动物胚胎发生中幼稚多能性的出现和进展。Dev细胞。35, 366-382(2015)。
阿什本,等人。基因本体论:生物学统一的工具。Nat,麝猫。25, 25-29(2000)。
碳,S.等。基因本体论资源:20年了,仍然很强大。核酸测定。47, d330-d338(2019)。
韦翰,H。ggplot2:数据分析的优雅图形(施普林格,2009);https://doi.org/10.1007/978-0-387-98141-3
冲积区:冲积区的分层语法。J。开源软件。https://doi.org/10.21105/joss.02017(2020)。
Bergmann, S, Penfold, C, Slatery, E.和Boroviak, T. E.灵长类动物植入阶段的空间胚胎分析(STEP)。协议交换https://doi.org/10.21203/rs.3.pex-1899/v1(2022)。
Hertig, a .关于人类卵子的着床前阶段:从发育的第2天到第5天,四个正常和四个异常标本的描述。普通发布版。Embryol。35, 119-220(1954)。
Hertig, A. T. & Rock, J.两个长绒毛前阶段的人类卵子,排卵年龄分别约为11天和12天。普通发布版。Embryol。29, 127-156(1941)。
确认
我们感谢A. Roberts和剑桥绒猴研究机构的工作人员提供绒猴幼崽;德国灵长类动物中心内分泌实验室的成员进行激素分析,退行性疾病平台的成员提供帮助;以及S. Li、C.芒格、M.辛德勒、I. Ampartzidis、G. Jowett、C. Kyprianou和G. Burton对手稿的讨论和评论。P. Humphreys和D. Clements支持成像;A. Surani和J. Gurdon为我们提供了使用Gurdon研究所计算设施的机会;C. Bradshaw提供高性能计算方面的帮助;以及X. Zhao和R. Hamilton的建议和生物信息学支持。本研究得到了威康基金会(WT RG89228)、滋养层研究中心、艾萨克·牛顿基金会和JSPS KAKENHI (15H02360, 19H05759)的支持。E.S.由威康基金会博士奖学金(WT108438/C/15/Z)支持。
作者信息
作者及隶属关系
贡献
t.e.b.、s.b.、e.s Slatery、C.A.P.和D.S.设计并进行了实验并撰写了手稿,C.D.建立了定时绒猴交配和提取子宫,T.N.K.对PSCs进行了染色和成像,C.A.P.、e.s Slatery和D.S.进行了生物信息学,s.b.、D.S.、S.E.S.和e.s Slatery建立了虚拟重建,C.A.P.开创了GPR建模3D转录组,K.K.和e.s Sasaki提供了绒猴植入前胚胎。R.B.提供了绒猴植后胚胎,A.V.和M.T.提供了专业知识并制作了微图案板,f.h.、W.R.和S.C.提供了成像和高通量RNA-seq文库制备的专业知识和设施,T.E.B.构思、协调和监督了该项目。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
同行评审
同行评审信息
自然感谢付建平、帕特里克·谭和其他匿名审稿人对本研究的同行评议所作的贡献。同行评审报告是可用的。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展的数据图形和表格
扩展数据图1 STEP方法概述
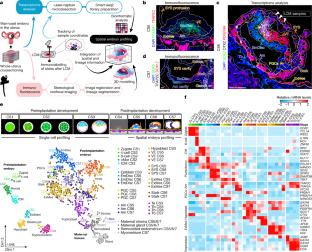
妊娠子宫冷冻切片:取出妊娠绒猴子宫,嵌入O.C.T.并快速冷冻。为了提供最好的RNA质量,对组织进行激光捕获显微解剖(LCM)介导的Smart-Seq2测序。2.立体免疫荧光成像:每隔一至三分之一包含植入胚胎的切片进行已建立的谱系标记的免疫染色。用共聚焦显微镜或原子显微镜生成磁片扫描图像。12µm厚的冷冻切片按升序编目,用于确定lcm转录组的z坐标。3.LCM辅助样本收集:每隔一至三分之一的切片进行LCM样本收集,所有LCM处理的切片随后进行带有谱系标记的免疫荧光(IF)。为了获取转录组样本,拍摄了该部分的照片。然后,使用蔡司PALM LCM软件在感兴趣的区域中选择一到三个细胞,并用激光切割出来。 In a second step, a pulse laser catapulted the sample into a collection tube with lysis buffer. Then, a second picture was taken of the section at the matching position with the sample removed and matched to the collection tube and image file. Each STEP-transcriptome was assigned an individual ID and lineage identity based on the original location within the embryo cryosection, i.e. the cut-out location of the LCM-sample. 4. Sample annotation: Collected LCM-samples were subjected to the Smart-Seq2 protocol and sequenced to an average depth of > 2 million 150bp paired reads. LCM-sample lineage identity was assigned based on the position within the embryo. Sample annotation was performed manually, side-by-side with phase contrast images acquired during sample collection and the confocal image with lineage markers (e.g. PDGFRA, OTX2, SOX2) of the same section. In addition, annotations were guided by the density and orientation of DAPI-labelled nuclei, which allowed us to discriminate between neighbouring tissues. We refined annotations by integration of lineage marker expression from immunofluorescence stainings or STEP-transcriptome data. Samples with more than one lineage signature were annotated as mixed and removed from downstream analysis. 5. Image registration and lineage segmentation: Images were aligned by image registration in Fiji, whereby each image was registered to the DAPI channel of the previous image. Next, nuclei were segmented into individual objects using Cell Profiler. For lineage segmentation, the segmented nuclei were assigned lineages based on lineage marker immunostaining (e.g. POU5F1 to demarcate the EmDisc and Amnion, PDGFRA for ExMes, VE and SYS, TFAP2C for trophoblast, Amnion and PGCs), and taking into account known anatomical features of the embryo (e.g. EmDisc resides in-between VE and Amnion). 6. Transcriptome coordinate integration: The X and Y coordinates of the annotated transcriptomes were compiled into MATLAB matrices. 7.Virtual 3D embryo reconstruction: We generated primary surfaces in MATLAB by triangulation (see Methods). In a second step, embryonic and extraembryonic surfaces were smoothened in Blender, an open-source 3D modelling and animation software. 8. Gaussian Process Regression (GPR) over LCM samples: LCM spatial transcriptome sample coordinates were integrated into the 3D embryo models and continuous expression patterns between discrete LCM samples were inferred using Gaussian Process Regression (see Methods). Since expression patterns may be discontinuous across tissue types, we inferred an independent GPR model for each tissue at each stage. 9. GPR spatial transcriptome models and virtual cross sections: Final smooth GPR gene expression patterns could be displayed in 3D on embryo models. Defined coordinates were used to extract expression patterns in each lineage for visualization of virtual cross sections. Scale bars represent 100 µm. CS, Carnegie stage; O.C.T, Optimal cutting temperature compound (used to mount uteri); EmDisc, Embryonic disc; Am, Amnion; SYS, Secondary Yolk Sac; VE, Visceral Endoderm; Tb, Trophoblast; ExMes, Extraembryonic mesoderm; PGCs, Primordial Germ Cells.
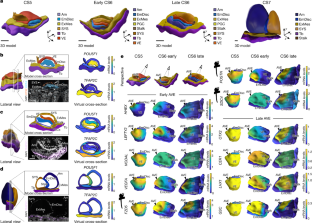
扩展数据图2绒猴胚胎发育阶段。
a,基于所列特征的绒猴胚胎分期允许我们分期囊胚(卡内基分期(CS) 3), CS5, CS6早期和晚期,以及CS7胚胎。中间图:各阶段胚胎模型的图解剖面图,各阶段在(1)次级卵黄囊、(2)Am、(3)茎部和(4)ExMes形成方面存在差异。底部面板:人类胚胎在相应卡内基阶段的代表性图像。CS3转载自74, CS5, CS6晚,CS7从47,和CS6早期从75.b,基于全转录组(>2万个基因)分期整合的绒猴CS5-7发育的主成分分析。c,绒猴胚胎切片的免疫荧光染色。比例尺代表100µm。EmDisc,胚胎盘;点,羊膜;SYS,次级卵黄囊;VE,内脏内胚层;结核病,滋养层;ExMes,胚胎外中胚层; PGCs, Primordial Germ Cells.
扩展数据图3小鼠和绒猴胚胎空间转录组的高斯过程回归模型。
a,基于已发表的小鼠胚胎前/后(a /P)外胚层(Epi)、原始条纹(PS)和前原始条纹(APS)代表性基因标记的表达数据,对小鼠植后胚胎进行空间建模。玉米样地上的每粒玉米都代表了参考文献中小鼠胚胎的显微解剖和测序部分。4,5.将玉米样地转化为空间模型,以匹配前后轴和左右轴。高斯过程回归可以可视化基因表达梯度,并将其与小鼠胚胎植入后的原位杂交进行比较,以验证高斯过程回归方法。在单个图像旁边标明来源出版物。b,镜头在三维虚拟重建上的投影。lcm样本在每个发育阶段的三维虚拟模型上投影为黑点,用于下游基因表达分析。EmDisc,胚胎盘;VE,内脏内胚层;ExMes,胚胎外中胚层;一个前; P, Posterior. c-d, Spatial embryo profiling LCM-sample lineage assignment examples. LCM-samples that were spatially close were assigned to amnion or ExMes based on PDGFRA expression. PDGFRA immunostaining of the CS6 early embryo demonstrates that amnion (nuclei adjacent to the amniotic cavity) is PDGFRA-negative (highlighted in inset below). RawPDGFRA覆盖在DAPI上的lcm样本特征计数与免疫染色模式相似,ExMes样本高表达,羊膜样本无表达。来自其他血统的lcm样本,显示混合血统身份,或未通过QC不显示。箭头指示pdgfr阴性羊膜。3d模型的横截面(右)代表了基于高斯过程回归的CS6早期胚胎中所有谱系的建模,概括了特定的PDGFRA通过免疫荧光和原始转录组数据观察表达模式。e-h,空间胚胎分析处理示例。SOX2 (e)和OTX2 (g)的空间胚胎分析和免疫荧光染色处理的CS6切片的代表性例子。基于高斯过程回归的SOX2 EmDisc和VE建模总结了免疫荧光和原始转录组数据观察到的EmDisc前面的表达模式(f),总结了OTX2的前面表达总结了免疫荧光和原始转录组数据观察到的VE前面的表达模式(h)。模型中基因表达的相对mRNA水平。下面板:mRNA表达沿前后轴的变化(虚线,EmDisc前(红色,A)到EmDisc后(蓝色,P), VE前(黄色,A)到VE后(绿色,P))沿A-P轴的变化,用贝叶斯因子(BF)量化。
图4绒猴原肠形成的表达梯度和信号环境。
a,基于高斯过程回归的CS5-7 EmDisc和VE三维模型中描述的后验标记。上面板:整个模型的相对mRNA水平。下面板:沿EmDisc前后轴mRNA表达变化(虚线表示;前(红色,A)到后(蓝色,P)),用贝叶斯因子(BF)量化(见图。3).b,在CS5-7模型横截面中描述的BMP信号相关基因表达。图(下)总结了CS5和cs6中EmDisc边界羊膜分化背景下的BMP信号通路。c,模型横截面中CS5-7中显示的WNT信号基因。原理图总结了原肠形成过程中CS6 EmDisc中的WNT信号模式。d, rtk相关基因在CS5-7模型横截面上的表达显示VE是主要的来源IGF1,低表达的FGFs参与小鼠原肠形成(FGF8,FGF5),以及存在FGF4和细胞内fgf (FGF12,FGF13).e,在CS5和6个三维模型中EmDisc和VE中rtk相关基因的表达。图中概述了CS6胚胎中的PDGFA和VEGFA。f,模型横截面中CS5-7所示IHH信号相关基因表达。示意图总结了提出的旁分泌IHH信号通路。g,在CS5-7 EmDisc和VE 3D模型中描述的代表性前多能性基因(与图5 - 7 EmDisc和VE 3D模型相关)。3 e).h,在E6.0、E6.5和E7.0的原肠发育小鼠胚胎中匹配多能基因的玉米图5.每个核代表小鼠胚胎的微解剖、空间定义部分的平均转录组。3 c).i,早期(NANOG,PRDM1,POU5F1,KLF4)和晚(DAZL,MAEL,PRAME) PGC标记和富集信号成分(FGF4,WNT8A)在PGCs中的表达,在CS6模型横截面中描述。EmDisc,胚胎盘;SYS,次级卵黄囊;VE,内脏内胚层;ExMes,胚胎外中胚层;点,羊膜;结核病,滋养层。
扩展数据图5羊膜与胚胎盘分离。
a, EmDisc和Am基于前5000个最易变基因的PCA, PC1 = 20.7%, PC2 = 13.6%。b, Marker表达描绘了EmDisc和Am的发散。EmDisc和Am中富集的基因分别用蓝色和紫色标记;着床前基因用绿色表示。流图轨道宽度缩放到相对表达式标准化到所有阶段显示的平均值。c, (a, b)所示胚胎谱系和胚胎外谱系中差异表达基因(DEG)表达的热图。左图为代表性基因(左图)和关键基因本体(GO)富集分析(右图)。Seurat函数热图显示的基因FindAllMarkers(最小百分比50%,最小对数折叠变化0.25),并通过调整后的p值< 0.05进行过滤。d - i, CS5、6和7处3d转录组的虚拟横切面,描绘了(d)新生Am中多能因子表达的代表性基因的mRNA水平,(e) Am- tb共享基因,(f) Am- exmes共享基因,(g) Am特异性基因,(h)上皮基因,(i) ecm相关基因。类别显示在每个面板的左边。EmDisc,胚胎盘;,羊膜。
扩展数据图6:绒猴2D-gastruloids的后路模式需要内源性WNT。
a - c,在第2天(a)或第4天(b)通过免疫染色检测到绒猴2D-gastruloids中pSMAD1/5(磷酸化的SMAD1/5)的活性,与在常规条件下的人类2D-gastruloids相比,在第2天(c)−1FGF + 20 ng ml−1激活素A),常规胃肠道条件(10 ng ml−1FGF + 20 ng ml−1激活素A + 50 ng ml−1BMP4)或WNT调节条件(10 ng ml−1FGF + 20 ng ml−1激活素A + 3 μ M IWP-2或10 ng ml−1FGF + 20 ng ml−1激活素A + 3 μ M CHIR99021)。具有代表性的免疫荧光图像最大投影(左)。定量图(右)在2个井中至少10个类原肠菌的平均+/−SEM。F/A = FGF/激活素A. pSMAD1/5梯度表明,在绒猴系统中,与人类系统类似,FGF/ActivinA/BMP4在第2天诱导对BMP信号的分级反应,在群体的最外层环中信号最强。d, 2D-gastruloids的分子特征。典型的原肠样免疫荧光图像在常规原肠样条件下分化(10 ng ml−1FGF + 20 ng ml−1激活素A + 50 ng ml−1BMP4) 2天。定量图(底部)显示了2个实验中至少10个类原肠杆菌的平均+/−SEM。前域(A, SOX2+, TBXT−, TFAP2C−),后域(P, TBXT高,CDX2异质,SOX17稀疏,LEF1稀疏),羊膜域(Am, TFAP2C高,SOX2−)划定。ISL1和TFAP2A主要分布在羊膜区。PDGFRA表达低,表明缺乏成熟的中胚层。e, 2D-gastruloids中存在的谱系特征示意图和定义每个区域的标记模式。f, 2D-gastruloids的wnt相关前/后模式表型。代表性的类原肠杆菌免疫荧光图像在左侧所列条件下分化2天。定量图(底部)在2个井中至少10个类原肠菌的平均+/−SEM。g, 2D-gastruloids的wnt相关羊膜发生表型。代表性的类原肠杆菌免疫荧光图像在左侧所列条件下分化2天。经3 μM IWP2处理后,CDX2的表达减少,TFAP2C和TFAP2A仍有表达。 3 μM CHIR treatment (WNT agonist) leads to low CDX2 expression but does not support upregulation of TFAP2C or TFAP2A. Quantification plots (bottom) mean +/− SEM across a minimum of 10 gastruloids across 2 wells. h, No evident change in expression profiles associated with Hedgehog signalling manipulation in 2D-gastruloids. Representative immunofluorescence images of gastruloids differentiated in conditions listed at left for 2 days. Exogenous Indian Hedgehog (IHH, 200 ng ml−1)不会导致FGF/激活素自我更新条件下多能性的丧失,也不会改变传统类原肠菌素条件下的表达模式。环巴胺(5 μM)抑制Hedgehog信号也未导致表达模式发生明显变化。定量图(底部)在2个井中至少10个类原肠菌的平均+/−SEM。
图7绒猴2D-gastruloids胚胎模式调控基因的功能分析。
a, siRNA敲除效率。代表性的免疫荧光图像(左)和转染siRNA后24小时的平均荧光强度定量(右)POU5F1,SOX2,或NANOG.与siGFP(绿色荧光蛋白)对照进行双尾Mann-Whitney检验(****,p < 0.0001)。siRNA:小干扰RNA。b, siRNA筛选方法示意图。cmPSCs(普通绒猴多能干细胞)于第1天在96孔微图版板中播种,用siRNA转染过夜。第0天,培养基改为原肠样诱导培养基(10 ng ml−1FGF + 20 ng ml−1激活素A + 50 ng ml−1BMP4)。72 h后固定2d -原肠样,染色以评估模式形成。c, siGFP表型与体内EmDisc模式的比较。sigfp处理的原肠样在常规原肠样条件下分化的代表性最大投影免疫荧光图像(10 ng ml−1FGF + 20 ng ml−1激活素A + 50 ng ml−1BMP4) 3天(左)。将单个通道(SOX2, TBXT, TFAP2C)归一化至最大强度的2d -原肠样对数表达模式与通过前后轴表达高斯过程回归生成的CS6 EmDisc表达模式虚拟胚胎模式并排绘制。定量图(右)显示至少10个前域类原肠腺的平均+/−扫描电镜(a, SOX2+, TBXT−, TFAP2C−),后域(P, TBXT高),羊膜域(Am, TFAP2C高,SOX2−)划定。d-f。siRNA敲低多能因子表型(siPOU5F1,siNANOG,siSOX2)、bmp相关基因(siID1/2/3,siTBX3)和wnt相关基因(siSFRP1,siSFRP2,siSFRP1/2).对于每个siRNA,有代表性的最大投影免疫荧光显示(左)。在每个原肠样体的每个标记物的细胞核阳性百分比(中心)附近,绘制单个通道(SOX2, TBXT, TFAP2C)的代表性表达模式。采用双尾Mann-Whitney检验进行比较(ns:不显著;P < 0.0332 (*);P < 0.0021 (**);p < 0.0002,(***);p < 0.1;P < 0.0001(****))。定量图(右)显示了2个井中至少10个类原肠菌的平均+/−SEM。 SOX2 = green, TFAP2C = red, TBXT = grey. siGFP patterns plotted for comparison in reduced opacity and dashed line with control anterior domain (A, SOX2+, TBXT−, TFAP2C−),后域(P, TBXT高),羊膜域(Am, TFAP2C高,SOX2−)划定。cmPSCs,普通绒猴PSCs。
扩展数据图8绒猴胚胎盘三维培养模型在体外.
a,间期培养概述示意图。为了在3D培养中模拟传统cmPSCs(普通绒猴多能干细胞)的自组织,cmPSCs被播种在100%的Matrigel碱基上,上面覆盖1%的Matrigel溶解在补充了信号因子的N2B27培养基中。间期培养可满足诱导信号的要求,促进胚前盘样多能性或向原肠胚胚层分化。所有实验均在两种不同的细胞系(细胞系1 (New4f), N = 2和细胞系2 (New2f), N = 2)中进行,结果一致。b,用于间期培养结构分子表征的标记基因热图。所有绒猴的相对mRNA水平均以中心和比例分布在活的有机体内样本。c,绒猴胚胎中节点和FGF信号传递的概要示意图。内脏内胚层是其主要来源节点而且IGF1在绒猴胚胎中,EmDisc表达低水平的FGF4.CS6横截面中总结的相对mRNA表达梯度。d, FGF间期培养的时间序列亮场图像(100 ng ml−1)和激活素A (20 ng ml−1).FGF/激活素A培养提供了一个信号环境,模拟来自VE的高node和IGF1。结构形成柱状上皮囊肿,使人联想到胚胎盘。结构首先在第3天打开腔,并扩展到第6天。e-f,第4天emdisc样结构的分子特征。第4天免疫染色的代表性最大投影图像,用于多能性(SOX2)、早期原肠形成(TBXT)、羊膜(TFAP2C, TFAP2A)、内胚层(SOX17)或中胚层(CDX2)标记。emdisc样结构均质表达SOX2, TBXT、SOX17和TFAP2C均质低表达,提示原肠胚形成启动,内胚层罕见出现。多能性EmDisc样结构支持FGF和激活素/节点信号通路在促进EmDisc多能性中的作用。g,绒猴胚胎中典型WNT信号的概述。EmDisc后部、茎部和PGCs表达WNT3.CS6横截面(左)总结了mRNA表达梯度。FGF和激活素A + CHIR (CHIR99021, WNT激动剂)间期培养的时间序列亮场图像(右)。第4天分化群体出现明显。h-i,第4天wnt处理的emdisc样结构的分子特征。第4天免疫染色的代表性最大投影图像,包括多能性(SOX2)、早期原肠形成(TBXT)、羊膜(TFAP2C、TFAP2A)、内胚层(SOX17)或中胚层(CDX2)标记。与FGF/Activin A培养相比,其结构表现出SOX2表达缺失,TBXT和SOX17表达上调,与羊膜、内胚层和中胚层种群分化一致。j,绒猴胚胎中WNT的抑制作用概述。VE和羊膜表达典型的WNT抑制剂DKK1.CS6横截面(左)总结了mRNA表达梯度。FGF和激活素A + IWP-2间期培养的时间序列亮场图像(右)。与FGF/激活素A培养相似,结构首先在第3天打开一个腔,并扩展到第6天。k-l,第4天wnt抑制emdisc样结构的分子特征。第4天免疫染色的代表性最大投影图像,包括多能性(SOX2)、早期原肠形成(TBXT)、羊膜(TFAP2C、TFAP2A)、内胚层(SOX17)或中胚层(CDX2)标记。与FGF/Activin A培养相比,EmDisc样结构均质表达SOX2, TBXT和SOX17下调,与WNT抑制在EmDisc中保持多能性的作用一致。m,绒猴胚胎中BMP信号传递的概要示意图。ExMes,羊膜,和PGCs是来源BMP4在胚胎中。CS6横截面(左)总结了mRNA表达梯度。FGF和激活素A + BMP4间期培养的时间序列亮场图像(右)。第4天出现了明显的无组织、分化的种群。n-o,第4天bmp处理的emdisc样结构的分子特征。第4天免疫染色的代表性最大投影图像,包括多能性(SOX2)、早期原肠形成(TBXT)、羊膜(TFAP2C、TFAP2A)、内胚层(SOX17)或中胚层(CDX2)标记。与FGF/Activin A培养相比,结构表现出SOX2表达缺失,TFAP2C、TFAP2A、CDX2和SOX17表达上调,与混合羊膜和后分化的原始条状结构一致。p,绒猴胚胎中BMP抑制的概要示意图。VE表达BMP抑制剂‘诺金’在胚胎中。CS6横截面(左)总结了mRNA表达梯度。FGF和激活素A + BMP4间期培养的时间序列亮场图像(右)。与FGF/Activin A培养相似,结构在第3天首先打开一个腔,形成均匀的球体。q-r,第4天bmp抑制emdisc样结构的分子特征。第4天免疫染色的代表性最大投影图像,包括多能性(SOX2)、早期原肠形成(TBXT)、羊膜(TFAP2C、TFAP2A)、内胚层(SOX17)或中胚层(CDX2)标记。比例尺代表100µm。与FGF/Activin A培养相比,emdisc样结构均表达SOX2, TBXT、SOX17和TFAP2C下调。这与BMP抑制在EmDisc中保持多能性和抑制羊膜分化中的作用一致。cmPSCs,普通绒猴PSCs。
图9灵长类动物中胚层分化的跨种分析在活的有机体内.
a,绒猴和人类emdisc衍生的植后谱系的比对。基于胚胎对齐的可视化在活的有机体内而且在体外食蟹猴植前及植后数据集25,在体外培养人的12,在活的有机体内人类CS79,植入前绒猴(参考文献;24本研究),和移植后绒猴胚胎数据(本研究)是一致的。统一流形近似和投影降维。b,绒猴CS5-7或人类晚期CS7谱系的UMAP图显示来自标记基因的归一化对数表达9.多能性EmDisc(胚胎椎间盘)SOX2,背景;原条:背景,置,TBXT;新生中胚层:TBXT,CDH2,新兴中胚层:OTX2,LHX1;先进的中胚层:FOXF1,HAND1,BMP4,GATA6;羊膜:VTCN1,HAND1,BMP4;内胚层亚簇也显示为共表达CDH2,OTX2,GATA6.c, (a)中UMAP所代表的原肠成形期谱系的无偏聚类通过共享最近邻聚类解决了9个聚类:多能性EmDisc(胚胎盘)、PS early(原始条纹早期)、PS late(原始条纹晚期)、PS advanced(原始条纹晚期)、Endoderm、Nasc Mes(新生中胚层)、Em Mes(新生中胚层)、Adv Mes(高级中胚层)、PGCs(原始生殖细胞)。d,内胚层和中胚层从扩散图中表示的原始条纹的分叉。人类CS7显示了从原始条纹到高级中胚层再到突现中胚层的额外路径。对齐包括(c)中绘制的数据,其中不包括羊膜和PGC簇,其中簇标识由颜色给出。e,绒猴CS5-7或人类晚期CS7谱系的扩散图显示原始条纹标记基因的归一化对数表达(TBXT)、内胚层(FOXA2)、泛中胚层(SNAI2,CDH2)、新兴中胚层(OTX2)或新生中胚层(FOXF1).f - g,人类与绒猴原始条纹与新生中胚层的散点图(f)或新生中胚层与内胚层的散点图(g)。高亮象限表示每个谱系的人绒猴保守标记,而白色象限表示种特异性表达模式。转录因子、配体和细胞外基质分子的基因名称被标记。
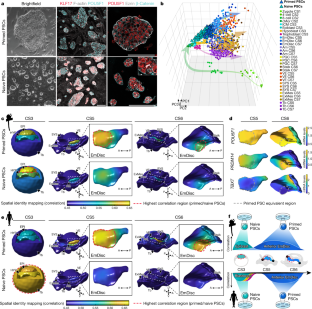
图10人的空间身份映射在体外模型。
a,幼稚和启动的人类和绒猴PSCs与在活的有机体内绒猴的数据。绒猴在活的有机体内植前和植后发育数据集,幼稚/引物的PSCs和人类幼稚/引物的PSCs43是一致的。通过主成分分析,对齐数据集的可视化显示了植入前(左侧)和植入后(右侧)样本的分离,绒猴PSCs位于植入前和植入后之间。用透明绒猴胚胎数据绘制人类PSCs。初代人类细胞比初代绒猴细胞更早地映射到PC1上,并且在映射到ICM、Hypoblast和Tb方面表现出更大的异质性。启动的人类细胞与绒猴EmDisc紧密相连。主成分分析;多能干细胞。ICM,内细胞团;忧郁,内胚层;SYS,次级卵黄囊; VE, Visceral Endoderm; ExMes, Extraembryonic Mesoderm; EmDisc, Embryonic Disc; Am, Amnion; Tb, Trophoblast. b, Expression of extraembryonic markers in marmoset在活的有机体内数据(左)和人类原始和引物PSCs(右)在(a)中对齐的pca中表示,显示滋养层标记的综合日志表达(JAM2)、内胚层标记物(加工,GATA6)和ICM/着床前囊胚标记物(ESRRB).在PCA上,绿松石线表示原始细胞的位置,蓝色线表示引物细胞的位置。c,人类微流体胚胎样囊模型与绒猴STEP数据的比对。绒猴的Smart-seq2剖析在活的有机体内世系(EmDisc、PGC、Amnion和Stalk群体)与微流控胚胎样囊模型的10x测序分析相一致16.主成分分析显示了hPSC、中胚层、PGCLCs和amlc的分离。主成分分析;PSC,人多能干细胞;hPGCLC,人原始生殖细胞样细胞(hAMLC);人羊膜样细胞;hMELC1/2,人中胚层样细胞群1或2。d, (c)中UMAP表示的无偏聚类通过共享最近邻聚类在绒猴数据中解析5个聚类。4个簇与人类微流体胚胎样囊模型的亚群一致,并被注释为EmDisc, PGC, Amnion和Mesoderm。绒猴数据中的第五个亚簇包含来自原肠形成的EmDisc、PGCs和羊膜的细胞,因此被标记为ps样(原始条纹样)。 e, Pearson’s correlation of marmoset and human microfluidic embryonic-like sac model of clusters identified in (d). f–h. Spatial identity mapping of human microfluidic embryonic-like sac model. Subpopulations of the human microfluidic embryonic-like sac model were mapped to the marmoset EmDisc, PGC, Stalk, and Amnion. Spatial identity displayed in the orientations described in (f) for hPS Cs (g), which mapped to the anterior EmDisc and hAMLCs (h) which mapped most strongly to the posterior amnion. cmPSCs, common marmoset PSCs; hPSCs, human PSCs.
补充信息
补充信息
补充图1-12
补充表1
转录组样本概述。Smart-seq2在当前研究中分析的与Ext相关的样本列表和描述。
补充表2
绒猴谱系和体外细胞的平均基因表达。所有样本的平均基因表达量(每10,000计数)。以首字母缩写标记的样品类型;完整的示例说明可在补充表1中找到。
补充表3
前、后基因。EmDisc在CS5、CS6和CS7位点的前、后隔室或在CS5和CS6位点的VE位点富集的基因。贝叶斯因子,δ:前后基因表达的差异。基因平均表达量(万分之数)转载自补充表2。
补充视频1
虚拟胚胎重建综述,合子到原肠胚形成。传统单细胞测序的胚胎植入前阶段和STEP 3d重建胚胎植入后阶段的虚拟概述。
补充视频2
卡内基第五阶段胚胎的虚拟重建。流水线的动画概述谱系分割,虚拟堆叠,表面生成和3D模型重建。完整的3D模型显示卡内基阶段5的特征,包括一个新生的羊膜腔,一个厚的胚胎盘和一个小的次级卵黄囊从内脏内胚层突出。代表性的免疫荧光染色为DAPI(白色)、POU5F1(绿色)和TFAP2C(红色)。
补充视频3
卡内基阶段6(晚期)胚胎的虚拟重建。流水线的动画概述谱系分割,虚拟堆叠,表面生成和3D模型重建。完整的3D模型显示了标志性的卡内基阶段6的特征,包括一个扩大的羊膜腔,次级卵黄囊腔的出现,围绕胚胎的胚外中胚层,以及通过连接胚胎后盘和滋养层的柄形成的原始条纹形成的证据。DAPI(蓝色)、PDGFRA(红色)、SOX2(绿色)和OTX2(白色)的代表性免疫荧光染色。
补充视频4
卡内基7期胚胎的虚拟重建。流水线的动画概述谱系分割,虚拟堆叠,表面生成和3D模型重建。完整的3D模型展示了卡内基阶段7的特征,包括大量扩大的羊膜和次级卵黄囊腔和胚胎盘的旋转垂直于间充质柄。代表性的免疫荧光染色为DAPI(蓝色)和TFAP2C(红色)。
源数据
权利和权限
根据与作者或其他权利持有人签订的出版协议,《自然》杂志或其许可方对本文拥有独家权利;作者对这篇文章接受的手稿版本的自我存档仅受此类出版协议的条款和适用法律的约束。
关于本文
引用本文
伯格曼,C.A.彭福尔德,E.斯拉特里。et al。早期灵长类动物子宫内原肠形成的空间特征。自然609, 136-143(2022)。https://doi.org/10.1038/s41586-022-04953-1
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586-022-04953-1
这篇文章被引用
绒猴胚胎空间基因活性分析
细胞研究(2022)
哺乳动物胚胎发生的六种转录组图谱描述了三种不同植入模式的代谢调节
自然通讯(2022)
人类生殖系干细胞
信号转导与靶向治疗(2022)
德国与麻省理工学院:德国与安文东
BIOspektrum(2022)
