摘要gydF4y2Ba
Holliday连接是在所有生命王国的DNA重组过程中形成的一个关键中间体gydF4y2Ba1克ydF4y2Ba.在细菌中,Holliday结由两个同型六聚体AAA+ atp酶RuvB马达处理,它们与RuvA-Holliday结复合体组装在一起,激发链交换反应gydF4y2Ba2gydF4y2Ba.尽管该复合体对染色体的维持很重要,但其促进分支迁移的结构和机制尚不清楚。在这里,使用时间分辨冷冻电子显微镜,我们获得了atp水解RuvAB复合体在7种不同构象状态下的结构,在组装和加工Holliday结过程中捕捉到。5个结构共同解析了完整的核苷酸周期,揭示了ATP水解、核苷酸交换和RuvB中特定构象变化之间的时空关系。由dna脱离的RuvB亚基形成的转换体中的协同运动刺激水解和核苷酸交换。转换器的固定化使RuvB能够将包含atp的能量转换为杠杆运动,从而产生驱动分支迁移的拉力。我们发现,RuvB马达与DNA底物一起旋转,这与一个进展的核苷酸循环一起,形成了通过持续分支迁移的DNA重组的机制基础。我们的数据解读了RuvAB配合物同源重组的分子原理,阐明了六聚体AAA+电机化学-机械耦合的离散和顺序过渡态中间体,并为针对AAA+电机的状态特异性化合物的设计提供了蓝图。gydF4y2Ba
主要gydF4y2Ba
同源重组是一种基本的细胞过程,涉及到遗传完整性的维护和遗传多样性的产生,跨越生命的所有领域。在基因重组、双链断裂修复和复制叉挽救过程中,中心和普遍的元素是一个被称为Holliday结的四向DNA异质双工gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba3 gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba.在原核生物中,RuvA和RuvB这两种蛋白质通过促进atp依赖性的单向链交换反应,即活性分支迁移,在Holliday连接的处理过程中发挥着关键作用gydF4y2Ba2gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10克ydF4y2Ba,gydF4y2Ba11gydF4y2Ba.先前的生物化学和结构证据表明,分支迁移是由一个三方复合体促进的:RuvA四聚体聚集在Holliday结交叉周围,为DNA分离和复卷提供结构指导,两侧是两个六聚体RuvB AAA+ atp酶,共同促进新出现的重组DNA的易位gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba.此外,这些研究表明,RuvA (RuvAgydF4y2BaD3gydF4y2Ba)结合到RuvB的显着素-1 β-发夹上,这是PS1插入超枝的一个显著特征gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba调节分支迁移,增加RuvB马达的atp酶活性gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba.此外,这种跨物种的异互补能力证实了RuvA-和RuvB AAA+协同作用机制在Holliday结合部的存在gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba.尽管有大量的知识,但RuvAB-Holliday连接复合体(以下简称RuvAB-HJ)的结构和RuvB AAA+马达驱动DNA易位的分子机制促进了生物体内最基本的生物过程之一,即遗传信息的维护和交换gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba仍然是未知的。为了揭示RuvAB机械的结构和工作原理,我们应用时间分辨低温电子显微镜(cryo-EM)和单粒子分析体外重组RuvAB配合物处理Holliday结。我们的结构分析揭示了一个活跃的RuvAB分支迁移复合体高度协调的构象景观,并揭示了旋转RuvB AAA+马达中完全分解的核苷酸循环和DNA转位之间的动态相互作用。此外,我们还发现,在atp依赖的功率冲程中,RuvB马达将DNA作为分子杠杆进行转移,将化学能转换为机械力。gydF4y2Ba
RuvAB-HJ配合物的结构gydF4y2Ba
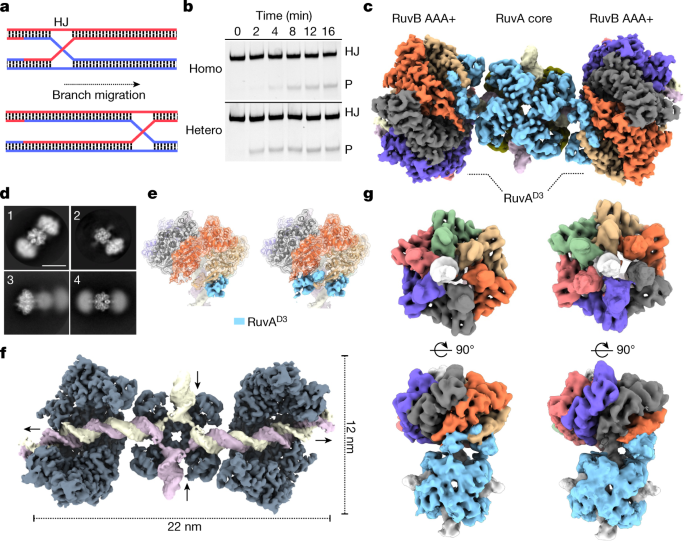
在DNA重组过程中,由RuvAB机制驱动的Holliday连接的分支迁移是一个快速和高度动态的过程gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba(无花果。gydF4y2Ba1gydF4y2Ba).为了使这一过程可视化,我们从来自于gydF4y2Ba鼠伤寒沙门氏菌gydF4y2Ba而且gydF4y2Ba乳酸链球菌gydF4y2Ba,并在分支迁移实验中测试了它们的功能(图。gydF4y2Ba1 bgydF4y2Ba).两者homo- (RuvA和RuvB fromgydF4y2Ba美国沙门氏菌感染gydF4y2Ba)和hetero- (RuvA fromgydF4y2Ba美国沙门氏菌感染gydF4y2Ba和RuvBgydF4y2Ba美国酸奶gydF4y2Ba)复合物在加入ATP后对Holliday结的处理类似,这表明由于单个组分的互换性,其潜在机制高度保守(图。gydF4y2Ba1 bgydF4y2Ba和扩展数据图。gydF4y2Ba1 a -gydF4y2Ba).为了捕捉这个快速过程的催化步骤,我们首先用等摩尔的缓慢水解的ATPγ s混合物取代ATP来减慢反应速度gydF4y2Ba30.gydF4y2Ba和ADP,并在冰上孵育反应30分钟(数据集t1)或5小时(数据集t2),以模拟RuvAB-HJ复合物的起始和平衡阶段(扩展数据图。gydF4y2Ba1 hgydF4y2Ba).随后的样品玻璃化导致了同质配合物的聚集和少量的单个颗粒,而异质配合物在网格上的分布很大程度上是单分散的,适合单颗粒分析(扩展数据图。gydF4y2Ba1 f jgydF4y2Ba).RuvAB-HJ配合物的低温- em结构分辨率为8 Å,显示出高度灵活和线性排列的三方组合,8个RuvA分子对称排列在两个四聚体(3.3 Å分辨率)和四向Holliday结两侧,并与1或2个RuvB六聚体(2.9-4.1 Å分辨率)和二聚体粒子(3.9 Å分辨率)灵活连接(图4)。gydF4y2Ba1汉英gydF4y2Ba和扩展数据图。gydF4y2Ba2 a - cgydF4y2Ba,gydF4y2Ba3模拟gydF4y2Ba而且gydF4y2Ba4 a - bgydF4y2Ba和扩展数据表gydF4y2Ba1克ydF4y2Ba).该体系结构与先前提出的RuvAB机制模型一致gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba31gydF4y2Ba.在这两种粒子类型中,DNA以双螺旋结构进入和离开RuvA核心,一个或两个六聚体RuvB马达与重新连接的DNA的小凹槽接合(图。gydF4y2Ba1 fgydF4y2Ba).RuvA核心通过RuvA物理连接到两个RuvB电机gydF4y2BaD3gydF4y2Ba(无花果。gydF4y2Ba1 cgydF4y2Ba).两边各有两个RuvAgydF4y2BaD3gydF4y2Ba结构域与相邻位置的RuvB子单元相结合,表明这些结构域能够协同控制两个RuvB AAA+电机(图1)。gydF4y2Ba1 c, egydF4y2Ba).值得注意的是,所有四个ruvb协调的RuvAgydF4y2BaD3gydF4y2Ba域本地化到Holliday结交叉的同一侧(扩展数据图。gydF4y2Ba4 fgydF4y2Ba),意味着一个单一的RuvA四聚体可能足以同时操作两个RuvB电机。这些发现也与拟议的RuvABC解析体结构一致,其中Holliday结被一个RuvA四聚体和一个解析体RuvC的二聚体夹在中间gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba(扩展数据图。gydF4y2Ba4 ggydF4y2Ba).gydF4y2Ba
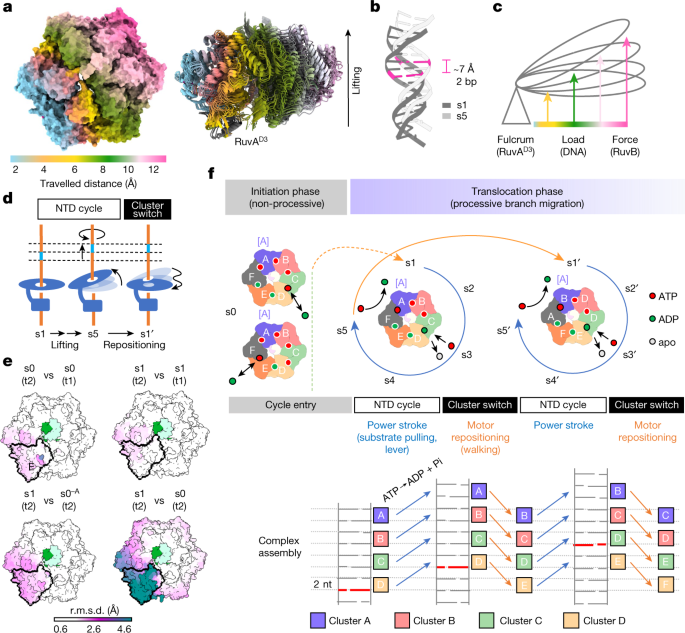
一个gydF4y2Ba, Holliday结分支迁移示意图。沪江,霍利迪结。gydF4y2BabgydF4y2Ba, RuvAB同源和杂合物在分支迁移中是活跃的。荧光标记Holliday结(8 nM)重组蛋白的活性比较gydF4y2Ba美国沙门氏菌感染gydF4y2BaRuvA (60 nM)和重组RuvBgydF4y2Ba美国沙门氏菌感染gydF4y2Ba(160 nM) (homo)或gydF4y2Ba美国酸奶gydF4y2Ba(160海里)(异性恋)。实验重复了三次。P,产品。gydF4y2BacgydF4y2Ba,结合到Holliday结的RuvAB复合体(分子质量约650 kDa)的Cryo-EM复合图。RuvA:RuvB的绝对化学计量是8:12。两个RuvA四聚体(浅蓝色(前)和橄榄色(后))夹在Holliday连接处。c端RuvAgydF4y2BaD3gydF4y2Ba域从中心核心延伸并绑定到RuvB电机。gydF4y2BadgydF4y2Ba, RuvAB-HJ粒子具有高度的灵活性。三部(1)和二部(2)粒子的代表性2D类。聚焦于RuvB电机(3)或中央RuvA-HJ核心(4)的分类突出了三方粒子的整体灵活性。比例尺,10nm。gydF4y2BaegydF4y2Ba, RuvB电机绑定到一个或两个RuvAgydF4y2BaD3gydF4y2Ba域(蓝色)。这两个RuvAgydF4y2BaD3gydF4y2Ba域绑定到RuvB电机中相邻的RuvB子单元。gydF4y2BafgydF4y2Ba, RuvAB-HJ复合体中分离的RuvB亚基和一个RuvA四聚体被移除,以可视化Holliday结和每个RuvB马达与其同源DNA底物的相互作用。箭头显示了进入RuvA核心的DNA的运动方向和从RuvB马达的新DNA双工的出口。复合体的尺寸已标明。gydF4y2BaggydF4y2Ba,相对于RuvA核心旋转60°的RuvB六聚体的配置。聚焦3D类(端到(上面板)和侧视图(下面板)使用一个包含一个RuvB电机和中央RuvA核心的掩模。RuvA互动gydF4y2BaD3gydF4y2Ba结构域和构象特异性RuvB亚基相对于RuvA-HJ核旋转60°。gydF4y2Ba
为了研究RuvAB-HJ配合物的柔韧性,我们对我们的粒子进行了进一步的三维分类。该分析表明,除了整体灵活性外,在约7%的二部粒子和约6%的三部粒子中,dna参与的RuvB相对于RuvA-HJ核心的位置旋转了约60°。这表明RuvB电机能够旋转,并且在完成60°旋转后,每个RuvB子单元都占据了旋转前相邻子单元的位置(图1)。gydF4y2Ba1克gydF4y2Ba,扩展数据图。gydF4y2Ba5 a、bgydF4y2Ba和补充视频gydF4y2Ba1克ydF4y2Ba).多体精细化分析进一步证明了旋转在粒子中占总柔性的45%左右(扩展数据图)。gydF4y2Ba5汉英gydF4y2Ba和补充视频gydF4y2Ba2gydF4y2Ba).因此,我们推断重组的RuvAB复合体具有酶活性,因此在不同的构象状态下成像。此外,我们的数据显示,之前描述的DNA底物的连续旋转gydF4y2Ba34gydF4y2Ba伴随着RuvB AAA+电机本身的旋转。gydF4y2Ba
RuvB电机的构象景观gydF4y2Ba
为了理解RuvB电机的旋转与分支迁移之间的联系,我们对来自t2数据集的RuvB六聚体应用了迭代聚焦细化和严格的三维分类。该分析揭示了9个结构上不同的RuvB马达映射,分辨率在2.9到4.1之间Å(扩展数据图。gydF4y2Ba2 c, egydF4y2Ba而且gydF4y2Ba3 e-ngydF4y2Ba和扩展数据表gydF4y2Ba1克ydF4y2Ba).其中两个映射(在3.9和4.1 Å分辨率)无法改进到允许明确分配核苷酸的分辨率,因此没有进一步考虑。其余7个RuvB电机可以根据绑定RuvA的数量进行分组gydF4y2BaD3gydF4y2Ba,有一张地图缺少RuvAgydF4y2BaD3gydF4y2Ba(s0gydF4y2Ba−一gydF4y2Ba),两张包含一个RuvA的地图gydF4y2BaD3gydF4y2Ba(s0和s1)和四张显示两界RuvA的地图gydF4y2BaD3gydF4y2Ba域(s2, s3, s4和s5),一起表明RuvA之间的动态相互作用gydF4y2BaD3gydF4y2Ba和RuvB电机。gydF4y2Ba
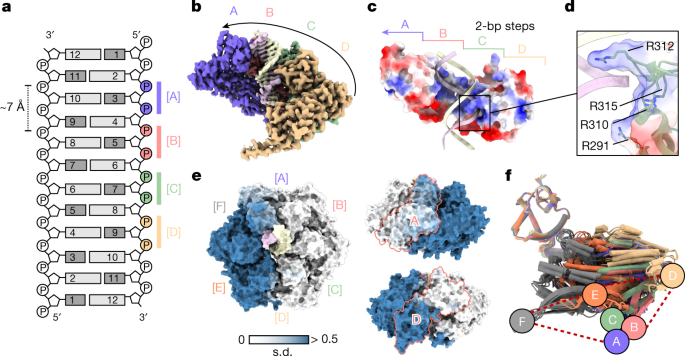
所有的RuvB马达组装成封闭的,不对称的六聚体,具有大约2纳米宽的中央孔,容纳DNA(扩展数据图。gydF4y2Ba3 e mgydF4y2Ba).与之前的结构和相互作用研究一致,RuvB的寡聚是由大的(RuvBgydF4y2BalgydF4y2Ba)和小的(RuvBgydF4y2Ba年代gydF4y2Ba)相邻亚基的atp酶结构域gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba35gydF4y2Ba(扩展数据图。gydF4y2Ba4汉英gydF4y2Ba).类似于其他AAA+易位酶gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,四个RuvB子单元(A, B, C和D)一起组装成一个“螺旋楼梯”。这产生了一个连续的界面,主要结合两个DNA链中的一个(图。gydF4y2Ba2 a, bgydF4y2Ba),强调每个进入RuvA核心的双链DNA中只有一条链是由一个RuvB马达控制的(扩展数据图。gydF4y2Ba4 hgydF4y2Ba).剩下的两个RuvB亚基(E和F)关闭了螺旋阶梯,但不与DNA结合。DNA参与的亚基(A, B, C和D)通过它们的C端头结构域(RuvB)与DNA结合gydF4y2BaHgydF4y2Ba).每个RuvBgydF4y2BaHgydF4y2Ba该结构域包含4个保守的精氨酸残基Arg291、Arg310、Arg312和Arg315,它们产生一个带正电荷的结合界面,与带负电荷的DNA主链互补(图1)。gydF4y2Ba2 c, dgydF4y2Ba).(为了便于比较gydF4y2Ba大肠杆菌gydF4y2BaRuvAB系统中,相应的残基列在补充信息表中gydF4y2Ba1克ydF4y2Ba而且gydF4y2Ba2gydF4y2Ba).来自每个亚基的精氨酸残基的重复结合模式与由两个核苷酸(大约7 Å)的距离隔开的DNA相结合。此外,由于RuvB子单元在RuvB六聚体中彼此相距约60°,这些数据进一步表明,RuvB马达的旋转与一个易位步骤中发生的事件有关。gydF4y2Ba
一个gydF4y2Ba, DNA底物和四个阶梯RuvB亚基(A, B, C和D)之间的界面示意图。亚基沿着沿着DNA轴间隔约7 Å的一条DNA链的磷酸主链与DNA底物相结合(每两个核苷酸)。gydF4y2BabgydF4y2Ba, Cryo-EM图突出显示DNA相互作用的亚基(a, B, C和D)形成的螺旋楼梯(在这个视图中,没有结合DNA的亚基E和F被删除)。gydF4y2BacgydF4y2Ba,由RuvB阶梯(A, B, C和D)形成的dna结合界面头部结构域的表面电荷表示。gydF4y2BadgydF4y2Ba螺旋阶梯形成一个由精氨酸残基(Arg291, Arg310, Arg312和Arg315)组成的从a到D的带正电荷的裂口,与双链DNA的一条链结合。gydF4y2BaegydF4y2Ba, RuvB的表面表征及变异分析。分析将RuvB六聚体分为刚性区(白色)和柔性区(钢蓝色),由边界亚基a和d连接。根据Cα原子距离的标准差上色(原子模型与RuvB亚基C对齐)。gydF4y2BafgydF4y2Ba,从五种六聚RuvB电机态(s1到s5)中提取的30个RuvB亚基的叠加。RuvB的亚基排列在RuvB的头部结构域。彩色标签表示相似的构象(构象簇[A]到[F])。gydF4y2Ba
为了研究六聚体的整体构象可塑性,我们分析了每一个Cα原子在所有七个不同的电机结构上的变异性,表示为与相应质心距离的标准偏差(图。gydF4y2Ba2 egydF4y2Ba).这表明,RuvB六聚体可分为刚性(白色)、柔性(蓝色)和中间区域。刚性区包含与dna结合的亚基B和C,而与dna分离的亚基E和F位于柔性区。值得注意的是,分别连接楼梯顶部和底部两个不等半段的dna结合亚基A和D位于中间区域,这表明六聚体内部的差异灵活性参与了ruvab介导的分支迁移。值得注意的是,变异性的程度并不一定局限于整个RuvB亚基,例如亚基A和D,它们显示出柔性和刚性区域(图。gydF4y2Ba2 egydF4y2Ba).为了进一步评估单个RuvB亚基的可塑性,我们确定了42个RuvB亚基之间的平均均方根偏差(r.m.s.d),以及它们各自的域之间:RuvBgydF4y2BalgydF4y2Ba(残留21 - 181),RuvBgydF4y2Ba年代gydF4y2Ba(残留182-254)和RuvBgydF4y2BaHgydF4y2Ba(残留255 - 330)。这一分析显示低平均r.m.s.d。(r.m.s.d。gydF4y2BaØgydF4y2Ba分别为1.2 Å, 0.48 Å和0.453 Å)。gydF4y2Ba6gydF4y2Ba),显示RuvB的整体结构gydF4y2BalgydF4y2Ba, RuvBgydF4y2Ba年代gydF4y2Ba和RuvBgydF4y2BaHgydF4y2Ba在很大程度上保持不变,但它们彼此之间的相对位置是不同的。gydF4y2Ba2 fgydF4y2Ba和扩展数据图。gydF4y2Ba6 a、bgydF4y2Ba).六聚体内DNA底物的存在进一步使我们能够确定RuvB亚基显示出位置特定的、不同的构象,以下称为簇(簇[A]对应于RuvB中亚基A的位置,簇[B]对应于亚基B的位置,等等)。gydF4y2Ba2 fgydF4y2Ba).gydF4y2Ba
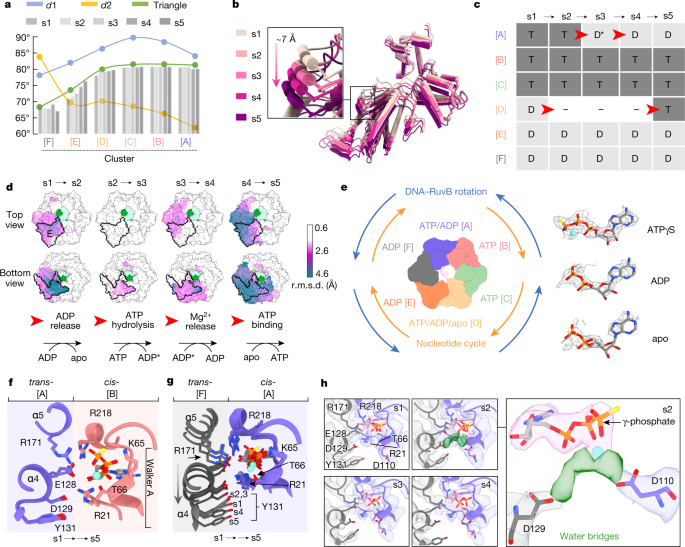
然后,我们通过测量两个二面角(gydF4y2BadgydF4y2Ba1 RuvB之间gydF4y2BalgydF4y2Ba和RuvBgydF4y2Ba年代gydF4y2Ba,gydF4y2BadgydF4y2Ba2 RuvB之间gydF4y2Ba年代gydF4y2Ba和RuvBgydF4y2BaHgydF4y2Ba)和一个三角形的角度,它提供了一个关于RuvB中发生的变化的更全面的视角(扩展数据图。gydF4y2Ba6摄氏度gydF4y2Ba).我们发现每个RuvB簇([A]到[F])的特征是三个角度的独特组合,因此包含一组具有更相似构象的RuvB亚基(图1)。gydF4y2Ba3gydF4y2Ba).RuvB在团簇内也会发生变形,在团簇中变化最大[E],其中三角形角的动态范围为5.6°(图5)。gydF4y2Ba3gydF4y2Ba和扩展数据图。gydF4y2Ba6摄氏度gydF4y2Ba).为了更好地描述RuvB六聚体这一灵活区域的运动,我们将s1到s5的5个结构与几乎不变的亚基C对齐,并分析所有其他亚基的运动(扩展数据图。gydF4y2Ba6 d-fgydF4y2Ba).该方法揭示了簇内连续的构象变化[E]可以沿着一条平均长度约为7 Å(范围:6-10 Å)的轨迹描述,这条轨迹指向RuvA-HJ核(图1)。gydF4y2Ba3 bgydF4y2Ba).值得注意的是,簇内轨迹的长度[E]很好地对应于两个核苷酸的RuvB阶梯的步长(DNA中核苷酸之间的距离约为3.5 Å),这表明五个RuvB结构(s1到s5)可以代表一个活跃的RuvB电机通过一个易位步骤时的连续原子快照。gydF4y2Ba
一个gydF4y2Ba, RuvB亚基从状态s1到s5构象簇的二面体分析(gydF4y2BadgydF4y2Ba1,gydF4y2BadgydF4y2Ba2)和三角形的角度(扩展数据图。gydF4y2Ba6gydF4y2Ba).列表示各个RuvB子单元跨状态的三角形角度。gydF4y2BabgydF4y2Ba,簇中亚单元的单向轨迹(箭头)[E],覆盖距离约7 Å。在六聚体的刚性区将RuvB六聚体叠加在亚基C上。gydF4y2BacgydF4y2Ba,在状态s1到s5的六聚体中所有RuvB亚基的核苷酸占用。ATP水解和核苷酸交换分别发生在聚类[A]和聚类[D]中,并遵循一个事件顺序(红色箭头),从ADP释放(聚类[D], s1→s2)、ATP水解(聚类[A], s2→s3→s4)和ATP摄取(聚类[D], s4→s5)开始。注:将这些事件的顺序联系起来,可以得到如图所示的构象轨迹gydF4y2BabgydF4y2Ba.gydF4y2BadgydF4y2Ba,通过状态s1到s5的RuvB六聚体的构象塑性区域,测量为Cα原子在两个连续的RuvB电机状态之间的均方根误差。所有的状态都与DNA对齐(绿色,浅绿色)。作为参考,子单元E在顶部和底部的视图中被概述。gydF4y2BaegydF4y2Ba,图中显示了核苷酸环周围的相对进展方向(橙色)和DNA旋转以及RuvB(蓝色)。右,核苷酸结合囊和ATPγS、ADP和apo的模拟核苷酸内的代表性低温-电磁密度。gydF4y2BafgydF4y2Ba, atp非水解核苷酸结合袋。形成亚基A核苷酸结合袋的界面的叠加(gydF4y2Ba反式gydF4y2Ba)和B (gydF4y2Ba独联体gydF4y2Ba)从s1到s5。配体囊基本上保持不变。gydF4y2BaggydF4y2Ba, atp水解核苷酸结合袋:亚基A之间形成核苷酸结合袋的界面叠加(gydF4y2Ba独联体gydF4y2Ba)和F (gydF4y2Ba反式gydF4y2Ba)从s1到s5。亚基F中的α-螺旋结构α4和α5逐渐被取代,导致ATP水解。gydF4y2BahgydF4y2Ba,放大突出显示状态s2中未建模的低温- em密度(绿色密度),这可能对应于有序的水分子开始对ATP γ-磷酸进行亲核攻击。gydF4y2Ba
核苷酸周期和构象态gydF4y2Ba
为了研究观察到的RuvB六聚体构象变化与ATP水解之间的相互依赖性,我们首先分析了所有30个核苷酸结合袋的核苷酸身份和占用(扩展数据图。gydF4y2Ba7 a、bgydF4y2Ba).我们发现位于阶梯顶端的簇[A]包含ATPγS (s1和s2), ADP + MggydF4y2Ba2 +gydF4y2Ba(s3)或ADP (s4和s5),这是一种与在这个口袋上进行的ATP水解反应一致的构型。在六聚体的另一侧,簇[D]包含ADP (s1)、碎片和中断密度(s2到s4)或ATPγS (s5)。碎片化和中断密度表明核苷酸占用率低,表明这些位点具有类似载脂蛋白的结构。dna结合簇[B]和[C]完全被ATPγS所占据,而dna游离簇[E]和[F]只与ADP结合(图5)。gydF4y2Ba3 cgydF4y2Ba).gydF4y2Ba
不考虑之前在轨迹上构象变化的顺序,五种状态的核苷酸循环揭示了相同的结构态序列(s1→→s5),从而独立验证了它们的顺序:循环从亚基D中的ADP释放(s1→s2)开始,然后在亚基a中通过三种状态(ATP→ADP + Mg)进行催化反应gydF4y2Ba2 +gydF4y2Ba→ADP (s2→s3→s4)),并通过亚基D (s5)吸收ATP完成(图5)。gydF4y2Ba3 dgydF4y2Ba).这些数据强调了ATP水解和核苷酸交换分别发生在位于阶梯顶部[A]和底部[D]的相反簇中,并且各个步骤在时空上是分离的(图4)。gydF4y2Ba3汉英gydF4y2Ba).结构内聚需要在相对位置的亚基之间循环,以及上面描述的伴随的构象变化,这表明亚基之间有一个连锁信号链,连接核苷酸循环,最终,DNA易位。gydF4y2Ba
值得注意的是,DNA在所有五种状态下仍然与所有四个阶梯亚基(A到D)结合,因此DNA底物与这些亚基的相互作用与核苷酸结合的类型无关,包括在ATP水解(亚基A)和在交换位置(亚基D)(扩展数据图)。gydF4y2Ba7 cgydF4y2Ba).因此,我们的数据显示,为了在中央RuvB马达孔内重新定位DNA底物,RuvB亚基必须服从核苷酸周期后附加的构象变化。因此,我们推断,核苷酸周期实际上首先在五个状态上启动RuvB亚基,然后获得它们各自邻近簇的构象(扩展数据图。gydF4y2Ba6 fgydF4y2Ba).这也得到了一个事实的支持,即状态s5中的核苷酸排列与状态s1中的配置相同,但六个亚基各自的构象向前移了一个,占据了新的后续状态(s5→s1 ': A(s5)→F(s1 '), B(s5)→A(s1 '),等等)。当这一事件发生时,所有6个RuvB亚基同时转移到下一个构象簇,而核苷酸排列没有任何额外的变化(s5和s1 '中的亚基具有相同的核苷酸占用),将整个六聚体的构象重置为状态s1。因此,我们把这个过程称为“集群开关”(s5→s1)。gydF4y2Ba8gydF4y2Ba).由此可见,所有后续的过程现在都发生在各自相邻的亚基中,这意味着核苷酸水解和所有其他过程都以重复的序列围绕六聚体环进行。gydF4y2Ba
催化中心的重组gydF4y2Ba
为了从结构上深入了解核苷酸结合囊发生的事件,我们首先分析了它们的共同特征。核苷酸在两个连续亚基的界面结合(gydF4y2Ba独联体gydF4y2Ba而且gydF4y2Ba反式gydF4y2Ba),核苷完全夹在RuvB之间gydF4y2Ba年代gydF4y2Ba和RuvBgydF4y2BalgydF4y2Ba一个子单元的定义域gydF4y2Ba35gydF4y2Ba,gydF4y2Ba40gydF4y2Ba(RuvBgydF4y2Ba独联体gydF4y2Ba)(图。gydF4y2Ba3 fgydF4y2Ba和扩展数据图。gydF4y2Ba9 a、bgydF4y2Ba).在所有含atp的囊区中,一个保守的Walker-A基序与ATPγS-Mg结合gydF4y2Ba2 +gydF4y2Ba其中Lys65与ATP γ-磷酸盐相互作用gydF4y2Ba41gydF4y2Ba, Thr66对应MggydF4y2Ba2 +gydF4y2Ba离子(图。gydF4y2Ba3 fgydF4y2Ba).另外的接触点由两个保守的gydF4y2Ba独联体gydF4y2Ba-作用精氨酸残基:Arg21和传感器2精氨酸Arg218gydF4y2Ba42gydF4y2Ba.Arg21位于N端,与ATP α-磷酸结合,而传感器2精氨酸Arg218位于小ATP酶结构域,介导核苷酸感知(图2)。gydF4y2Ba3 fgydF4y2Ba).与之前的研究一致,ATPγS-MggydF4y2Ba2 +gydF4y2Ba反式gydF4y2Ba-传感由两个元件实现:位于α-螺旋α4上的保守特征基序(Glu127-Asp130)和gydF4y2Ba反式gydF4y2Ba-作用于α-螺旋α5上的Arg171gydF4y2Ba40gydF4y2Ba,gydF4y2Ba43gydF4y2Ba(无花果。gydF4y2Ba3 fgydF4y2Ba).因此,Arg171代表典型的精氨酸指,在大多数AAA+ atp酶中被保守,并直接与γ-磷酸盐协调gydF4y2Ba44gydF4y2Ba.此外,两个额外的酸性gydF4y2Ba反式-gydF4y2Ba残基,Glu128和Asp129,感觉gydF4y2Ba顺式gydF4y2Ba分别残基Arg21和Arg218,从而间接稳定核苷酸结合(图。gydF4y2Ba3 fgydF4y2Ba).gydF4y2Ba
为了了解ATP水解和信号转导耦合的分子机制和化学,我们在亚基A中跟踪ATPγ s在(s1)之前、(s2)期间和(s3-s5)水解后的命运,其核苷酸结合囊在gydF4y2Ba反式gydF4y2Ba在通过催化态(s1→→s5)的过渡过程中,来自亚基F的螺旋α4和α5进行协调运动,这使得不同的局部重排成为可能gydF4y2Ba反式gydF4y2Ba-残基对亚基A中的ATP水解至关重要(图。gydF4y2Ba3 ggydF4y2Ba).特别是分子间的相互作用gydF4y2Ba反式gydF4y2Ba-Glu128和gydF4y2Ba独联体gydF4y2Ba-Arg21在s1状态下维护,在以下状态下丢失,使能gydF4y2Ba反式gydF4y2Ba-Glu128来代替精氨酸指gydF4y2Ba反式gydF4y2Ba-Arg171。进一步,在状态s2,残差gydF4y2Ba反式gydF4y2Ba-Tyr131连接gydF4y2Ba独联体gydF4y2Ba-Arg21在协调gydF4y2Ba反式gydF4y2Ba-Asp129,一个事件的出现与连续密度之间gydF4y2Ba反式gydF4y2Ba-Asp129和ATPγS-MggydF4y2Ba2 +gydF4y2Ba复杂的(图。gydF4y2Ba3 hgydF4y2Ba和扩展数据图。gydF4y2Ba9 cgydF4y2Ba).连接密度最好描述为有序的水分子,这是促进ATP γ-磷酸亲核攻击所必需的。这一特征母题的重要性已在突变研究中得到强调gydF4y2Ba反式gydF4y2Ba-Asp129显著降低了分支迁移活性和突变gydF4y2Ba反式gydF4y2Ba-Glu128导致细菌生长缺陷gydF4y2Ba45gydF4y2Ba.作为对发生在s2态亚基A中的ATP水解反应的额外验证,ATP γ-磷酸盐和Walker-B基序残基之间也出现了连接密度gydF4y2Ba独联体gydF4y2Ba-Asp110,类似于gydF4y2Ba反式gydF4y2Ba-Asp129,已被证明对ATP水解很重要gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba(无花果。gydF4y2Ba3 hgydF4y2Ba和扩展数据图。gydF4y2Ba9gydF4y2Ba).gydF4y2Ba
在接下来的状态中,可以观察到ATP水解反应的进展,它首先导致γ-磷酸(s2→s3)的释放(图。gydF4y2Ba3 hgydF4y2Ba和扩展数据图。gydF4y2Ba9gydF4y2Ba).因此,传感器的绑定2gydF4y2Ba独联体gydF4y2Ba-Arg218向核苷酸释放,而MggydF4y2Ba2 +gydF4y2Ba离子通过gydF4y2Ba顺式gydF4y2BaThr66仍然完好无损(图。gydF4y2Ba3 hgydF4y2Ba和扩展数据图。gydF4y2Ba9gydF4y2Ba).在下一个跃迁(s3→s4)中,Mg的损失gydF4y2Ba2 +gydF4y2Ba离子释放gydF4y2Ba独联体gydF4y2Ba-Thr66,它现在协调ADP β-磷酸盐。随后,(s4→s5)gydF4y2Ba独联体gydF4y2Ba-传感器2的arg218离开自己的绑定口袋,标定子单元A,准备进行集群切换。gydF4y2Ba
信息通过变频器进行中继gydF4y2Ba
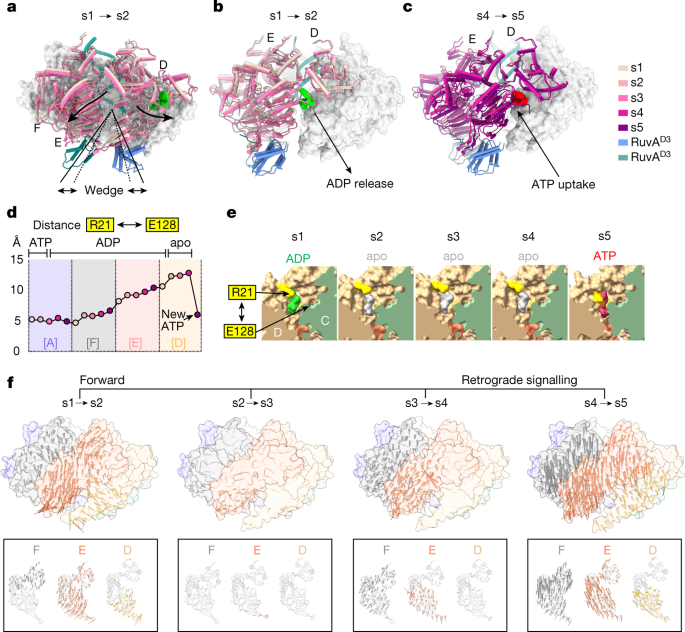
我们观察到RuvA的特殊结合gydF4y2BaD3gydF4y2Ba在亚基A中催化中心对面的RuvB六聚体通过楼梯底部的所有状态(s1到s5)不能解释在RuvA存在时atp酶活性的增加gydF4y2Ba9gydF4y2Ba.相反,它建议RuvAgydF4y2BaD3gydF4y2Ba在这样的安排中,核苷酸循环上产生了调节功能,并直接协调分支迁移。特别是,我们发现一个单一的RuvAgydF4y2BaD3gydF4y2Ba在所有五种状态下都与亚基D结合,这表明在整个核苷酸周期中,RuvA-HJ复合体在三个部分的粒子中被拴在两个相反的RuvB马达上。相比之下,第二个RuvAgydF4y2BaD3gydF4y2Ba在状态s2到s5中,与子单元E独占绑定(扩展数据图。gydF4y2Ba10gydF4y2Ba).两个RuvAgydF4y2BaD3gydF4y2Ba在它们各自的RuvB亚基中结合到先前描述的疏水复合界面,该亚基由RuvB组成gydF4y2BalgydF4y2Baα-螺旋α3和显子-1 β-发夹gydF4y2Ba15gydF4y2Ba(扩展数据图。gydF4y2Ba10 bgydF4y2Ba),在PS1的其他六聚体AAA+电机中直接或间接地插入超支链坐标与其基板gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba47gydF4y2Ba.分析了RuvA的作用gydF4y2BaD3gydF4y2Ba结合(s1→s2)到子基E表明结合事件对RuvB六聚子产生楔形作用,使子基E和d的大结构域分离gydF4y2BaD3gydF4y2Ba结合是通过诱导配合机制实现的。gydF4y2Ba4gydF4y2Ba,扩展数据图。gydF4y2Ba10 cgydF4y2Ba和补充视频gydF4y2Ba3 gydF4y2Ba).亚基E的重新定位导致亚基D的大atp酶结构域的同时移位,进而促进其核苷酸结合袋的打开,从而使ADP分子逃脱(图。gydF4y2Ba4 bgydF4y2Ba,扩展数据图。gydF4y2Ba10 dgydF4y2Ba和补充视频gydF4y2Ba4 gydF4y2Ba而且gydF4y2Ba5gydF4y2Ba).因此,我们的数据显示RuvAgydF4y2BaD3gydF4y2Ba(结合到亚基E上)通过作用于亚基d发挥核苷酸交换因子的作用。值得注意的是,与此同时,E的重新定位引起邻近的、dna分离的亚基F的运动gydF4y2Ba反式gydF4y2Ba-作用残基Glu128, Asp129和Arg171促进了A中的ATP水解反应,如上所述。gydF4y2Ba3 g hgydF4y2Ba,扩展数据图。gydF4y2Ba10 egydF4y2Ba和补充视频gydF4y2Ba4 gydF4y2Ba而且gydF4y2Ba5gydF4y2Ba).在此观察的基础上,我们假设RuvAgydF4y2BaD3gydF4y2Ba它还通过与亚基E上的存在体-1 β-发夹结合发挥作用,作为ATP酶激活域,通过前向协调的亚基间信号通路刺激A中的ATP水解。gydF4y2Ba
一个gydF4y2Ba, RuvAgydF4y2BaD3gydF4y2Ba对RuvB电机中变换器的楔状效应进行了研究。颜色对应着核苷酸周期的状态(s1和s2)。箭头表示RuvB的域核心的位移gydF4y2BalgydF4y2Ba(亚基D)和RuvBgydF4y2BalgydF4y2Ba(子单元E)通过结合第二个RuvAgydF4y2BaD3gydF4y2Ba(绿色)。gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba,由RuvA触发的远程亚基间信号gydF4y2BaD3gydF4y2Ba结合导致亚基D的构象变化,导致ADP释放(s1→s2) (ADP绿色)(gydF4y2BabgydF4y2Ba)和ATP摄取(s4→s5) (ATP红)(gydF4y2BacgydF4y2Ba).在s1到s2期间,亚基D的通道打开允许ADP释放,而在过渡s4到s5期间的通道关闭与ATP分子的吸收有关。子单元D、E和F以卡通视图显示;子单元A, B和C显示在表面视图。gydF4y2BadgydF4y2Ba,之间的距离gydF4y2Ba独联体gydF4y2Ba-Arg21和gydF4y2Ba反式gydF4y2Ba-Glu128作为核苷酸周期中RuvB N端打开和关闭门运动的测量。从[F]开始,在[E]继续,在[D]达到最大,导致ADP的释放。gydF4y2BaegydF4y2Ba,集群中闸门开启的特写视图[D]。gydF4y2Ba独联体gydF4y2Ba-Arg21和gydF4y2Ba反式gydF4y2Ba-Glu128用黄色表示。结合核苷酸显示为ADP(绿色)和ATP(红色)。apo样状态用包围ADP分子的体积表示(灰色)。gydF4y2BafgydF4y2Ba,转换器序列信号经过s1→→s5的时空反褶积。构象变化在六聚体或单个亚基(F, E和D;底下一行)。箭头表示所示跃迁的方向和幅度,其中箭头的底部和尖端表示Cα原子在跃迁开始和结束时的坐标。箭头的颜色是根据它们的亚单位标识,每隔一段大于1 Å的距离才显示一次,箭头长度乘以2.5倍。在表面视图中显示的结构表示在过渡开始时各自状态的六聚体。gydF4y2Ba
值得注意的是,这gydF4y2Ba反式gydF4y2Ba亚基F中的-作用残基仅在Mg丢失时才与核苷酸断开gydF4y2Ba2 +gydF4y2Ba这反过来又允许子单元F (s4→s5)的大尺度运动(扩展数据图。gydF4y2Ba6 e, fgydF4y2Ba而且gydF4y2Ba9gydF4y2Ba).在RuvB亚基A中释放与ADP结合的亚基F会引发连锁反应,这也会影响dna分离的亚基e的位置。因此,我们的数据揭示了MggydF4y2Ba2 +gydF4y2Ba离子触发了限制在柔性RuvB亚基(D, E和F)内的逆行间亚基信号传导(图)。gydF4y2Ba2 egydF4y2Ba和扩展数据图。gydF4y2Ba6 e, fgydF4y2Ba).作为后果之一,守门gydF4y2Ba独联体gydF4y2Ba亚基E的-Arg21不能再与ADP α-磷酸在其核苷酸结合口袋中协调,进而导致整个N端从口袋折叠(图。gydF4y2Ba4 d, egydF4y2Ba).这为下一个易位步骤中ADP的释放做好了准备,当集群切换发生,亚基E已经过渡到构象集群[D],在那里它最终受到核苷酸交换。这进一步体现在不断增加gydF4y2BadgydF4y2Ba1和聚类E中的三角形(图。gydF4y2Ba3gydF4y2Ba和扩展数据图。gydF4y2Ba6摄氏度gydF4y2Ba),在分子水平上削弱了n端之间的疏水相互作用gydF4y2Ba独联体gydF4y2Ba-Leu20及其gydF4y2Ba顺式gydF4y2Ba与Thr193、Ile196、Phe197和Asn221等合作伙伴绑定。因此,不稳定gydF4y2Ba独联体gydF4y2Ba-Leu20损害的能力gydF4y2Ba独联体gydF4y2Ba-Arg21来配合ADP α-磷酸盐(扩展数据图。gydF4y2Ba10 fgydF4y2Ba和补充视频gydF4y2Ba6gydF4y2Ba).此外,逆行信号通路影响楼梯底部的亚基D,在状态4时达到其结合袋的最大开口,这表明尽管ADP已经在s2中释放,核苷酸交换在四个状态(s2→→s5)中进行(图4)。gydF4y2Ba4 d, egydF4y2Ba和扩展数据图。gydF4y2Ba10克gydF4y2Ba).一个新的ATP分子(s4→s5)的获得伴随着亚基E和F的协调运动以及亚基D的大区域(以下称为“转换器”:F - E - DgydF4y2BalgydF4y2Ba)(图。gydF4y2Ba4 d, egydF4y2Ba和扩展数据图。gydF4y2Ba10 hgydF4y2Ba).作为这种运动的一部分,新获得的ATP分子的配位通过亚基D中的N端恢复(扩展数据图)。gydF4y2Ba10我,jgydF4y2Ba).因此,RuvB N末端的门打开(簇[E])和门关闭(簇[D])运动可作为核苷酸循环方向性的额外证明。最后,逆行信号传导导致亚基D(大区域)成为RuvB马达中刚性区域的一部分,这标志着核苷酸循环的完成(图。gydF4y2Ba4汉英gydF4y2Ba和补充视频gydF4y2Ba6gydF4y2Ba).gydF4y2Ba
总之,我们的研究结果表明,所有RuvB子基的构象在六聚体和转换体(F-E-DgydF4y2BalgydF4y2Ba)作为一个RuvB马达操作的多域模块,在核苷酸循环过程中进行高度协调的运动。子单元E在这个模块中心的关键位置使RuvA能够绑定gydF4y2BaD3gydF4y2Ba通过亚基间信号传递信息,刺激远端亚基A中的ATP水解和邻近亚基D中的核苷酸交换(图1)。gydF4y2Ba4 fgydF4y2Ba和补充视频gydF4y2Ba7gydF4y2Ba).gydF4y2Ba
杠杆机制gydF4y2Ba
来洞察构象变化的联系中观察到的转换器RuvB电机和DNA易位,我们调查了五个核苷酸的结构由校准周期(s5 s1)所有国家的中心转换器(亚基E)。分析表明,遵循一个连续运动轨迹转化为RuvB电动机的升降运动,单个地区的六聚体提升比例距离单元E(无花果。gydF4y2Ba3 bgydF4y2Ba而且gydF4y2Ba5gydF4y2Ba).这将导致DNA结合接口及其结合的DNA被从RuvA-HJ核心提升7.0 Å左右。因此,我们的数据提供了证据,RuvB马达作为分子杠杆,将整个核苷酸循环中获得的能量转化为拉力,物理上移动DNA约7.0 Å-that is,两个核苷酸,从而实现DNA重组过程中的分支迁移(图。gydF4y2Ba5罪犯gydF4y2Ba和补充视频gydF4y2Ba8gydF4y2Ba而且gydF4y2Ba9gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba,左,RuvB六聚体的表面视图,根据高度的变化作为到聚类距离的函数着色[E]。右,RuvB子基E上单个RuvB运动状态的侧视图叠加(卡通)。gydF4y2BabgydF4y2Ba,位于RuvB六聚体中心的DNA及其结合的RuvB亚基被提升了大约7 Å,相当于沿着DNA的两个核苷酸的距离(处于状态s1(灰色)和s5(白色)的DNA)。gydF4y2BacgydF4y2Ba, RuvB电机杠杆机构原理图。结合RuvAgydF4y2BaD3gydF4y2Ba与底物分离的RuvB亚基E产生一个支点,使RuvB将ATP中包含的能量转化为杠杆作用。gydF4y2BadgydF4y2Ba,说明伴随旋转的马达提(拉)DNA底物与马达重坐(走)之间的区别。gydF4y2BaegydF4y2Ba,由时间分辨低温电磁(t1和t2)得到的起始态(s0)和递进态(s1)之间的转换器的结构相似性,并表示为对应Cα原子之间的每残基r.m.s.d.。两个数据集中的s0状态非常相似,但与s1状态不同。类似地,两个s1态彼此非常相似,但与s0态不同。状态与DNA一致。gydF4y2BafgydF4y2Ba, RuvAB介导的holday结分支连续易位迁移模型。起始状态提供了一个潜在的进入路径(s0→s1)进入核苷酸循环,从状态s1开始。核苷酸周期由状态s1到s5表示,核苷酸在亚基界面上的占用用彩色圆圈表示(红色(ATP),绿色(ADP)和灰色(apo)。在核苷酸周期中,ATP中包含的能量被转化为杠杆作用或功率冲程,导致每个水解ATP分子的两个碱基对的DNA易位。霍利迪结交叉的易位碱基对(青色和红色)也表明了这一点。RuvB亚基发生“寄存器转移”的簇开关,导致中央孔中DNA底物的重新定位(行走)和状态s1的再生。这使得RuvB电机能够产生迭代的功率冲程,从而为连续支路迁移提供了机制基础。gydF4y2Ba
值得注意的是,后续的簇切换仅在核苷酸循环后重新定位RuvB六聚体(沿着DNA底物移动),而不会对DNA施加直接的机械力,因此不会积极促进RuvA-HJ核心中的链交换(分支迁移)gydF4y2Ba48gydF4y2Ba(无花果。gydF4y2Ba5 dgydF4y2Ba和补充视频gydF4y2Ba10克ydF4y2Ba).随着第二个RuvA的加入,RuvB电机的变流器发生了最大的构象变化gydF4y2BaD3gydF4y2Ba(s1→s2),伴随ADP弹射(s1→s2)和ATP摄取(s4→s5)的核苷酸交换反应,说明这两个事件对DNA易位的贡献最大(扩展数据图)。gydF4y2Ba6 e, fgydF4y2Ba).与核苷酸交换相关的运动最近也被认为是26S蛋白酶体的AAA+ atp酶马达的发力步骤gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba.基于我们的发现,我们假定RuvA作为一个支点,这使得RuvB马达通过产生一个功率冲程将DNA拉过RuvA核心,从而促进分支的迁移(图1)。gydF4y2Ba5度gydF4y2Ba).总之,RuvB AAA+ atp酶马达经历了两个连续的过程(核苷酸循环和簇切换),这两个过程既能维持DNA结构不变,又能在分支迁移过程中进行旋转。gydF4y2Ba
时间分辨的低温电子显微镜gydF4y2Ba
在对t2数据集进行结构分析的过程中,我们发现了两个附加的粒子子集,它们显示出核苷酸占用,这与上面描述的顺序核苷酸周期不一致。第一个亚群含有缺乏中心定位的RuvA寡聚体(s0gydF4y2Ba−一gydF4y2Ba(扩展数据图gydF4y2Ba2 egydF4y2Ba).这清楚地表明,4个RuvB亚基A到D被ATPγS所占据,亚基E和F被ADP所占据(扩展数据图。gydF4y2Ba7 a、bgydF4y2Ba).值得注意的是,比密度在低密度阈值可见,表明部分存在ATPγS和MggydF4y2Ba2 +gydF4y2Ba,从而确定一个不对称形成的RuvB六聚体可以携带多达5个ATP分子(扩展数据图。gydF4y2Ba7 a、bgydF4y2Ba).另一个亚群(s0, RuvA结合)的粒子与缺少RuvA的粒子(s0, RuvA结合)具有相同的核苷酸构型gydF4y2Ba−gydF4y2Ba一个gydF4y2Ba)(扩展数据图gydF4y2Ba2 c, egydF4y2Ba而且gydF4y2Ba7 a、bgydF4y2Ba).因为ATP水解(s2→s3)先于核苷酸循环中新的ATP分子(s4→s5)的获得,在亚基a和D中同时存在ATP表明s0状态不是水解循环的一部分。此外,我们还注意到状态s0的转换体采用了一种混合构象,这与核苷酸周期(s1到s5)中看到的任何构象都不同(图5)。gydF4y2Ba5 egydF4y2Ba和扩展数据图。gydF4y2Ba6 bgydF4y2Ba).因此,我们假设这种状态类似于尚未进入核苷酸周期的RuvB六聚体,因此必须首先经过ATP水解或交换,以采用核苷酸排列的位置和构象依赖序列,如图s1到s5状态所示。我们将这样的状态称为“初始状态”(s0)。gydF4y2Ba
为了验证这一假设,我们在相同的条件下对RuvAB-HJ粒子进行了低温em,但在体外重建后不久将样本玻璃化(在30分钟(t1数据集)而不是5小时(t2))(扩展数据图)。gydF4y2Ba1 hgydF4y2Ba).只有两个状态(s0gydF4y2Bat1gydF4y2Ba和s1gydF4y2Bat1gydF4y2Ba)可以从该数据集以高分辨率(3.3 Å)恢复(扩展数据图。gydF4y2Ba二维gydF4y2Ba而且gydF4y2Ba3 i ngydF4y2Ba和扩展数据表gydF4y2Ba1克ydF4y2Ba).在两个t1状态下,只有一个RuvAgydF4y2BaD3gydF4y2Ba结合了RuvB六聚体中的亚基D(扩展数据图)。gydF4y2Ba2 d, egydF4y2Ba),这意味着在孵育5小时(t2)后观察到的s2到s5状态确实是由不断进行的核苷酸循环主动产生的。此外,这一发现证实了RuvAB-HJ配合物(t2)在活性支迁移过程中被玻璃化。在结构级别,状态s0gydF4y2Bat1gydF4y2Ba与s0 (t2)相似(图5)。gydF4y2Ba5 egydF4y2Ba和扩展数据图。gydF4y2Ba10 kgydF4y2Ba),但它在亚基f中包含第五个ATP分子。这一发现证实了s0 (t2)状态最终可以由s0产生的概念gydF4y2Bat1gydF4y2Ba通过ATP水解在亚基F(非过程)。考虑到在细菌细胞中ATP水平通常超过ADP水平gydF4y2Ba51gydF4y2Ba研究表明,在体内RuvB电机首先通过在RuvB亚基(s0有4或5个ATP)上随机优先加载ATP来组装起始状态,然后进入连续序列核苷酸周期(s0→s1→s5),促进分支迁移。gydF4y2Ba
分支迁移的集成模型gydF4y2Ba
我们的研究结果使我们提出了一个起始和分支迁移的模型,该模型假设DNA易位通过一个杠杆机制发生,由ruva -栓系的RuvB六聚体结合DNA旋转执行和控制gydF4y2Ba34gydF4y2Ba(无花果。gydF4y2Ba5 fgydF4y2Ba).gydF4y2Ba
非渐进起始阶段(随机):(1)RuvA四聚体结合在Holliday结及其柔性RuvA上gydF4y2BaD3gydF4y2Ba将RuvB亚基以六聚体的形式组装在新形成的DNA周围,并在ruva结合的Holliday连接(RuvAB-HJ三联体复合体)的每一边以相反的方向排列。(2) RuvB六聚体随机加载核苷酸(ATP或ADP),初始的失配位ATP水解和/或核苷酸交换发生,采用顺序核苷酸排列,如状态s1 (a - b - c - d - e - f: ATP - ATP - ATP - ADP - ADP)。gydF4y2Ba
渐进式易位阶段(顺序):(1)六聚体RuvB马达作为一个单元工作,经历由转换器介导的正向和逆行信号波,并由核苷酸循环提供燃料:首先ADP在亚基D的阶梯底部被弹出,导致阶梯顶部亚基A的ATP水解,随后ATP在亚基D中被摄取。(2)由于在核苷酸周期中,RuvB固定在RuvA的第III域,伴随着RuvB的旋转,DNA被拉出RuvA核心,通过两个核苷酸推进分支迁移(功率冲程)。(3)随着核苷酸周期的进行,RuvB马达被重新定位(簇开关),从而RuvB亚基将采用其相邻邻居的构象。(4)集群切换完成后,RuvAgydF4y2BaD3gydF4y2Ba由于系绳的物理约束,必须游离,并可以自由地重新结合下一个前进的RuvB子单元。通过将构象团簇[E]和[D]限制在RuvA可及的范围内,可以重置电机。要完成360°的完整旋转,这个过程要重复6次。每个亚基将经历至少5个位置特异性构象,分支迁移复合体总共消耗12个ATP分子(每个RuvB马达6个ATP分子),并通过12个核苷酸推进重组DNA。gydF4y2Ba
讨论gydF4y2Ba
这项工作揭示了脱离底物的RuvB亚基的关键作用,其高度协调的运动控制着RuvB六聚体中的核苷酸循环。这些子单元是一个转换器的一部分,通过它来绑定RuvAgydF4y2BaD3gydF4y2Ba到亚基E可以刺激远距离亚基间信号通路,从而导致ATP水解和核苷酸交换。基片脱离子单元是大多数环形AAA+电机的统一特征gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba39gydF4y2Ba这表明该转化器的变体可能也作用于其他AAA+ atp酶。能够在旋转的RuvB电机RuvA上反复发挥其临界功能gydF4y2BaD3gydF4y2Ba结构域需要不断地从RuvB六聚体中释放出来,并与核苷酸循环产生的新生成的结合接口结合。虽然这种旋转背后的驱动力还有待确定,但这种运动的能量来自于核苷酸循环似乎是合理的。由于DNA底物在双四聚体RuvA核的限制下已经折叠成双螺旋结构,我们认为RuvB马达的旋转是由移位DNA的倒卷提供动力的。从RuvAB机械的这种观点来看,双RuvA四聚体在稳定Holliday结中起着重要作用,确保两个DNA底物可以倒回成双螺旋,并为RuvB电机的旋转提供了理论基础。gydF4y2Ba
通过5个不同的过渡态中间产物(s1到s5),我们的数据从结构上证实了在RuvB马达中,核苷酸循环围绕环进行,为六聚体AAA+ atp酶转位酶的保守核心机制原理提供了概念证明gydF4y2Ba37gydF4y2Ba.在RuvAB复合体中,旋转的RuvB马达的序列核苷酸循环使转换器维持在与中心RuvA-HJ复合体相同的区域。因此,单个RuvA四聚体可能足以控制两个RuvB马达的核苷酸循环。然而,在其他六聚体AAA+ ATP酶马达中,连续的ATP水解事件应该导致相应的底物脱离亚基绕环进行。为了在这些马达中操作核苷酸循环,假定的转换器相互作用物必须能够到达AAA+ atp酶马达的每个亚基。这可能为环状AAA+ atp酶马达嵌入多聚支架内,如蛋白酶体或ClpA/X-P提供了理论依据gydF4y2Ba50gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba.或者,RuvA的调节功能可以由底物直接执行。gydF4y2Ba
进一步,我们表明核苷酸周期是构象变化的时空连续体,通过这种构象变化,RuvB AAA+ ATP酶马达将ATP中保留的化学能转化为杠杆作用。的RuvAgydF4y2BaD3gydF4y2Ba转换器中的-结合亚基是这一过程的核心,因为它们与RuvA核心复合物的物理连接产生了将RuvB电机转变为分子杠杆所需的支点。值得注意的是,当DNA被撬动时,它仍然与它的结合界面相联系;因此,我们的数据使我们能够从簇开关(遵循核苷酸周期)中分解杠杆动作(核苷酸周期中的连续步骤)。这揭示了核苷酸循环有助于促进DNA拉拔,同时也启动了RuvB六聚体的簇开关。这个启动事件,虽然不是核苷酸循环本身的一部分,但对于核苷酸循环在环周围的传播至关重要,因此,对于持续的DNA易位至关重要。gydF4y2Ba5 fgydF4y2Ba和补充视频gydF4y2Ba11gydF4y2Ba).值得注意的是,针对核酸和蛋白质易位的六聚体AAA+ ATP酶在其同源底物周围共享一个保守的不对称螺旋结构,进一步认为每个水解ATP分子具有相似的易位率gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba54gydF4y2Ba.类似地,DNA、RNA和蛋白质底物的牵引被认为是由一个共同的序列核苷酸循环驱动的gydF4y2Ba21gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba.基于它们共同的几何和机制特性,我们的研究结果表明,大多数环状AAA+ atp酶易位酶可能作为分子杠杆,有效地将与其核苷酸周期相关的构象变化的一致波转化为其中央孔的固定高度,作为促进底物易位的共同基本机制。gydF4y2Ba
最后,我们的研究结果表明,RuvB电机在变换器中是最可变的,它从初始态(s0和s0)的混合构象变化gydF4y2Bat1gydF4y2Ba)到核苷酸周期(s1-s5)中观察到的时空连续统。由于功能性DNA损伤反应对于细胞内的细菌病原体应对细胞内的氧化环境至关重要,状态特异性靶向转导器可能为抑制RuvB马达——进而通过小分子干扰进行同源重组提供了一种有前途的途径。gydF4y2Ba
方法gydF4y2Ba
蛋白质工程,表达和纯化gydF4y2Ba
RuvA从gydF4y2Ba美国沙门氏菌感染gydF4y2Ba融合到C-端四组氨酸标签上,利用NcoI和SacI限制位点克隆到pET-52b(+)表达载体(Novagen)。重组蛋白的表达gydF4y2Ba大肠杆菌gydF4y2Ba应变BL21 (DE3)。细菌细胞在37℃的LB培养基中培养,LB培养基中添加100µg mlgydF4y2Ba−1gydF4y2Ba氨苄西林在600 nm处吸光度约为0.6。添加1 mM β-异丙基诱导RuvA表达gydF4y2BadgydF4y2Ba-1-巯基半乳糖吡喃苷(IPTG)和培养物在37°C下进一步培养3 h。细胞在4250的温度被制成颗粒gydF4y2BaggydF4y2Ba在4℃下放置10分钟,在20 mM NaCl, 1 mM EDTA, 20 mM Tris-HCl pH 8缓冲液(缓冲液1)中洗涤,在100 mM NaCl, 5%甘油,100 mM Tris-HCl pH 8缓冲液(缓冲液2)中重悬,并在−80℃保存。为了纯化蛋白质,将细胞悬浮液解冻,添加完整的蛋白酶抑制剂鸡尾酒(Sigma Aldrich),通过超声裂解,并通过离心(Beckman jia -25.50, 17,500 rpm, 1 h, 4℃)清除产生的细胞裂解液。将上清液应用于与缓冲液2平衡的5ml HisTrap色谱柱(GE Healthcare)上,用添加500 mM咪唑的缓冲液2梯度洗脱固定化蛋白。将峰组分汇集在一起,对缓冲液2进行透析,并加载到Superdex 200 10/300GL尺寸排除柱(GE Healthcare)中,平衡在100 mM NaCl, 1 mM EDTA, 0.5 mM DTT, 5%甘油,100 mM Tris-HCl ph8缓冲液(缓冲液3)中。收集含有RuvA的峰组分,并在液氮中冷冻,并在−80℃保存。n端截断的RuvB(16-333)从gydF4y2Ba美国酸奶gydF4y2Bac -末端融合到烟草蚀刻病毒(TEV)蛋白酶切割位点上,接着是连接子和HA标签,并使用NcoI和HindIII限制位点克隆到pProEX HTB表达载体(赛默飞世尔科学公司)。蛋白的表达和纯化如描述的那样进行gydF4y2Ba美国沙门氏菌感染gydF4y2Ba.在透析步骤中进行TEV裂解。重组RuvA和RuvB蛋白通过SDS-PAGE进行纯度评估,然后用考马斯西R-250染色,估计纯度高于95%(扩展数据图)。gydF4y2Ba1 a, bgydF4y2Ba表3)。gydF4y2Ba
DNA基板gydF4y2Ba
假日连接与移动(HJ-X26)gydF4y2Ba55gydF4y2Ba和不可移动(HJ-Y2Ap,由Y2AgydF4y2Ba17gydF4y2Ba)的核心是通过退火合成的寡核苷酸(Sigma Aldrich)制备的补充信息表3,按照先前发表的协议gydF4y2Ba56gydF4y2Ba.简单地说,用天然6% PAGE (TAE缓冲液)纯化寡核苷酸,并在退火缓冲液(缓冲液4)(25 mM NaCl, 10 mM Tris-HCl pH 8)中按适当比例混合。退火反应在0.2 ml的试管中进行,并覆盖一层薄薄的矿物油以防止水分蒸发。混合物加热到95°C 10分钟,然后每10分钟以10°C的温度步骤降低温度。为了得到均匀的四向Holliday结制剂,退火反应中加入DNA样品缓冲液(New England Biolabs),用天然6% PAGE (TAE缓冲液)分离。从凝胶中切割出四向Holliday结对应的条带,在5 mM Tris-HCl ph8中孵育洗脱。在dna结合试验(电迁移位移试验(EMSA))中,一个寡核苷酸链被标记为放射性gydF4y2Ba32gydF4y2BaP (3000 Ci mmolgydF4y2Ba−1gydF4y2Ba)在5 '端退火之前。在分支迁移活性测定中,用ATTO 647N荧光标记一条寡核苷酸链。gydF4y2Ba
RuvAB-HJ的体外重建gydF4y2Ba
RuvAB-HJ粒子按照所述进行了重组gydF4y2Ba17gydF4y2Ba,只是稍加修改。将纯化的Holliday结和RuvA混合,加入5mm MgClgydF4y2Ba2gydF4y2Ba.混合物在37°C孵育30分钟,并应用于Superdex 200 10/300GL柱上的大小排除层析与100 mM NaCl和5 mM MgCl平衡gydF4y2Ba2gydF4y2Ba, 5 mM Tris-HCl pH 8缓冲液(缓冲液5)。含有RuvA-HJ配合物的峰组分与纯化的RuvB在10 mM MgCl存在下混合gydF4y2Ba2gydF4y2BaATPγS和ADP等摩尔比(1 mM)。为了形成RuvAB-HJ配合物,混合物在37℃下孵育10分钟,然后冷却到4℃。在玻璃化之前,所有样品都用负染电镜分析RuvAB-HJ络合物的形成。gydF4y2Ba
分支迁移活性测定gydF4y2Ba
按所述测量分支迁移活性gydF4y2Ba57gydF4y2Ba.简单地说,分支迁移反应(20 μl)包含20 nM纯化的荧光标记合成HJ-X26和不同数量的纯化RuvA和RuvB蛋白在缓冲6 (15 mM MgCl)中gydF4y2Ba2gydF4y2Ba, DTT 1 mM, 50 μg mlgydF4y2Ba−1gydF4y2BaBSA, 2 mM ATP, Tris-HCl pH 8)。在37°C孵育指定时间后,用蛋白酶K处理(2 mg ml)消化RuvA和RuvB蛋白gydF4y2Ba−1gydF4y2Ba)和0.5% SDS在37°C孵育10分钟。在反应中加入甘油(最终浓度为30%),在6%聚丙烯酰胺凝胶中通过电泳(135 V, TAE缓冲液,35分钟)测定分支迁移。ChemoStar Touch ECL和荧光图像(INTAS科学成像)显示Holliday结和Holliday结衍生物对应的条带。gydF4y2Ba
电迁移位移凝胶试验gydF4y2Ba
不同数量的纯化RuvA蛋白与5 ' -孵育gydF4y2Ba32gydF4y2Bap标记合成Holliday结(HJ-Y2Ap), 37℃,5 mM EDTA, 1 mM DTT, 100 μg mlgydF4y2Ba−1gydF4y2BaBSA, 30 mM Tris-HCl 8缓冲液(缓冲液7)。在反应中加入DNA样品缓冲液(New England Biolabs),在6%聚丙烯酰胺凝胶(1× TAE)中电泳分析复合物形成。电泳在0.5× TAE缓冲液中,4℃,150 V, 1.5 h。凝胶被晒干,用放射自显影技术观察DNA条带。gydF4y2Ba
为低温电子显微镜准备网格gydF4y2Ba
在新切割的云母片上沉积无定形碳(1-1.5 nm)(徕卡ACE60碳涂层),在120°C下烘烤0.5 h。用氯仿浸渍60 s,晾干30 min清洗定量网格。连续碳网格是通过在水面上漂浮新鲜制备的无定形碳和清洁的强辉光放电(在25 mA下3分钟)Quantifoil网格。网格干燥1小时,然后在120°C烘焙30分钟,并在控制真空下保存最多2周。gydF4y2Ba
负染色电镜gydF4y2Ba
在使用样品之前,使用GloQube Plus辉光放电系统(电子显微镜科学)在25 mA的条件下对栅格进行30秒的正辉光放电。4微升新鲜制备的RuvAB-HJ配合物被应用到碳涂层铜网格上,并孵育30秒。吸除样品,然后用4µl的染色液(2%乙酸铀酰)染色30 s。多余的污渍被擦掉,网格风干至少2分钟。使用Thermo Fisher Scientific Talos L120C TEM和4K Ceta CEMOS相机对网格进行成像。gydF4y2Ba
低温电镜样品制备和数据收集gydF4y2Ba
新鲜的体外重组RuvAB-HJ复合体在玻璃化之前在冰上孵育30分钟(数据集t1)或约5小时(数据集t2)。gydF4y2BaNgydF4y2Ba-十二烷基-β-麦芽糖苷(DDM)添加到浓度~0.005%之前,将蛋白质样品应用到网格。4微升的最终RuvAB-HJ样品被两次应用到辉光放电(30秒,25毫安)的黄金量化箔网格(2/2 300网格)上,其中包含一层薄的(1-1.5纳米)无定形碳(内部制造)。简单地说,在4°C水平位置涂抹第一个样品1分钟后,液体从侧面被吸掉。重复该过程,将样品浸入丙烷:乙烷(63:37)混合物中,使用Vitrobot Mark V (Thermo Fisher Scientific),设置为100%的湿度和4°C。印迹时间从4-7秒不等。玻璃化样品在Thermo Fisher Scientific Titan Krios TEM上成像,工作电压为300 kV,配有场发射枪(XFEG)和带10 eV狭缝的Gatan生物量子能量过滤器和Gatan K3电子探测器。在数据采集过程中,狭缝每6小时重新居中一次。对于t1数据集,在×81,000公称放大倍率(样本水平每像素1.1 Å)的电子计数模式下,共记录了10057张显微照片,包括33帧,时长3秒(总电子曝光为53 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba,对应1.6 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba)使用Thermo Fisher Scientific EPU数据收集软件。离焦范围设置在−0.3 ~ 3 μ m之间。对于t2数据集,使用在电子计数模式下操作的Gatan K2 Summit直接电子探测器和狭缝为10 eV的Gatan能量滤波器,在×130,000公称放大倍率下(在样本水平上每像素1.09 Å)分别记录了30,053张包括20或25帧的显微照片。累积电子暴露量为30.7 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba(对应1.24或1.55 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba在−0.3 ~ 4 μ m离焦范围内曝光5 s(扩展数据表1)。gydF4y2Ba
Cryo-EM图像处理和原子模型构建gydF4y2Ba
使用Relion (3.0b和3.1版本)进行单粒子分析gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba.使用MOTIONCOR2(在Relion中实现)对显微图像帧(电影)进行运动校正gydF4y2Ba60gydF4y2Ba,剂量加权(使用1.24或1.55 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba每一帧是t2和1.55 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba使用CTFFIND4 (v4.1.14)估计对比度传递函数(CTF)参数。gydF4y2Ba61gydF4y2Ba.使用CrYOLO (v1.4)自动从运动校正显微照片中提取粒子gydF4y2Ba62gydF4y2BaGautomatch (v0.56)gydF4y2Ba63gydF4y2Ba或者用人工挑选的粒子子集训练的Relion Autopick。在t1数据集中,大约选取了400万个坐标。粒子图像提取盒大小为80像素(bin = 4),并进行多轮2D分类。只保留同质类的粒子,共计948,812个粒子(重复删除后)。通过重新提取360像素的盒子大小的粒子,以RuvB环(1,881,624个粒子)和中央的RuvA-HJ(948,812个粒子)为中心进行聚焦分类。随后,进行了三轮细化、逐粒子CTF和贝叶斯抛光。此外,对于RuvA-HJ重建,RuvB环出现的信号被减去。对于t2数据集,大约900万个坐标用于粒子提取,随后进行4次分组和多轮二维分类,总共得到1,786,669个粒子。从这些粒子中,确定了三组粒子,并生成了三个粒子子集:(1)含有两个RuvB马达的三方RuvAB-HJ粒子(717,780),(2)含有一个RuvB马达的二部RuvAB-HJ粒子(549,364个粒子),以及(3)缺少RuvA的RuvB - hj复合物(519,525个粒子)。在重建RuvA-RuvB-HJ三方配合物时,只使用了第1组的粒子。 At first, an ab initio model was created in Relion using a smaller subset of particles (ngydF4y2Ba= 50000)。随后的分类和改进导致了一致重建,产生了~8的分辨率Å。第2组的粒子被用来重建RuvAB-HJ二部结构(~3.9 Å)。将一个RuvB电机对应的信号减去后的第1组粒子生成伪二部粒子。聚焦重建程序按照t1数据集的描述执行,结果分别是RuvB电机和中央RuvA-HJ亚复合体的三维重建。使用组合粒子堆栈(第1组和第2组)中的粒子重建了RuvA-HJ亚复合体。对于RuvB结构,总共提取了约230万RuvB电机(从所有三组中),集中,3D分类,并独立细化子集。随后,每粒子CTF、贝叶斯抛光和3D细化进行了两次。应用这个过程可以得到9种不同的RuvB电机结构,分辨率从2.9到4.1 Å不等。使用Relion 3.1计算局部分辨率估计值、金标准分辨率(傅立叶壳相关系数= 0.143)和锐化映射(b因子范围:每聚焦细化30-80)和多体细化gydF4y2Ba64gydF4y2Ba.gydF4y2Ba
首先用SWISS-MODEL生成RuvA和RuvB的同源模型gydF4y2Ba65gydF4y2Ba.对于RuvA,蛋白数据库(PDB)条目1BVS作为结构模板,PDB条目1HQC作为结构模板gydF4y2Ba66gydF4y2Ba作为RuvB的参考模型。使用UCSF Chimera中的fit-in-map工具将模型安装到电子显微镜地图中(v1.13)gydF4y2Ba67gydF4y2Ba.使用Rosetta (v3.12)对初始模型进行细化gydF4y2Ba68gydF4y2Ba通过StarMap v.1.1.12控制gydF4y2Ba69gydF4y2Ba.在ISOLDE (v1.1.2)中进行了进一步的交互改进gydF4y2Ba70gydF4y2Ba, UCSF ChimeraX中的分子动力学引导结构细化工具(v1.2.5)gydF4y2Ba71gydF4y2Ba.最后,用Phenix对得到的坐标文件进行优化。real_space_refine (v1.19.1 - 4122)gydF4y2Ba72gydF4y2Ba使用参考模型约束,严格的旋转器匹配和禁用网格搜索设置。MolProbity服务器gydF4y2Ba73gydF4y2Ba, EMringergydF4y2Ba74gydF4y2Ba(通过凤凰)gydF4y2BaZgydF4y2Ba-score用于验证模型几何形状和模型与地图的拟合(扩展数据图。gydF4y2Ba3 e mgydF4y2Ba,扩展数据表1)。gydF4y2Ba
可视化和分析gydF4y2Ba
使用UCSF Chimera(1.13)、ChimeraX (v1.1和v1.2.5)和PyMOL(2.4.1)进行可视化和分析。二面角分析使用以下残基:(1)大atp酶残基:残基36、73、80、174、55、155、170、94和121;(2)小atp酶:残基249、227、209、196;(3)头:余数282、284、265、306和263。在三角角分析中,质心确定的残基如下:(1)大atp酶:残基20-180;(2)小atp酶:残基181-256,(3)头:残基257-325。对所有模型中每个Cα原子与相应质心的距离进行方差分析(模型与RuvB亚基C对齐)。gydF4y2Ba
报告总结gydF4y2Ba
关于研究设计的进一步信息可在gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到本文。gydF4y2Ba
数据可用性gydF4y2Ba
本研究得到的Cryo-EM密度图已存入电子显微镜数据库(EMDB) (gydF4y2Bawww.emdataresource.orggydF4y2Ba)根据加入守则:gydF4y2Baemd - 13294gydF4y2Ba,gydF4y2Baemd - 13295gydF4y2Ba,gydF4y2Baemd - 13296gydF4y2Ba,gydF4y2Baemd - 13297gydF4y2Ba,gydF4y2Baemd - 13298gydF4y2Ba,gydF4y2Baemd - 13299gydF4y2Ba,gydF4y2Baemd - 13300gydF4y2Ba,gydF4y2Baemd - 13301gydF4y2Ba,gydF4y2Baemd - 13302gydF4y2Ba,gydF4y2Baemd - 13303gydF4y2Ba,gydF4y2Baemd - 13304gydF4y2Ba,gydF4y2Baemd - 13305gydF4y2Ba,gydF4y2Baemd - 15085gydF4y2Ba而且gydF4y2Baemd - 15126gydF4y2Ba.相应坐标已存入蛋白质数据库(PDB) (gydF4y2Bahttps://www.pdb.orggydF4y2Ba)根据加入守则:gydF4y2Ba7培养gydF4y2Ba,gydF4y2Ba7 pbmgydF4y2Ba,gydF4y2Ba7 pbngydF4y2Ba,gydF4y2Ba7 pbogydF4y2Ba,gydF4y2Ba7 pbpgydF4y2Ba,gydF4y2Ba7的精神gydF4y2Ba,gydF4y2Ba7 pbrgydF4y2Ba,gydF4y2Ba7 pbsgydF4y2Ba,gydF4y2Ba7 pbtgydF4y2Ba而且gydF4y2Ba7试译gydF4y2Ba.所有凝胶和印迹的未裁剪版本在补充图中提供。gydF4y2Ba2gydF4y2Ba.所有其他数据均可根据合理要求从相应作者处获得。gydF4y2Ba
参考文献gydF4y2Ba
真菌中基因转换的机制。gydF4y2Ba麝猫。Res。gydF4y2Ba5gydF4y2Ba, 282 - 304(1964)。gydF4y2Ba
RuvABC蛋白重组中间体的加工。gydF4y2Ba为基础。启麝猫。gydF4y2Ba31gydF4y2Ba, 213 - 244(1997)。gydF4y2Ba
比安科,P. R.和吕阳,单分子对大肠杆菌停滞复制叉拯救的洞察。gydF4y2Ba核酸Res。gydF4y2Ba49gydF4y2Ba, 4220 - 4238(2021)。gydF4y2Ba
复制叉反转与基因组稳定性的维持。gydF4y2Ba核酸Res。gydF4y2Ba37gydF4y2Ba3475(2009)。gydF4y2Ba
A.帕森斯,Tsaneva, I. Lloyd, R. G. & S.韦斯特gydF4y2Ba大肠杆菌gydF4y2Ba合成Holliday连接的RuvA和RuvB蛋白。gydF4y2Ba美国国家科学院学报。美国gydF4y2Ba89gydF4y2Ba, 5452 - 5456(1992)。gydF4y2Ba
Tsaneva, i.r ., Müller, B. & West, S. C.节假日连接的atp依赖的分支迁移由RuvA和RuvB蛋白促进gydF4y2Ba大肠杆菌gydF4y2Ba.gydF4y2Ba细胞gydF4y2Ba69gydF4y2Ba, 1171 - 1180(1992)。gydF4y2Ba
岩崎,高桥,M,中田,a,品川,H。gydF4y2Ba大肠杆菌gydF4y2BaRuvA和RuvB蛋白专门与holday连接相互作用并促进分支迁移。gydF4y2BaDev的基因。gydF4y2Ba6gydF4y2Ba, 2214 - 2220(1992)。gydF4y2Ba
Hiom, K. & West, s.c.同源重组中的分支迁移:RuvAB-Holliday连接复合体的体外组装。gydF4y2Ba细胞gydF4y2Ba80gydF4y2Ba, 787 - 793(1995)。gydF4y2Ba
Shiba T, Iwasaki H, Nakata A. & Shinagawa H., so诱导的DNA修复蛋白RuvA和RuvBgydF4y2Ba大肠杆菌gydF4y2Ba: RuvA和RuvB在ATP水解和超卷曲DNA十字形结构复性中的功能相互作用。gydF4y2Ba美国国家科学院学报。美国gydF4y2Ba88gydF4y2Ba, 8445 - 8449(1991)。gydF4y2Ba
RuvB蛋白介导的ATP水解:RuvB六聚体的功能不对称。gydF4y2Ba生物化学gydF4y2Ba34gydF4y2Ba, 9809 - 9818(2002)。gydF4y2Ba
RuvA蛋白、ATP和DNA对RuvB蛋白介导的ATP水解的变构效应。gydF4y2Ba生物化学gydF4y2Ba35gydF4y2Ba, 11228 - 11238(1996)。gydF4y2Ba
Rafferty, J. B.等。DNA重组蛋白RuvA的晶体结构及其与Holliday结结合的模型。gydF4y2Ba科学gydF4y2Ba274gydF4y2Ba, 415 - 421(1996)。gydF4y2Ba
帕森斯,c.a. &韦斯特,s.c.。RuvAB-Holliday结络合物的体外形成。gydF4y2Baj·摩尔,杂志。gydF4y2Ba232gydF4y2Ba, 397 - 405(1993)。gydF4y2Ba
帕森斯,斯塔西亚克,贝内特,R. J.和韦斯特,S. C.促进DNA分支迁移的多亚基复合体结构。gydF4y2Ba自然gydF4y2Ba374gydF4y2Ba, 375 - 378(1995)。gydF4y2Ba
Yu, X, West, s.c. & Egelman, E. H. RuvAB-Holliday结复合体的结构和亚基组成。gydF4y2Baj·摩尔,杂志。gydF4y2Ba266gydF4y2Ba, 217 - 222(1997)。gydF4y2Ba
罗伊,S. M.等。一种八聚RuvA-Holliday结络合物的晶体结构。gydF4y2Ba摩尔。细胞gydF4y2Ba2gydF4y2Ba, 361 - 372(1998)。gydF4y2Ba
Mayanagi, K, Fujiwara, Y, Miyata, T. & Morikawa, K.四聚体RuvA/RuvB/Holliday结DNA复合体的电子显微镜单粒子分析。gydF4y2Ba物化学。Biophys。Commun >,gydF4y2Ba365gydF4y2Ba, 273 - 278(2008)。gydF4y2Ba
Ohnishi T, Hishida T, Harada Y, Iwasaki H. & Shinagawa H. RuvB DNA马达蛋白三个结构域的结构功能分析。gydF4y2Ba生物。化学。gydF4y2Ba280gydF4y2Ba, 30504 - 30510(2005)。gydF4y2Ba
Nishino, T.等。Holliday结识别蛋白RuvA的活动结构域III对RuvB功能的调控。gydF4y2Baj·摩尔,杂志。gydF4y2Ba298gydF4y2Ba, 407 - 416(2000)。gydF4y2Ba
杰瑟普,菲利克斯,杰瑟普,古切。AAA+ atp酶:放大镜下的结构插入。gydF4y2Ba咕咕叫。当今。结构体。医学杂志。gydF4y2Ba66gydF4y2Ba, 119 - 128(2021)。gydF4y2Ba
Khan, Y. A., White, K. I. & Brunger, A. T. AAA+超家族:这些分子机器的结构和机制原理综述。gydF4y2Ba暴击。学生物化学启。摩尔。杂志。gydF4y2Ba57gydF4y2Ba, 156 - 187(2022)。gydF4y2Ba
山田,K.等。RuvA-RuvB配合物的晶体结构:Holliday结迁移电机的结构基础。gydF4y2Ba摩尔。细胞gydF4y2Ba10克ydF4y2Ba, 671 - 681(2002)。gydF4y2Ba
韩玉文,韩玉文等。从RuvB运动蛋白的AAA+ atp酶结构域突出的一个独特的β-发夹参与了与RuvA DNA识别蛋白的相互作用,以促进Holliday连接的分支迁移。gydF4y2Ba生物。化学。gydF4y2Ba276gydF4y2Ba, 35024 - 35028(2001)。gydF4y2Ba
arena - licea, J.等。功能的相互作用gydF4y2Ba麻风杆菌gydF4y2BaRuvA与gydF4y2Ba大肠杆菌gydF4y2Ba节假日路口的RuvB和RuvC。gydF4y2Baj·摩尔,杂志。gydF4y2Ba301gydF4y2Ba, 839 - 850(2000)。gydF4y2Ba
Khanduja, J. S., Tripathi, P. & Muniyappa, K。gydF4y2Ba结核分枝杆菌gydF4y2BaRuvA在同源和异源Holliday连接之间诱导两种不同类型的结构扭曲。gydF4y2Ba生物化学gydF4y2Ba48gydF4y2Ba27-40(2009)。gydF4y2Ba
怀亚特,h。d。m。韦斯特,s。c。霍利迪联合解决。gydF4y2Ba冷泉哈布。教谕。医学杂志。gydF4y2Ba6gydF4y2Baa023192(2014)。gydF4y2Ba
Yamada, K. Ariyoshi, M. & Morikawa, K. DNA同源重组中分支迁移和拆分的三维结构视图。gydF4y2Ba咕咕叫。当今。结构体。医学杂志。gydF4y2Ba14gydF4y2Ba, 130 - 137(2004)。gydF4y2Ba
Amit, R., Gileadi, O. & Stavans, J. ruvab催化的单一Holliday结分支迁移的直接观察。gydF4y2Ba美国国家科学院学报。美国gydF4y2Ba101gydF4y2Ba, 11605 - 11610(2004)。gydF4y2Ba
Dawid, A., Croquette, V., Grigoriev, M. & Heslot, F. ruvab介导的holliday结迁移的单分子研究。gydF4y2Ba美国国家科学院学报。美国gydF4y2Ba101gydF4y2Ba, 11611 - 11616(2004)。gydF4y2Ba
川喜田,贺冈。腺苷-5 ' -的相互作用gydF4y2BaOgydF4y2Ba——(3-thiotriphosphate)与CagydF4y2Ba2 +gydF4y2Ba、镁gydF4y2Ba2 +gydF4y2Ba肌浆网-腺苷三磷酸酶。gydF4y2Ba学生物化学j。gydF4y2Ba91gydF4y2Ba, 1629 - 1637(1982)。gydF4y2Ba
西野真,有吉M,岩崎H, Shinagawa H, Morikawa K. Holliday结结合蛋白RuvA结构域的功能分析。gydF4y2Ba结构gydF4y2Ba6gydF4y2Ba乳(1998)。gydF4y2Ba
van Gool, a.j., Hajibagheri, n.m.a, Stasiak, A. & West, s.c.gydF4y2Ba大肠杆菌gydF4y2BaRuvABC解析体指导Holliday结解析的方向。gydF4y2BaDev的基因。gydF4y2Ba13gydF4y2Ba, 1861 - 1870(1999)。gydF4y2Ba
Eggleston, A. K, Mitchell, A. H. & West, S. C.基因重组后期步骤的体外重建gydF4y2Ba大肠杆菌gydF4y2Ba.gydF4y2Ba细胞gydF4y2Ba89gydF4y2Ba, 607 - 617(1997)。gydF4y2Ba
汉族,Y.-W。et al。直接观察霍利迪结DNA分支迁移过程中的DNA旋转gydF4y2Ba大肠杆菌gydF4y2BaRuvA-RuvB蛋白质复杂。gydF4y2Ba美国国家科学院学报。美国gydF4y2Ba103gydF4y2Ba, 11544 - 11548(2006)。gydF4y2Ba
山田,K.等。霍利迪结迁移动力蛋白RuvB的晶体结构gydF4y2Ba栖热菌属酸奶gydF4y2BaHB8。gydF4y2Ba美国国家科学院学报。美国gydF4y2Ba98gydF4y2Ba, 1442 - 1447(2001)。gydF4y2Ba
李文华,李文华。复制性六聚解旋酶中DNA易位的机理。gydF4y2Ba自然gydF4y2Ba442gydF4y2Ba, 270 - 275(2006)。gydF4y2Ba
普查德斯,桑迪特,C. R. &兰德,G. C.支配AAA+蛋白活性和功能多样性的分子原理。gydF4y2Ba分子细胞生物学。gydF4y2Ba21gydF4y2Ba43-58(2019)。gydF4y2Ba
奈奎斯特,K.和马丁,A.随着环的节拍行进:AAA+蛋白酶的多肽易位。gydF4y2Ba学生物化学的发展趋势。ScigydF4y2Ba39gydF4y2Ba53-60(2014)。gydF4y2Ba
转位阶梯:AAA+电机结构揭示了atp依赖性底物转位的机制。gydF4y2Ba蛋白质科学。gydF4y2Ba29gydF4y2Ba, 407 - 419(2020)。gydF4y2Ba
普特南,c.d.等人。RuvB假日结支路迁移电机的结构与机理。gydF4y2Baj·摩尔,杂志。gydF4y2Ba311gydF4y2Ba, 297 - 310(2001)。gydF4y2Ba
Hishida, T., Iwasaki, H., Yagi, T. & Shinagawa, H. RuvB蛋白Walker motif A在促进假日连接分支迁移中的作用:Walker motif A突变影响RuvB的ATP结合、ATP水解和DNA结合活性。gydF4y2Ba生物。化学。gydF4y2Ba274gydF4y2Ba, 25335 - 25342(1999)。gydF4y2Ba
温德勒,Ciniawsky, Kock, M. & Kube, S. AAA+核苷酸结合袋的结构和功能。gydF4y2BaBiochim。Biophys。学报gydF4y2Ba1823gydF4y2Ba2 - 14(2012)。gydF4y2Ba
石田,田田,韩,y - w。,Fujimoto, S., Iwasaki, H. & Shinagawa, H. Direct evidence that a conserved arginine in RuvB AAA ATPase acts as an allosteric effector for the ATPase activity of the adjacent subunit in a hexamer.中国科学院学报。美国gydF4y2Ba101gydF4y2Ba, 9573 - 9577(2004)。gydF4y2Ba
AAA+蛋白家族结构和功能的基本特征。gydF4y2Ba古生菌gydF4y2Ba2016gydF4y2Ba, 9294307(2016)。gydF4y2Ba
岩崎,H.等。AAA+类解旋酶RuvB和霍利德结分支迁移运动蛋白功能基序的突变分析。gydF4y2Ba摩尔。Microbiol。gydF4y2Ba36gydF4y2Ba, 528 - 538(2000)。gydF4y2Ba
Mézard, C., Davies, A. A., Stasiak, A. & West, S. C. RuvBD113N的生化特性:RuvB六聚体解旋酶基序II的突变影响DNA结合和atp酶活性。gydF4y2Baj·摩尔,杂志。gydF4y2Ba271gydF4y2Ba, 704 - 717(1997)。gydF4y2Ba
Shin, M.等。AAA+蛋白酶Lon中不同运作模式和蛋白酶活化的结构基础。gydF4y2Ba科学。睡觉。gydF4y2Ba6gydF4y2Baeaba8404(2020)。gydF4y2Ba
Tsaneva, i.r. & West, s.c.的靶向与非靶向DNA解旋酶活性的RuvA和RuvB蛋白gydF4y2Ba大肠杆菌gydF4y2Ba.gydF4y2Ba生物。化学。gydF4y2Ba269gydF4y2Ba, 26552 - 26558(1994)。gydF4y2Ba
董燕,等。人26S蛋白酶体底物参与的低温电镜结构和动力学。gydF4y2Ba自然gydF4y2Ba565gydF4y2Ba49-55(2019)。gydF4y2Ba
Peña, A. H. D. L., Goodall, E. A., Gates, S. N., Lander, G. C. & Martin, A.底物参与的26S蛋白酶体结构揭示了atp水解驱动易位的机制。gydF4y2Ba科学gydF4y2Ba362gydF4y2Baeaav0725(2018)。gydF4y2Ba
Meyrat, A.和von Ballmoos, C.生理核苷酸浓度下的ATP合成。gydF4y2Ba科学。代表。gydF4y2Ba9gydF4y2Ba3070(2019)。gydF4y2Ba
洛佩兹,K. E.等人。ClpAP AAA+蛋白酶的构象可塑性与蛋白质展开和蛋白质水解有关。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba27gydF4y2Ba, 406 - 416(2020)。gydF4y2Ba
里普斯坦,Z. A.,瓦希迪,S., Houry, W. A.,鲁宾斯坦,J. L. &凯,L. E.在ClpXP降解机制中,一个过程旋转机制耦合了底物展开和蛋白质水解。gydF4y2BaeLifegydF4y2Ba9gydF4y2Bae52158(2020)。gydF4y2Ba
O 'Shea, V. L.和Berger, J. M.环状核酸易位酶和解旋酶的装载策略。gydF4y2Ba咕咕叫。当今。结构体。医学杂志。gydF4y2Ba25gydF4y2Ba, 16(2014)。gydF4y2Ba
沙阿,班尼特,R. J.和韦斯特,S. C. RuvC假日结的体外激活。gydF4y2Ba核酸Res。gydF4y2Ba22gydF4y2Ba, 2490 - 2497(1994)。gydF4y2Ba
Kallenbach, n.r, Ma, r.i. & Seeman, n.c.由寡核苷酸构成的不可移动的核酸连接。gydF4y2Ba自然gydF4y2Ba305gydF4y2Ba, 829 - 831(1983)。gydF4y2Ba
Constantinou, A. & West, s.c. Holliday结分支迁移和分辨率测定。gydF4y2Ba摩尔。生物方法。gydF4y2Ba262gydF4y2Ba, 239 - 253(2004)。gydF4y2Ba
齐万诺夫,J.等。用于RELION-3中自动高分辨率低温-电磁结构测定的新工具。gydF4y2BaeLifegydF4y2Ba7gydF4y2Bae42166(2018)。gydF4y2Ba
zvanov, J., Nakane, T. & Scheres, S. H. W.从RELION-3.1中的低温电磁数据集估计高阶像差和各向异性放大率。gydF4y2BaIUCrJgydF4y2Ba7gydF4y2Ba253(2020)。gydF4y2Ba
郑世强,郑世强等。MotionCor2:用于改进低温电子显微镜的光束诱导运动的各向异性校正。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 331 - 332(2017)。gydF4y2Ba
CTFFIND4:快速准确的电子显微图像离焦估计。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba192gydF4y2Ba, 216 - 221(2015)。gydF4y2Ba
spire - cryolo粒子采集技术的发展及其在自动低温电磁处理工作流程中的应用。gydF4y2BaCommun。医学杂志。gydF4y2Ba3 gydF4y2Ba1 - 5(2020)。gydF4y2Ba
张凯,l.m.和s.f. Gautomatch:一种高效、方便的基于gpu的自动粒子选择程序(2011)。gydF4y2Bahttp://www.mrc-lmb.cam.ac.uk/kzhang/gydF4y2Ba
Nakane, T., Kimanius, D., Lindahl, E. & Scheres, S. H. W. RELION中通过多体细化的低温电磁单粒子数据中的分子运动特征。gydF4y2BaeLifegydF4y2Ba7gydF4y2Bae36861(2018)。gydF4y2Ba
Bordoli, L.等。利用SWISS-MODEL工作空间进行蛋白质结构同源性建模。gydF4y2BaProtoc Nat。gydF4y2Ba4 gydF4y2Ba1-13(2009)。gydF4y2Ba
山田,K.等。霍利迪结迁移动力蛋白RuvB的晶体结构gydF4y2Ba栖热菌属酸奶gydF4y2BaHB8。gydF4y2Ba美国国家科学院学报。gydF4y2Ba98gydF4y2Ba, 1442 - 1447(2001)。gydF4y2Ba
Pettersen, E. F.等。UCSF chimera -一个用于探索性研究和分析的可视化系统。gydF4y2Baj .第一版。化学。gydF4y2Ba25gydF4y2Ba, 1605 - 1612(2004)。gydF4y2Ba
dimao, F., leaf - fay, A., Bradley, P., Baker, D. & André, I. Rosetta3中对称大分子结构的建模。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba6gydF4y2Bae20450(2011)。gydF4y2Ba
Lugmayr, W.等。StarMap:一个用户友好的工作流程,用于罗塞塔驱动的分子结构优化。gydF4y2BaProtoc Nat。gydF4y2Bahttps://doi.org/10.1038/s41596-022-00757-9gydF4y2Ba(2022)。gydF4y2Ba
伊索尔德:一个物理上真实的环境,用于建立低分辨率电子密度图的模型。gydF4y2BaActa Crystallogr。DgydF4y2Ba74gydF4y2Ba519(2018)。gydF4y2Ba
Pettersen, E. F.等。UCSF ChimeraX:为研究人员、教育工作者和开发人员提供结构可视化。gydF4y2Ba蛋白质科学。gydF4y2Ba30.gydF4y2Ba, 70 - 82(2021)。gydF4y2Ba
Liebschner, D.等。利用x射线、中子和电子测定大分子结构:凤凰的最新进展。gydF4y2BaActa Crystallogr。DgydF4y2Ba75gydF4y2Ba861(2019)。gydF4y2Ba
威廉姆斯,C. J.等。MolProbity:改进全原子结构验证的更多更好的参考数据。gydF4y2Ba蛋白质科学。gydF4y2Ba27gydF4y2Ba, 293 - 315(2018)。gydF4y2Ba
巴拉德,B. A.等。EMRinger:三维冷冻电子显微镜侧链定向模型和地图验证。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 943 - 946(2015)。gydF4y2Ba
确认gydF4y2Ba
我们感谢Marlovits实验室的所有现任和前任成员对这个项目的支持;S. C. West对研究的初步设计提出了重要建议,并提供了纯化的RuvA和RuvB蛋白(gydF4y2Ba大肠杆菌gydF4y2Ba),在我们早期的实验中,它们是积极的对照;H. Kotisch和T. Heuser在初始低温电子显微镜样品制备试验中的支持;M. Strauss为改进低温-电磁玻璃化技术提供有益建议;F. DiMaio在罗塞塔(星图)上寻求帮助;T. I. Croll向ISOLDE寻求帮助;K. Karius为Holliday连接的初始模型提供帮助;以及H. Sondermann和R. Hennell James对手稿的批判性意见。Cryo-EM优化和初始数据收集在VBC电子显微镜设施(奥地利维也纳)完成。最后的低温电镜数据收集在汉堡国家安全局的低温电镜设施进行(由汉堡大学、汉堡大学医学中心和DFG资助号INST152/772-1、152/774-1、152/775-1和152/777-1 FUGG支持)。高性能计算(HPC)可以通过访问位于汉堡(德国)和维也纳科学集群(奥地利)的HPC实现。 This project was supported by funds available to T.C.M. through the Behörde für Wissenschaft, Forschung und Gleichstellung of the city of Hamburg at the Institute of Structural and Systems Biology at the University Medical Center Hamburg–Eppendorf (UKE), Deutsches Elektronen Synchrotron (DESY), the Institute for Molecular Biotechnology (IMBA) of the Austrian Academy of Sciences, and the Research Institute of Molecular Pathology (IMP). L.C. was supported by the Austrian Science Fund (FWF) (I 2408-B22 granted to T.C.M).
资金gydF4y2Ba
开放获取资金由Universitätsklinikum Hamburg-Eppendorf (UKE)提供。gydF4y2Ba
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
T.C.M.是本文的主要通讯作者。J.W.设计实验,生成结构,表达和纯化蛋白质,进行生化分析,组装配合物,进行负染色电子显微镜筛选,优化玻璃化,收集和处理所有低温- em数据。D.F N.G.-M。J.W.建立了原子模型。j.w., D.F.和T.C.M.对数据进行解释和概念化,准备数据和视频,并撰写手稿。W.L.执行和调整了HPC的脚本。j.w., d.f., T.C.M, L.C.和O.V.进行数据可视化。所有作者阅读、修改和批准了稿件。tc.m.构思了这个项目,获得了资金并监督了这个项目。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有竞争利益。gydF4y2Ba
同行评审gydF4y2Ba
同行审查的信息gydF4y2Ba
自然gydF4y2Ba感谢Matthew Jessop和其他匿名审稿人对这项工作的同行评议做出的贡献。同行评审报告可用。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构附属的管辖权要求保持中立。gydF4y2Ba
扩展的数据图和表gydF4y2Ba
图1 RuvAB-HJ复合物的蛋白纯化和体外重建。gydF4y2Ba
a, b, RuvA的域组织和净化(gydF4y2Ba美国沙门氏菌感染gydF4y2Ba) (a)及RuvB (gydF4y2Ba美国酸奶gydF4y2Ba(b)凝胶过滤,SDS-PAGE和考马斯色染色分析单个组分。RuvA洗脱峰为13.25 ml,对应于92 kDa,可能代表四聚体物种(4 x 24 = 96 kDa)。同样,RuvB以12.05 ml洗脱,对应的分子量物种为153 kDa,代表溶液中的四聚体络合物(4 x 37 kDa =1 48 kDa)。分子量是根据凝胶过滤标准的保留时间来估计的。实验重复了至少十次,得到了相似的结果。c, d,重组蛋白HJ分支迁移活性的比较gydF4y2Ba美国沙门氏菌感染gydF4y2BaRuvA或者重组gydF4y2Ba美国沙门氏菌感染gydF4y2BaRuvB (c)或重组gydF4y2Ba美国酸奶gydF4y2BaRuvB (d).随着RuvB浓度的增加,评估两种配合物的分支迁移。分支迁移实验采用荧光标记(红星)的HJs X26在37°C下进行。HJ在肩部含有一个26碱基对同源核和异源序列,以破坏自发分支迁移。实验重复了三次,得到了相似的结果。e,在缺少核苷酸(巷1-2)、存在ATPγ s(巷3-4)或ATP(巷5-6)的情况下,直接比较同源和异质复合体的HJ分支迁移。用8 nM荧光标记(红星)HJs X26、60 nM RuvA和320 nM RuvB在37°C培养16分钟进行分支迁移实验。实验重复了三次,得到了相似的结果。f,电泳迁移率转移试验(EMSA)使用恒定的gydF4y2Ba32gydF4y2Bap标记的HJ (Y2AP)和不断增加的重组RuvA,以确认其HJ结合能力。在ruva浓度为> - 500 nM时,获得了配合物II(两个四聚体与一个HJ结合)的结合饱和度,随后用于体外重建实验。在较低的RuvA浓度(<200 nm)下观察到复合物I,代表一个RuvA四聚体与一个HJ结合。实验重复了三次,得到了相似的结果。g,体外重建的RuvA-HJ配合物的凝胶过滤谱,洗脱峰为9.7 ml,对应分子量约为500 kDa,表明双四聚体结合的Holliday结颗粒均质。这个实验至少重复了十次,都得到了类似的结果。h,本研究中应用的体外重建策略示意图。i、负染RuvAB-HJ分支迁移杂络合物的电镜分析。j.用CTFFIND4对玻璃化RuvAB-HJ分支迁移杂合物的电镜分析和相应的功率谱分析,以测定欠焦和散光。这个实验至少重复了十次,都得到了类似的结果。gydF4y2Ba
图2 Cryo-EM数据收集和单粒子处理。gydF4y2Ba
a, RuvAB-HJ配合物单粒子处理流程。b-d, RuvA- hj核心复合体和RuvB- hj复合体(数据集c中的t2,数据集d中的t1)。e, RuvA对应的最终后处理RuvB地图和密度gydF4y2BaD3gydF4y2Ba,分别对每个RuvB map进行提取。f,典型例子的低温- em密度和建立模型的不同状态的RuvB(残基64-81),RuvA (65-80), RuvB内的DNA和RuvA内的解析HJ。g,低分辨率和高分辨率复合地图的汇编策略。三维重构的伪二部粒子(单RuvB电机信号减除后的三部粒子)(上)被刚体拟合到三部RuvAB-HJ粒子的一致性重构中(中)gydF4y2Ba适应地图gydF4y2Ba在ChimeraX工具。然后将各个映射组合成一个复合映射1(底部),使用gydF4y2Ba体积增加gydF4y2Ba在ChimeraX工具。为了生成高分辨率复合图,使用了4个组件:复合图1中HJ对应的低温-电磁密度、两台聚焦细化的RuvB电机(s2)和聚焦细化的RuvA-HJ复合体。将四个组件拟合到复合映射1中,然后按前面所述进行组合。gydF4y2Ba
图3单粒子重建。gydF4y2Ba
a - d,三方RuvAB-HJ复合体(a)的C1重建的角分布图、局部分辨率估计和傅里叶壳相关(FSC)图,伪二部RuvAB-HJ复合体(单个RuvB电机信号减去后的三方粒子[数据集的“左”或“右”和随后的合并])(b),二部RuvAB-HJ复合体(c)和来自t2数据集的RuvA-HJ核心复合体(d)。RuvB电机状态的局部分辨率估计和z - score: s1 (e), s2 (f), s3 (g), s4 (h), s5 (i), s0 (j), s0gydF4y2Ba——一个gydF4y2Ba(k), s1gydF4y2Bat1gydF4y2Ba(左)和s0gydF4y2Bat1gydF4y2Ba(m). n, RuvB电机重构的傅里叶壳相关(FSC)图:s1 (1), s2 (2), s3 (3), s4 (4), s5 (5), s0 (6), s0gydF4y2Ba——一个gydF4y2Ba(7)、s1gydF4y2Bat1gydF4y2Ba(8)和s0gydF4y2Bat1gydF4y2Ba(9)。gydF4y2Ba
图4 RuvA和RuvB亚基的结构及其在RuvAB-HJ分支迁移复合体中的寡聚组织。gydF4y2Ba
a-c, RuvA由三个域组成。结构域I和II负责将HJ和寡聚体结合成四聚体,而结构域III (n端)从RuvA核心(结构域I和II)延伸并与RuvB电机结合。b,在完全组装的RuvAB-HJ配合物的背景下,RuvA的双四聚体组织的俯视图和侧视图。通过不同的颜色,放大突出了构成中心核心的单个RuvA子单元。c, RuvB由三个领域组成。一个大(RuvBgydF4y2BalgydF4y2Ba)和一个小的(RuvBgydF4y2Ba年代gydF4y2Ba) atp酶子结构域,共同形成atp结合结构域和c端“头”结构域(RuvBgydF4y2BaHgydF4y2Ba)通过带翼螺旋基序与DNA底物结合。作为PS1插入超枝的一个显著特征,RuvB的present -1 β-发夹是RuvB的一部分gydF4y2BalgydF4y2Ba但用蓝色表示。d, RuvB马达的六聚体组装,使用c中的结构域颜色编码。类似于其他六聚体AAA+ atp酶易位酶,核苷酸结合袋位于相邻亚基之间,实现核苷酸依赖的亚基间信号传导。e, RuvB (gydF4y2Ba美国沙门氏菌感染gydF4y2Ba)氨基酸序列,二级结构元素可视化。f,整个RuvAB-HJ三方(低分辨率)三维重建、切点视图和复合图。四个RuvAgydF4y2BaD3gydF4y2Ba域本地化到HJ交叉的同一侧。放大图突出显示了RuvA的两个相邻位置的绑定接口gydF4y2BaD3gydF4y2Ba到RuvB的D, E亚基。g, RuvC二聚体在RuvABC-HJ分辨体中的假定位置。一个RuvA四聚体(绿色)结合HJ交叉并同时操作两个RuvB电机。第二个RuvA四聚体被RuvC的二聚体所取代。h, RuvB电机中DNA结合界面的螺旋阶梯组织。RuvB的电荷分布表示gydF4y2BaHgydF4y2Ba领域在RuvB电机阶梯。在一起,RuvBgydF4y2BaHgydF4y2BaRuvB亚基A, B, C, D的结构域形成一个带正电荷的坑,稳定地容纳双链DNA底物的一条链。每个母系DNA底物(粉色/黄色)的一条链由一个RuvB马达处理。gydF4y2Ba
图5 RuvB电机的旋转。gydF4y2Ba
a, RuvAB-HJ二部粒子的3d分类。反复分类表明,在大约。~ 7%的粒子,RuvB电机相对于RuvA-HJ芯配合物旋转60°。b, RuvAB-HJ三部(伪二部)粒子的3d分类。反复分类表明,在大约。~ 6%的粒子,RuvB电机相对于RuvA-HJ核心配合物旋转60°。c, RuvAB-HJ粒子的多体细化。主成分分析后最丰富的七个特征向量对应的映射分别显示在顶视图和侧视图中。沿着单个特征向量(灰色和红色对应运动的起点和终点)重新定位重构的身体密度。体1和体2表示RELION中多体精化的体定义。 d, Contribution of each of the twelve eigenvectors to the overall variance (in %). The first 7 eigenvectors cover ~95 % of all movements. The rotational motions of eigenvectors 1 and 2, together, cover ~45 % of the variance. A wobbling motion of the RuvB motor with respect to the RuvA core is represented by eigenvectors 3-6 and amount to ~47.5 % of the variance. The motion increasing the gap size (bouncing) between the two bodies (eigenvector 7) covers ~2.5 % of the observed variance. e, Illustration of the directionalities corresponding to the three predominant trajectories: rotation (45 %), wobbling (47.5 %) and bouncing (2.5 %).
图6 RuvB结构域、子基和六元体的结构分析。gydF4y2Ba
a, RuvB的叠加gydF4y2BalgydF4y2Ba, RuvBgydF4y2Ba年代gydF4y2Ba和RuvBgydF4y2BaHgydF4y2Ba域。域属于从t2数据集(s1-s5, s0和s0)获得的RuvB电机状态的RuvB子单元gydF4y2Ba——一个gydF4y2Ba).低RMSDgydF4y2BaØgydF4y2Ba以及σ值表明,在RuvB电机的核苷酸循环中,RuvB畴以刚体的形式运动。注意,更高的RMSDgydF4y2BaØgydF4y2Ba值RuvBgydF4y2BalgydF4y2Ba主要是由于存在体-1 β-发夹的灵活性。当从分析中排除时,RMSDgydF4y2BaØgydF4y2Baσ = 0.25时降至0.57。b,在RuvB上比对RuvB子单元的结构gydF4y2BaHgydF4y2Ba域。分析表明,RuvB亚基形成构象簇,这也反映了它们在RuvB六聚体中的位置。颜色指示构象团簇[A]-[F]。值得注意的是,起始态s0和s0的亚基EgydF4y2Ba——一个gydF4y2Ba聚类[F]和非聚类[E],突出显示转换器在这些状态下的混合构象。c,核苷酸周期s1-s5中RuvB亚基的角度测量(t2数据集)。两个二面角(RuvB之间的d1gydF4y2BalgydF4y2Ba和RuvBgydF4y2Ba年代gydF4y2Ba和d2之间的RuvBgydF4y2Ba年代gydF4y2Ba和RuvBgydF4y2BaHgydF4y2Ba)和一个三角形(在RuvB的质心之间)gydF4y2BalgydF4y2Ba: RuvBgydF4y2Ba年代gydF4y2Ba: RuvBgydF4y2BaHgydF4y2Ba绘制。当从左到右读取(聚类[F]到[A])时,绘制的角度变化对应于RuvB亚基的构象变化,在RuvB马达的核苷酸循环中进展。条形的颜色对应着核苷酸循环的状态。d, RuvB六聚体的运动分析,突出显示转换器的运动(gydF4y2BaF: E: DgydF4y2BalgydF4y2Ba)。RuvB s1-s5原子模型的俯视图和侧视图(六聚体对齐到不变的RuvB子单元C)。箭头表示匹配C之间运动的幅度(在Å中距离)和方向性gydF4y2BaαgydF4y2Ba-原子对[残基19-330]。为了提高可见性,我们选择了1 Å的截断值,并且只在C处显示箭头gydF4y2BaαgydF4y2Ba-原子对对应每一秒残基。为了便于可视化,每个箭头的长度是测量距离的2.5倍。e, (d)中所有单个子单元的运动分析的反褶积。箭头表示匹配C之间运动的幅度(Å中的距离)和方向gydF4y2BaαgydF4y2Ba原子对。为了提高可见性,我们选择了1 Å的截断值,并且只在C处显示箭头gydF4y2BaαgydF4y2Ba-原子对对应每一秒残基。为了便于可视化,每个箭头的长度是测量距离的2.5倍。f,绘制匹配C的距离[残差20-330](在Å)gydF4y2BaαgydF4y2Ba基于(D)所示的叠加,分别测量了RuvB亚基A、F、E和D中的-原子对。分析表明,RuvB亚基E和F以及RuvBgydF4y2BalgydF4y2Ba(子单元D)作为转换器(gydF4y2BaF: E: DgydF4y2BalgydF4y2Ba)具有高度的灵活性(图。gydF4y2Ba2 egydF4y2Ba,扩展数据图。gydF4y2Ba6 egydF4y2Ba).方框表示在大atp酶域中观察到的RuvB亚基E的轨迹。在亚单元A中,整体运动较小(<3 Å),主要局限于RuvBgydF4y2Ba年代gydF4y2Ba和RuvBgydF4y2BaHgydF4y2Ba域(图。gydF4y2Ba2 egydF4y2Ba,扩展数据图。gydF4y2Ba6 egydF4y2Ba).gydF4y2Ba
扩展数据图7不同RuvB电机状态下共存的核苷酸。gydF4y2Ba
a,从t2 (s1, s2, s3, s4, s5, s0, s0)中获得的核苷酸结合口袋内的低温电磁密度和所有状态的模拟核苷酸gydF4y2Ba——一个gydF4y2Ba和t1 (s1gydF4y2Bat1gydF4y2Ba和s0gydF4y2Bat1gydF4y2Ba)数据集,建立核苷酸模型。注意,标有“ATP”的标签指的是用于结构测定的ATPγ s。“apo”标记指的是核苷酸结合囊内的不连续密度。' ADP* '表示法是指[ADP + MggydF4y2Ba2 +gydF4y2Ba].所有低温-电磁密度显示在相同的等值面上(在每个数据集内)阈值。b,根据RuvB亚基/簇和RuvB电机状态列出了RuvB核苷酸结合袋中的核苷酸。该表举例说明核苷酸周期(s1-s5)由3个ATP和3个ADP结合配体开始并结束(状态s1: ATP结合在亚基A, B, C;状态5:ATP结合在亚基B, C, D)。C,核苷酸周期的线性表示(s1-s5),通过RuvB六聚体可视化其进展。为了简化过程,忽略了RuvB电机的旋转。gydF4y2Ba
图8核苷酸周期、构象簇和亚基位置。gydF4y2Ba
a, RuvB在一个核苷酸周期中的构象簇和亚基的位置。RuvB六聚体中的六个亚基(a - f)在整个核苷酸周期s1-s5中采用了相似但不同的构象(构象簇)(颜色表示六聚体中特定的构象簇)。六聚体的旋转使得在一个核苷酸周期后,亚基的位置发生了变化。60˚;例如,s5中的子单元B现在位于s1中的子单元A的位置。为了为下一轮核苷酸循环做准备,六聚体马达被重置(s5>s1’),通过获得s1表示的构象,但被一个亚基移位(簇开关;例如,s5中的子单元B将改变其构象,成为s1)中的子单元A的构象。这一重新定位过程保持了亚基的构象受限,并在RuvA可及的范围内gydF4y2BaD3gydF4y2Ba,将六聚子连接到RuvA核心是必要的,可以被描述为“RuvB马达沿着DNA行走”。值得注意的是,重复的簇开关可能是前面描述的孤立RuvB马达解旋酶活性的机制基础gydF4y2Ba10克ydF4y2Ba).b,在三个连续的核苷酸周期和簇切换期间,特定亚基的核苷酸身份和构象簇的成员关系。例如,在第一个核苷酸周期中,亚基F保持在其构象簇(即[F])内,并在第二个核苷酸周期中改变其构象成为[E]簇的成员,在第三个核苷酸周期中成为[D]簇的成员。gydF4y2Ba
图9核苷酸循环过程中RuvB核苷酸结合袋在亚基间界面上的打开和关闭运动。gydF4y2Ba
a, RuvB中核苷酸结合袋亚基间界面的低温-电磁密度(灰色)和相应的原子模型(卡通)。在相同的阈值水平(0.026)上绘制了低温-电磁密度曲线。有助于ATP结合和ATP水解的残基用棒表示。b, RuvB核苷酸结合口袋间亚基界面根据其簇[A]-[F]的叠加,使用各自的核苷酸结合gydF4y2Ba独联体gydF4y2Ba-subunit from state s1作为对齐参考,以说明在5个核苷酸周期状态(s1-s5)中参与接口的两个亚基的移动。集群[A]和[B]以及集群[B]和[C]之间的接口几乎是不变的。簇[F]和[A]之间的界面突出了在簇[A]中触发ATP水解的运动。c, RuvB亚基A中的核苷酸结合袋被放大gydF4y2Ba顺式gydF4y2Ba亚基f的残基放大突出了s2状态下未模拟的低温- em密度(绿色密度),这可能对应于在ATP水解反应过程中,有序的水分子启动了对ATP γ-磷酸的亲核攻击。注意,残基D129在s2状态下稳定(并被EM密度完全覆盖),而在其他状态下则不稳定。gydF4y2Ba
图10变流器的构象变化。gydF4y2Ba
a,核苷酸周期中RuvB马达状态(s1-s5)和起始状态(s0 .)对应的原子模型并排比较gydF4y2Ba——一个gydF4y2Ba, s0)从数据集t2中获取。b, RuvA的疏水相互作用gydF4y2BaD3gydF4y2Ba用α -螺旋α3和RuvB亚基D或e的前驱体-1 β发夹建立了RuvB的亲水性氨基酸在绿松石中显示,疏水性残基在sepia中显示。只有一个RuvA的卡通模型gydF4y2BaD3gydF4y2Ba显示。c, RuvB亚基D和E的运动分析集中在RuvAgydF4y2BaD3gydF4y2Ba装订界面突出楔形效果。箭头表示匹配C之间运动的大小和方向gydF4y2BaαgydF4y2Ba原子对。d,与簇内核苷酸交换相关的结构域重排[d]从状态s1到s2的转变(ADP→apo)。核苷酸以表面表示,并用红色突出显示。为了使运动可视化,将RuvB亚基叠加在亚基d的头部区域上。e,在核苷酸周期中,亚基F相对于RuvB亚基A的单向运动。最大的运动发生在状态s4(黄色)到状态s5(红色)的转变,当ATP水解反应完成,MggydF4y2Ba2 +gydF4y2Ba在RuvB亚基A. f的核苷酸结合袋中与ADP分离,在核苷酸循环过程中,RuvB亚基E n端打开运动。注意,开口运动主要在集群中可见[E]。g,促进RuvB的核苷酸交换的叠加分析gydF4y2BaDgydF4y2Ba状态s2, s3和s4的子单元。较低的平均RMSDgydF4y2BaØgydF4y2Ba0.3的Å表明,在三种APO状态下,亚基RuvB D几乎保持不变。h,变频器在RuvB电机中的位置。该转炉由RuvB亚基E和F以及亚基D的大atp酶结构域(均以粉色表示)组成。该转换器连接了RuvB亚基A的atp水解核苷酸结合袋和亚基d的核苷酸交换核苷酸结合袋。i, RuvB亚基D n端在核苷酸循环过程中的闭合运动。注意,闭合运动与新ATP分子的获取相关,因此只能在亚基D中观察到。由于RuvB n端的打开和关闭发生在三个RuvB构象簇上([F], [E]和[D]),这些运动发生在三个易位步骤/核苷酸周期中。j,从s4到s5状态(apo→ATP)转变过程中与核苷酸交换相关的结构域重排[D]。核苷酸以表面表示,并用红色突出显示。为了使运动可视化,将RuvB子单元叠加在头部区域上。k,本研究中获得的所有RuvB电机状态之间的结构可塑性(t1和t2数据集)。 States were aligned to the DNA. Colours indicate the RMSD (in Å). In the top panel, initiation state s0 served as a reference. The two most similar states are the states s0t1gydF4y2Ba(盒装)和RuvAgydF4y2BaD3gydF4y2Ba无状态s0gydF4y2Ba——一个gydF4y2Ba,从RuvB-HJ粒子中获得。在下方的面板中,核苷酸周期状态s1作为参考。最相似的状态是s1gydF4y2Bat1gydF4y2Ba(盒装)。在这两种情况下,与状态s2到s5的比较突出表明,RuvB子单元的运动最大限度地局限于转换器。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
此文件包含补充图1,补充表1 - 3和补充视频图例gydF4y2Ba
补充视频1gydF4y2Ba
RuvB电机相对于RuvA核心复合体旋转。gydF4y2Ba
补充视频2gydF4y2Ba
二部RuvAB-HJ粒子具有高度的柔韧性。gydF4y2Ba
补充视频3gydF4y2Ba
RuvAgydF4y2BaD3gydF4y2Ba绑定在RuvB六分体上施加了一个楔形效应。gydF4y2Ba
补充视频4gydF4y2Ba
RuvB六聚体中与ATP水解相关的运动。gydF4y2Ba
补充视频5gydF4y2Ba
RuvB亚基E和D的运动与核苷酸交换有关。gydF4y2Ba
补充视频6gydF4y2Ba
RuvB N端与核苷酸交换相关的门保持运动。gydF4y2Ba
补充视频7gydF4y2Ba
RuvB六聚体中的前向和逆行信号。gydF4y2Ba
补充视频8gydF4y2Ba
旋转的RuvB马达提升DNA底物的动画。gydF4y2Ba
补充视频9gydF4y2Ba
反褶积I - RuvB马达提升DNA底物的动画。gydF4y2Ba
补充视频10gydF4y2Ba
反褶积II - RuvB马达在DNA底物上行走的动画。gydF4y2Ba
补充视频11gydF4y2Ba
RuvAB-HJ复合体装配加工Holliday接点的动画。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循创作共用署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),该协议允许在任何媒体或格式中使用、分享、改编、分发和复制,只要您给予原作者和来源适当的署名,提供创作共用许可协议的链接,并说明是否有更改。本文中的图片或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果材料不包含在文章的创作共用许可中,并且您的预期用途不被法律法规允许或超出了允许的使用范围,您将需要直接从版权所有者那里获得许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
瓦尔德,J.,法朗坎普,D.,戈斯韦纳-莫尔,N.。gydF4y2Baet al。gydF4y2BaAAA+ atp酶介导的RuvAB-Holliday结分支迁移机制gydF4y2Ba自然gydF4y2Ba609gydF4y2Ba, 630 - 639(2022)。https://doi.org/10.1038/s41586-022-05121-1gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05121-1gydF4y2Ba
这篇文章被引用gydF4y2Ba
StarMap:一个用户友好的工作流,用于罗塞塔驱动的分子结构优化gydF4y2Ba
自然的协议gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba