摘要gydF4y2Ba
细胞rna在加工和二级结构方面是异质的,但这种复杂性的功能重要性仍然知之甚少。一组交替处理的反义非编码转录本,统称为gydF4y2BaCOOLAIRgydF4y2Ba,在gydF4y2Ba拟南芥gydF4y2Bafloral-repressor轨迹gydF4y2Ba开花位点cgydF4y2Ba(gydF4y2Ba方法gydF4y2Ba)gydF4y2Ba1gydF4y2Ba.不同异构体gydF4y2BaCOOLAIRgydF4y2Ba影响gydF4y2Ba方法gydF4y2Ba在温暖和寒冷条件下的转录输出gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba.在这里,进一步研究的功能gydF4y2BaCOOLAIRgydF4y2Ba,我们开发了一种RNA结构分析方法来确定单个RNA分子的体内结构,而不是RNA种群的平均水平。这揭示了个体异构体gydF4y2BaCOOLAIRgydF4y2Ba转录本结构多样,构象动力学不同。主要的远端多腺苷酸化gydF4y2BaCOOLAIRgydF4y2Ba暖条件下的异构体具有三种主要结构构象,冷暴露后其比例和构象发生变化。交替接合的,强烈冷上调的远端gydF4y2BaCOOLAIRgydF4y2Ba同种型gydF4y2Ba6gydF4y2Ba显示高度的结构多样性,与近端聚腺苷化形成对比gydF4y2BaCOOLAIRgydF4y2Ba.一个hyper-variablegydF4y2BaCOOLAIRgydF4y2Ba结构元素被确定为补充gydF4y2Ba方法gydF4y2Ba转录起始位点。突变改变了这个区域的结构gydF4y2Ba方法gydF4y2Ba表达与开花时间一致,具有重要的调节作用gydF4y2BaCOOLAIRgydF4y2Ba结构gydF4y2Ba方法gydF4y2Ba转录。我们的工作表明,非编码RNA转录物的异构体采用多种不同的和功能相关的结构构象,这些结构构象在丰度和形状上随外部条件的变化而变化。gydF4y2Ba
主要gydF4y2Ba
COOLAIRgydF4y2Ba转录本可在近端位点多聚腺苷化,产生约400个核苷酸(nt)的I类转录本,或在远端位点多聚腺苷化,产生约600 - 750个核苷酸(nt)的II类转录本gydF4y2Ba1gydF4y2Ba(无花果。gydF4y2Ba1gydF4y2Ba).不同的gydF4y2BaCOOLAIRgydF4y2Ba异构体在功能上与r -环介导的染色质沉默、转录解抑制有关gydF4y2Ba2gydF4y2Ba,gydF4y2Ba7gydF4y2Ba而且gydF4y2Ba方法gydF4y2Ba寒冷条件下的转录沉默gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba6gydF4y2Ba通过尚不为人所知的机制。RNA的二级结构正在成为RNA功能的重要调节因子gydF4y2Ba8 gydF4y2Ba.体外合成的结构分析gydF4y2BaCOOLAIRgydF4y2Ba揭示了II类的进化守恒gydF4y2BaCOOLAIRgydF4y2Ba结构,尽管低核苷酸序列相同gydF4y2Ba5gydF4y2Ba.然而,知识的gydF4y2BaCOOLAIRgydF4y2Ba体内结构是了解其功能和复杂性的必要条件gydF4y2BaCOOLAIRgydF4y2Ba在活细胞中。目前的化学探测方法限制了这一目的,原因有两个:第一,不可能使用短读测序平台准确地描绘全长结构景观,并区分异构体之间共享区域的结构;其次,RNA构象的异质性使化学探测后RNA二级结构的查询变得复杂。尽管这些技术最近有所改进gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba(gydF4y2Ba补充讨论gydF4y2Ba),直接识别不同RNA异构体和确定单分子体内构象的能力仍然困难。因此,我们开发了一种基于单分子的RNA二级结构探测方法,能够直接确定单个RNA异构体的结构构象。gydF4y2Ba
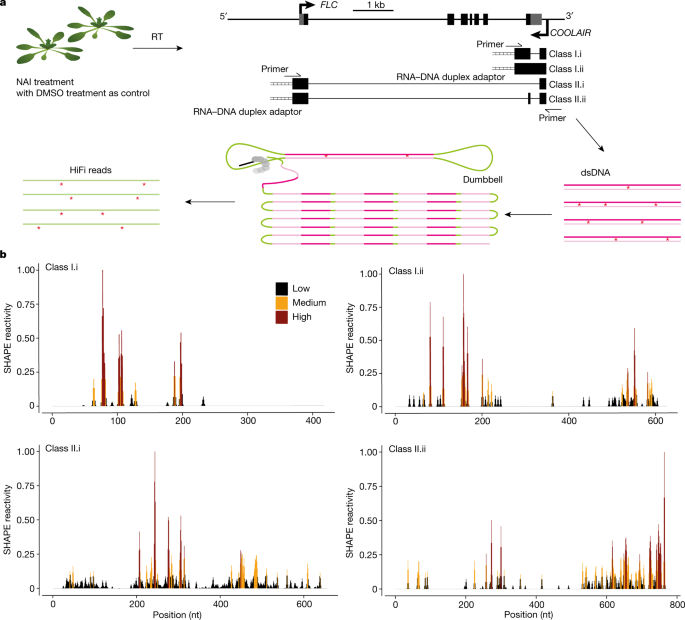
一个gydF4y2Ba,用于RNA二级结构探测的smStructure-seq设计示意图gydF4y2BaCOOLAIRgydF4y2Ba同种型。的gydF4y2Ba拟南芥gydF4y2Ba用NAI ((+)SHAPE)或DMSO((−)SHAPE)处理幼苗。提取总RNA,使用tgrt - iii酶将RNA - dna杂交适配器(梯子符号)加入反转录(RT)反应中。dsdna是通过为所有的dna添加特定的引物而生成的gydF4y2BaCOOLAIRgydF4y2Ba亚型。然后将哑铃适配器连接到结果的dsdna上以生成PacBio库。原始子读数被转换为高精度的HiFi读数(或循环一致序列)gydF4y2Ba14gydF4y2Ba生成突变率分布图。gydF4y2BabgydF4y2Ba,从突变率剖面中得到的归一化SHAPE反应性被绘制为不同的I类(冷生长条件下)和II类(温生长条件下)gydF4y2BaCOOLAIRgydF4y2Ba成绩单亚型。归一化SHAPE反应性由合并计算gydF4y2BangydF4y2Ba= 2个生物重复。这些反应性值用颜色编码并显示在gydF4y2BaygydF4y2Ba轴。gydF4y2Ba
结构多样性gydF4y2BaCOOLAIRgydF4y2Ba亚型gydF4y2Ba
COOLAIRgydF4y2Ba都参与了调节gydF4y2Ba方法gydF4y2Ba决定暖生植物冬季一年生或快速循环繁殖策略的转录输出gydF4y2Ba2gydF4y2Ba以及促进冷诱导的转录关闭,在春化过程中Polycomb抑制性复合物2稳定的表观遗传沉默之前gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.因此,我们描绘了所有主要异构体的体内RNA二级结构景观,即I类和II类gydF4y2BaCOOLAIRgydF4y2Ba转录异构体(图;gydF4y2Ba1gydF4y2Ba和扩展数据图。gydF4y2Ba1gydF4y2Ba)在野生型植物(ColgydF4y2Ba星期五gydF4y2Ba)在温暖的条件下生长,经过两周的寒冷暴露gydF4y2Ba方法gydF4y2Ba转录下调了吗gydF4y2Ba1gydF4y2Ba,gydF4y2Ba12gydF4y2Ba.RNA结构测定采用引物延伸(SHAPE)化学探针分析体内选择性2 ' -羟基酰化gydF4y2Ba拟南芥gydF4y2Ba幼苗。SHAPE试剂,2-甲基烟酸咪唑内酯(NAI),修饰所有四种RNA核苷酸的单链位点gydF4y2Ba13gydF4y2Ba.提取的rna逆转录,修饰位点导致互补DNA (cDNA)突变(图。gydF4y2Ba1gydF4y2Ba).然后,我们将得到的cdna适配到PacBio平台进行单分子实时测序,我们称之为单分子RNA结构测序(smStructure-seq)。对得到的原始读数进行处理,以获得高精度的HiFi读数gydF4y2Ba14gydF4y2Ba根据nai -加合突变谱生成SHAPE反应性(图;gydF4y2Ba1gydF4y2Ba).为了对我们的smStructure-seq数据的重复性和准确性进行基准测试,我们计算了18S rRNA的SHAPE反应性。我们发现我们的smStructure-seq库具有很高的可重复性,Pearson相关性为0.95 (gydF4y2BaPgydF4y2Bavalue = 0.2 × 10gydF4y2Ba−16gydF4y2Ba).通过比较我们的SHAPE反应与18S rRNA系统发育二级结构gydF4y2Ba15gydF4y2Ba,我们发现我们的smStructure-seq分析可以准确地研究体内的全长RNA结构(详细解释在扩展数据图的图例中提供。gydF4y2Ba1 bgydF4y2Ba).gydF4y2Ba
接下来,我们直接计算了I.i类,I.ii类,II类的SHAPE反应性配置文件。i和ii类gydF4y2BaCOOLAIRgydF4y2Ba在温暖和寒冷条件下的异构体(图;gydF4y2Ba1 bgydF4y2Ba和扩展数据图。gydF4y2Ba1 cgydF4y2Ba).I. I类和I.ii类显示了相对较少的具有SHAPE反应性的核苷酸(超过95%的I类异构体的核苷酸在温暖生长的植物中没有显示nai -加合物突变)(扩展数据图。gydF4y2Ba1 cgydF4y2Ba).的gydF4y2BaCOOLAIRgydF4y2BaI类转录本与稳定的R-loop结构相关gydF4y2Ba2gydF4y2Ba,这可能是低反应性的原因。在同一样品中,II类异构体的SHAPE反应性在温暖生长的植物中要高得多(图2)。gydF4y2Ba1 bgydF4y2Ba和扩展数据图。gydF4y2Ba1 cgydF4y2Ba).II类之间的整体SHAPE剖面显著不同。i和ii类(无花果。gydF4y2Ba1 bgydF4y2Ba),尽管这两种亚型大多由相同的序列组成。gydF4y2Ba
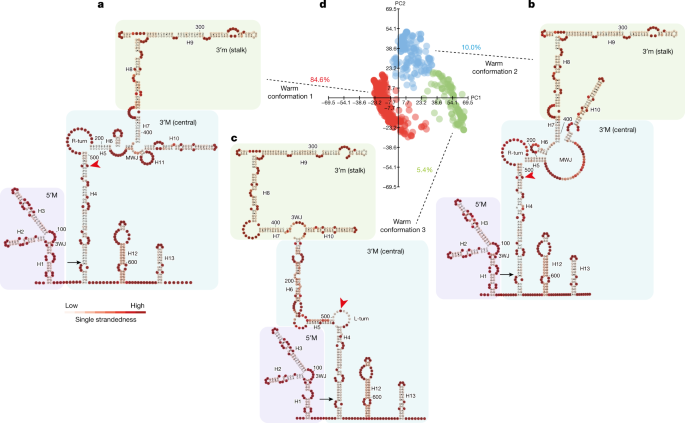
基于热力学参数的RNA结构分析旨在寻找热力学上有利的RNA结构gydF4y2Ba16gydF4y2Ba.然而,长链非编码rna (lncRNAs),如gydF4y2BaCOOLAIRgydF4y2Ba,在共转录调控中动态参与,因此,热力学可能在确定体内RNA结构中起不完全作用gydF4y2Ba17gydF4y2Ba.因此,我们为我们的smStructure-seq开发了一种分析方法,该方法采用了受单个SHAPE反应性谱约束的随机上下文自由语法(SCFG),能够独立于热力学确定单RNA分子的RNA结构。我们将这种结构分析方法命名为DaVinci(通过随机上下文自由语法确定RNA结构构象的变化)。DaVinci可以从每个体内的SHAPE突变剖面中生成单个RNA结构的构象,从而构建广泛的RNA结构景观(扩展数据图)。gydF4y2Ba2gydF4y2Ba).因为DaVinci利用每个单一的突变谱而不是平均的SHAPE突变谱,它可以在单分子分辨率上识别每种可能的构象。为了举例说明这一点,我们发现DaVinci可以识别HIV Rev反应元件(RRE)的一种神秘构象(构象3)。gydF4y2Ba18gydF4y2Ba基于化学反应性的聚类方法无法识别gydF4y2Ba11gydF4y2Ba(扩展数据图。gydF4y2Ba2中gydF4y2Ba).当在RRE61中引入突变时,这种隐式构象成为主要构象(gydF4y2Ba补充讨论gydF4y2Ba;更多的验证显示在扩展数据图中。gydF4y2Ba2 f-hgydF4y2Ba而且gydF4y2Ba3.gydF4y2Ba).利用达芬奇,我们确定了至少三种主要的结构构象gydF4y2BaCOOLAIRgydF4y2BaII级。我,the most abundant (Extended Data Fig.1gydF4y2Ba) II类异构体在温暖条件下(84.6%为温暖构象1;10%温构象2和5.4%温构象3;无花果。gydF4y2Ba2模拟gydF4y2Ba).这些在体内的结构构象被组织成三个域(图。gydF4y2Ba2 a - cgydF4y2Ba):外显子1的5’结构域;外显子2的3 '主结构域(3m)或中心结构域;外显子2中也有3 '小结构域(3m)和柄结构域。所有三种热构象都显示出与体外II类的某种相似性。我的结构gydF4y2Ba5gydF4y2Ba在5 '域和3m域有明显的差异,但在中心3m域有明显的差异(扩展数据图。gydF4y2Ba4 a、c、dgydF4y2Ba).拓扑相似性(树对齐,TA)和碱基配对相似性(阳性预测值,PPV)的测量结果一致表明,离体结构与温暖条件下构象之间的大部分差异都在中心结构域(3m结构域)(扩展数据图)。gydF4y2Ba4模拟gydF4y2Ba).值得注意的是,这一区域被认为是由一个天然的核苷酸多态性改变的gydF4y2Ba答:芥gydF4y2BaVar2-6(参考。gydF4y2Ba7gydF4y2Ba),以提高第II类的产量。iv(扩展数据图。gydF4y2Ba5gydF4y2Ba),这是一份罕见的ColgydF4y2Ba星期五gydF4y2Ba7gydF4y2Ba.II级。我v increases方法gydF4y2Ba通过一种共转录机制的表达,该机制涉及gydF4y2Ba方法gydF4y2Ba新生的成绩单gydF4y2Ba7gydF4y2Ba.我们对携带Var2-6的基因型进行了smStructure-seqgydF4y2Ba方法gydF4y2Ba等位基因渗入ColgydF4y2Ba星期五gydF4y2Ba(扩展数据图。gydF4y2Ba5 bgydF4y2Ba).II类的体内结构。我v has a very short helix 4 (H4) and a merged H5 to extend H6 (Extended Data Fig.5 b, cgydF4y2Ba).这些结构变化发生在与之相辅相成的地区gydF4y2Ba方法gydF4y2Ba转录起始位点(TSS)(扩展数据图)gydF4y2Ba5 b, cgydF4y2Ba).因此,最大的构象变化在远端聚腺苷酸化gydF4y2BaCOOLAIRgydF4y2Ba暖生植物的H4和H6之间的区域,称为超变区;的序列是互补的gydF4y2Ba方法gydF4y2BaTSS(扩展数据图。gydF4y2Ba4 egydF4y2Ba而且gydF4y2Ba5度gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba,暖构象的代表性结构模型1 (gydF4y2Ba一个gydF4y2Ba)、温暖构象2 (gydF4y2BabgydF4y2Ba)和温暖构象3 (gydF4y2BacgydF4y2Ba)gydF4y2BadgydF4y2Ba.根据单链可能性对模型进行着色。控件所对应的站点为红色箭头gydF4y2Ba方法gydF4y2BaTSS。gydF4y2BadgydF4y2Ba, II类在体结构构象的可视化。我喜欢温暖生长的植物。从3061个个体突变谱中直接生成结构。数据采用主成分分析(PCA)进行可视化。每个点代表一个独特的单一结构,源自每个单分子突变谱。3WJ,三通;H#,螺旋数;MWJ,多路结;L-turn,左手转向母题;PC,主成分;r转,右转的主题。 Black arrow,COOLAIRgydF4y2Ba外显子边界;红色箭头,反向互补gydF4y2Ba方法gydF4y2BaTSS。gydF4y2Ba
COOLAIRgydF4y2Ba构象在寒冷中发生变化gydF4y2Ba
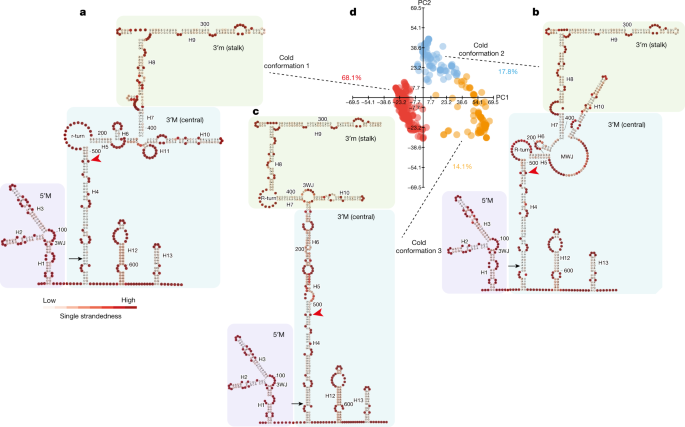
然后我们确定gydF4y2BaCOOLAIRgydF4y2Ba暴露在寒冷环境中两周的植物的异构体特异性结构。冷处理后,I类转录本的SHAPE谱仍然显示出较低的修饰率(图2)。gydF4y2Ba1 bgydF4y2Ba)和第II类。我was still the most abundant class II isoform (Extended Data Fig.1gydF4y2Ba).我们确定了至少三个II类。我conformations (68.1% cold conformation 1; 17.8% cold conformation 2 and 14.1% cold conformation 3 in Fig.3.gydF4y2Ba).冷构象1和2在结构上与热构象1和2相似,但它们的相对比例略有变化(图2)。gydF4y2Ba2gydF4y2Ba而且gydF4y2Ba3.gydF4y2Ba).冷构象3与热构象3不同,在冷构象3中,H4和H6之间的区域连接成一个长茎(图2)。gydF4y2Ba2gydF4y2Ba而且gydF4y2Ba3.gydF4y2Ba).总的来说,有两种主要的II类结构构象。我,the relative proportions of which change in response to cold, with a new conformation emerging in cold-grown plants (cold conformation 3). Comparing the warm-specific (warm conformation 3) and cold-specific (cold conformation 3) structural landscapes of class II.i, the greatest structural difference again occurs in the hyper-variable H4–H6 region complementary to the方法gydF4y2Ba扩展数据图。gydF4y2Ba4 fgydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba、冷构象代表性结构模型1 (gydF4y2Ba一个gydF4y2Ba)、冷构象2 (gydF4y2BabgydF4y2Ba)和冷构象3 (gydF4y2BacgydF4y2Ba)gydF4y2BadgydF4y2Ba.根据单链可能性对模型进行着色。控件所对应的站点为红色箭头gydF4y2Ba方法gydF4y2BaTSS。gydF4y2BadgydF4y2Ba, II类在体结构构象的可视化。我喜欢寒冷生长的植物。从1269个个体突变谱中直接生成结构。使用pca对数据进行可视化。每个点代表一个独特的单一结构,源自每个单分子突变谱。gydF4y2Ba
相比之下,强冷的上调gydF4y2BaCOOLAIRgydF4y2Baii类异构体gydF4y2Ba6gydF4y2Ba,与第II类相比,它包含一个额外的外显子。我,was found not to adopt major conformations (Extended Data Fig.6 a、bgydF4y2Ba).一类整体平均结构模型。ii揭示了四个域(扩展数据图。gydF4y2Ba6 a、bgydF4y2Ba),显示了该亚型的高结构多样性,如高shannon熵所示(扩展数据图。gydF4y2Ba6 c, dgydF4y2Ba).这一特性可能涉及到与friida (FRI)隔离相关的功能。gydF4y2Ba6gydF4y2Ba的主要激活剂gydF4y2Ba方法gydF4y2Ba转录。FRI与一系列与RNA聚合酶II相关的共转录调控因子相关gydF4y2Ba方法gydF4y2Ba启动子区域在温暖条件下是隔离的,在ii类。二、依赖方式,转化成生物分子凝聚物gydF4y2Ba方法gydF4y2Ba冷暴露后的启动子gydF4y2Ba6gydF4y2Ba.gydF4y2Ba
COOLAIRgydF4y2Ba结构解剖gydF4y2Ba
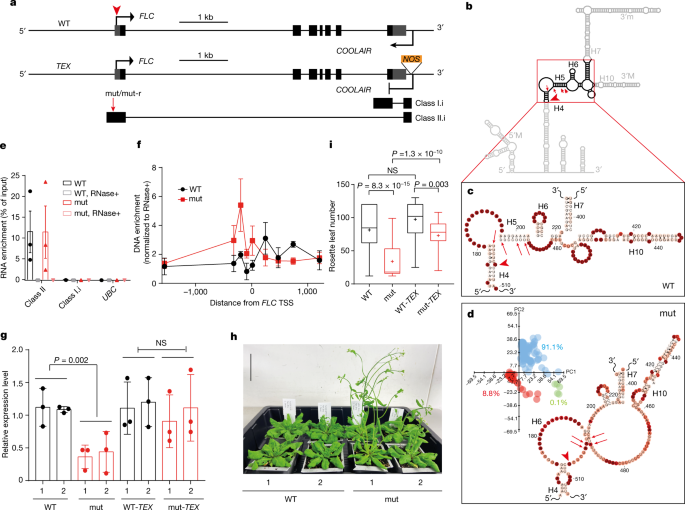
我们的多个结构比较已经确定H4-H6是一个高度可变的区域(扩展数据图。gydF4y2Ba4 e, fgydF4y2Ba而且gydF4y2Ba5度gydF4y2Ba).为了分析该区域的潜在功能作用,我们生成了转基因植物,其中DNA包含四个核苷酸突变(mut),旨在通过缩短H4和H5来增加H4 - h6区域的隆起(图4 - h6)。gydF4y2Ba4模拟gydF4y2Ba和扩展数据图。gydF4y2Ba7一个gydF4y2Ba).这四个突变的结构效应被smStructure-seq证实(图。gydF4y2Ba4 dgydF4y2Ba).然后,我们进行了系统的特征gydF4y2BaCOOLAIRgydF4y2Bamut系中的转录异构体:基因的剪接模式和表达水平gydF4y2BaCOOLAIRgydF4y2Ba不受影响(扩展数据图。gydF4y2Ba8个模拟gydF4y2Ba).然而,染色质结合的比例II类。i增加了mut行(扩展数据图。gydF4y2Ba8 egydF4y2Ba),表明第II类之间的相互作用增强gydF4y2BaCOOLAIRgydF4y2BaRNA和gydF4y2Ba方法gydF4y2Ba染色质。通过RNA纯化(ChIRP)分离染色质证实了这一点,这表明II类染色质的关联增加gydF4y2BaCOOLAIRgydF4y2Ba在整个gydF4y2Ba方法gydF4y2Bamut线中的TSS区域(图;gydF4y2Ba4 e, fgydF4y2Ba).这个5 ' ChIRP信号之前已经被证明对蛋白酶K敏感gydF4y2Ba4 gydF4y2Ba.杂种系产生的未剪接和剪接蛋白水平均较低gydF4y2Ba方法gydF4y2Ba成绩单(无花果。gydF4y2Ba4 ggydF4y2Ba和扩展数据图。gydF4y2Ba8 fgydF4y2Ba),因此开花较早(图;gydF4y2Ba4 h,我gydF4y2Ba).第二个突变体(mut-r),其中引入核苷酸以减少凸起并增加H4-H6螺旋,表现与野生型转基因相似(扩展数据图)。gydF4y2Ba7得了gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba,原理图gydF4y2Ba方法gydF4y2Ba而且gydF4y2BaCOOLAIRgydF4y2Ba野生型(WT)和gydF4y2Ba特克斯gydF4y2Ba转基因线。灰色盒子,未翻译的区域;黑盒子,外显子。gydF4y2BabgydF4y2Ba,主构象突变示意图,温构象1(图;gydF4y2Ba2gydF4y2Ba).gydF4y2BacgydF4y2Ba, II类H4-H6区域。我在the wild-type line from Fig.2gydF4y2Ba.gydF4y2BadgydF4y2Ba, II类H4-H6区域。我在杂种队。插图,达芬奇的二类分析。我在warm-grown mut plants from around 300 individual mutational profiles. The mutation sites are indicated by red arrows in一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba.节点所对应的站点为红色箭头gydF4y2Ba方法gydF4y2BaTSS在gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba.gydF4y2BaegydF4y2Ba, ChIRP-qPCR富集II类RNA。数据为均数±s.e.m.;gydF4y2BangydF4y2Ba= 3个生物独立实验。第I类及gydF4y2Ba哥伦比亚大学gydF4y2Barna,阴性对照。在杂交过程中加入RNase+, RNase A/T1 mix。gydF4y2BafgydF4y2Ba, DNA富集在gydF4y2Ba方法gydF4y2BaTSS区域由II类介导gydF4y2BaCOOLAIRgydF4y2BaChIRP-qPCR。数据为均数±s.e.m.;gydF4y2BangydF4y2Ba= 3个生物独立实验。0表示gydF4y2Ba方法gydF4y2BaTSS。gydF4y2BaggydF4y2Ba,未拼接的相对表达量gydF4y2Ba方法gydF4y2Ba在温暖条件下,RT-qPCR检测到相应基因型的转录。数据为均值±标准差,gydF4y2BangydF4y2Ba= 3个生物独立实验。1和2表示独立的转基因株系。gydF4y2BahgydF4y2Ba,野生型和杂种植物受寒后开花表型。比例尺,50毫米。gydF4y2Ba我gydF4y2Ba,箱形图显示在温暖条件下生长的转基因植物的开花时间,通过莲座叶数测量。中线表示中位数,方框边缘描绘出第25和第75百分位,条形延伸到最小值和最大值,十字表示平均值。gydF4y2BaPgydF4y2Ba值gydF4y2BaggydF4y2Ba而且gydF4y2Ba我gydF4y2Ba均采用单因素方差分析计算。对于每种基因型,从左到右分析混合T3株系的群体,gydF4y2BangydF4y2Ba= 36, 35, 36, 36。gydF4y2Ba
因为引入的突变很接近gydF4y2Ba方法gydF4y2BaTSS,它们可能会影响感觉gydF4y2Ba方法gydF4y2Ba转录活性本身。因此,我们将相同的突变引入转基因,其中反义gydF4y2BaCOOLAIRgydF4y2Ba由于插入了gydF4y2Ba号gydF4y2Ba《终结者》(gydF4y2Ba特克斯gydF4y2Ba2.0)gydF4y2Ba3.gydF4y2Ba(无花果。gydF4y2Ba4gydF4y2Ba).gydF4y2Ba方法gydF4y2Ba转录水平在杂种gydF4y2Ba特克斯gydF4y2Ba和野生型的相似吗gydF4y2Ba特克斯gydF4y2Ba行(WT -gydF4y2Ba特克斯gydF4y2Ba),高于杂种系(图;gydF4y2Ba4 ggydF4y2Ba和扩展数据图。gydF4y2Ba8 fgydF4y2Ba),支持的要求gydF4y2BaCOOLAIRgydF4y2Ba在开花时间的变化引起的突变。的必要性gydF4y2BaCOOLAIRgydF4y2Ba为了与染色质相关联以影响这些功能变化,通过与野生型杂交携带mut转基因的一条线进行了测试。F分析gydF4y2Ba1gydF4y2Ba植物使我们能够检查是否gydF4y2BaCOOLAIRgydF4y2Ba源自mutt转基因的影响gydF4y2Ba方法gydF4y2Ba野生型等位基因表达。我们发现gydF4y2Ba方法gydF4y2BaF的表达水平gydF4y2Ba1gydF4y2Ba株系约为野生型亲本株系的50%(扩展数据图。gydF4y2Ba8 ggydF4y2Ba);因此,结构突变仅在局部起作用gydF4y2Ba方法gydF4y2Ba表达式。综上所述,增加H4-H6区域周围的隆起促进了agydF4y2BaCOOLAIRgydF4y2Ba- - - - - -gydF4y2Ba方法gydF4y2Ba染色质关联,减少转录输出gydF4y2Ba方法gydF4y2Ba缩短了开花的时间。gydF4y2Ba
考虑到H4-H6区域与gydF4y2Ba方法gydF4y2BaTSS区,我们推断其构象依赖gydF4y2BaCOOLAIRgydF4y2Ba- - - - - -gydF4y2Ba方法gydF4y2Ba染色质结合可能包括直接结合gydF4y2BaCOOLAIRgydF4y2Ba来gydF4y2Ba方法gydF4y2BaDNA。潜在的,gydF4y2BaCOOLAIRgydF4y2Ba可以补充gydF4y2Ba方法gydF4y2Ba沃森链形成DNA-RNA双链,虽然我们还没有发现gydF4y2BaCOOLAIRgydF4y2Ba在5 '端形成一个显著的r环gydF4y2Ba方法gydF4y2Ba19gydF4y2Ba.另外,gydF4y2BaCOOLAIRgydF4y2Ba能与双链DNA (dsDNA)结合形成DNA - rna三联体吗gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba(扩展数据图。gydF4y2Ba9gydF4y2Ba);H4-H6区域周围的序列含量(图;gydF4y2Ba4 b, cgydF4y2Ba)能够与dsDNA在位点形成三重结构gydF4y2Ba方法gydF4y2Ba体外TSS(扩展数据图。gydF4y2Ba9 bgydF4y2Ba).但是,由于蛋白酶K的敏感性gydF4y2Ba4 gydF4y2Ba啁啾信号,我们赞成的模型gydF4y2BaCOOLAIRgydF4y2Ba与一种蛋白复合物结合,该蛋白复合物与gydF4y2Ba方法gydF4y2BaTSS。FRI是建立局部染色体环境的关键gydF4y2Ba方法gydF4y2Ba22gydF4y2Ba,因此我们测试了FRI在功能中的参与程度gydF4y2BaCOOLAIRgydF4y2Ba构象通过分析结构突变的转基因(mut)在两种活性gydF4y2Ba星期五gydF4y2Ba和零gydF4y2Ba星期五gydF4y2Ba基因型(扩展数据图。gydF4y2Ba8 hgydF4y2Ba).结构突变影响gydF4y2Ba方法gydF4y2Ba表达式只在gydF4y2Ba星期五gydF4y2Ba基因型(扩展数据图。gydF4y2Ba8 hgydF4y2Ba).因此,除了物理上的FRI与gydF4y2BaCOOLAIRgydF4y2BaII级。我我在cold conditions, the structurally variable region ofCOOLAIRgydF4y2BaII级。我genetically interacts with FRI to regulate方法gydF4y2Ba在温暖的条件下表达。个人如何gydF4y2BaCOOLAIRgydF4y2Ba不同异构体的结构构象影响gydF4y2Ba方法gydF4y2Ba转录将是一个令人兴奋的未来研究领域。gydF4y2Ba
综上所述,单分子RNA结构分析方法的发展使我们能够直接确定反义转录物的体内RNA结构gydF4y2BaCOOLAIRgydF4y2Ba.这种方法使每一种交替处理的结构构象成为可能gydF4y2BaCOOLAIRgydF4y2Ba待描述的异构体。在应对寒冷的条件下,比例gydF4y2BaCOOLAIRgydF4y2Ba采用某种构象会发生变化,新的构象会出现。横跨整个结构景观gydF4y2BaCOOLAIRgydF4y2Ba,我们发现了一种构象变化最大的结构元素,它与gydF4y2Ba方法gydF4y2BaTSS。我们验证了这种结构元素在调节中的功能作用gydF4y2BaCOOLAIR-FLCgydF4y2Ba染色质协会gydF4y2Ba方法gydF4y2Ba这表明RNA构象变化在植物的环境反应中起着功能性作用gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba.我们的研究为lncRNA转录异构体如何采用不同的RNA结构构象,以及这些结构构象如何在功能上影响与染色质的关联并控制转录提供了见解。gydF4y2Ba
方法gydF4y2Ba
统计数据gydF4y2Ba
没有使用统计方法来预先确定样本量。实验不是随机的,研究人员在实验和结果评估期间没有对分配盲目。在所有情况下的采样都是通过从不同的工厂独立收集材料进行的。gydF4y2Ba
植物材料和生长条件gydF4y2Ba
Col基因型gydF4y2Ba星期五gydF4y2BaSF2gydF4y2Ba(坳gydF4y2Ba星期五gydF4y2Ba)和Var2-6近等基因系已在前面描述过gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba7gydF4y2Ba.gydF4y2Ba方法gydF4y2BaWTgydF4y2Ba,gydF4y2Ba方法gydF4y2BaWTgydF4y2Ba-gydF4y2Ba特克斯gydF4y2Ba,gydF4y2Ba方法gydF4y2Ba无足轻重的人gydF4y2Ba,gydF4y2Ba方法gydF4y2Bamut-rgydF4y2Ba,gydF4y2Ba方法gydF4y2Ba无足轻重的人gydF4y2Ba-gydF4y2Ba特克斯gydF4y2Ba而且gydF4y2Ba方法gydF4y2Bamut-rgydF4y2Ba-gydF4y2Ba特克斯gydF4y2Ba转基因系是否携带约12 kb的野生型或突变型gydF4y2Ba方法gydF4y2Ba基因组片段。gydF4y2Ba方法gydF4y2Ba无足轻重的人gydF4y2Ba通过位点定向诱变引入四个核苷酸突变产生。gydF4y2Ba方法gydF4y2BaWTgydF4y2Ba-gydF4y2Ba特克斯gydF4y2Ba而且gydF4y2Ba方法gydF4y2Ba无足轻重的人gydF4y2Ba-gydF4y2Ba特克斯gydF4y2Ba是通过插入gydF4y2Ba号gydF4y2Ba的第一个外显子中的终结者片段gydF4y2BaCOOLAIRgydF4y2Ba在野生型或突变型gydF4y2Ba方法gydF4y2Ba分别是基因组片段gydF4y2Ba3.gydF4y2Ba.gydF4y2Ba方法gydF4y2Bamut-rgydF4y2Ba通过位点定向诱变插入与H4-H6区域大凸起互补的片段(GAAATAAAGCGAGAACAAATGAAAACCCAGGT)产生。施工所用底漆见补充表gydF4y2Ba1gydF4y2Ba.这些片段随后被克隆到SLJ77515中。gydF4y2Ba26gydF4y2Ba),并转化为gydF4y2Ba拟南芥flc-2 FRIgydF4y2Ba基因型gydF4y2Ba3.gydF4y2Ba用浸花的方法。在水稻中鉴定了单插入的抗Basta基因分离为3:1的转基因株系gydF4y2Ba2gydF4y2Ba生成纯合子TgydF4y2Ba3.gydF4y2Ba行。TgydF4y2Ba3.gydF4y2Ba与的纯合子系gydF4y2Ba方法gydF4y2Ba无足轻重的人gydF4y2Ba在gydF4y2Baflc-2星期五gydF4y2Ba背景与Col交叉gydF4y2Ba星期五gydF4y2Ba(WT)为FgydF4y2Ba1gydF4y2Ba(扩展数据图。gydF4y2Ba8 ggydF4y2Ba)或与gydF4y2Baflc-2星期五gydF4y2Ba背景gydF4y2Ba方法gydF4y2Ba无足轻重的人gydF4y2Ba星期五gydF4y2Ba(扩展数据图。gydF4y2Ba8 hgydF4y2Ba).gydF4y2Ba
种子表面消毒后,在半强度Murashige和Skoog培养基上播种。4℃保存2-3天。对于暖生植物,幼苗在温暖的条件下(光照16小时,黑暗8小时,恒定20°C)生长10天。对于冷处理,在温暖条件下进行10天的预生长期后,在5°C(8小时光照和16小时黑暗条件)下进行2周的处理。gydF4y2Ba
(+)SHAPE和(−)SHAPE smStructure-seq库的构造gydF4y2Ba
我们使用SHAPE试剂NAI进行体内RNA二级结构化学探测。NAI是按照以前报告的那样准备的gydF4y2Ba13gydF4y2Ba.简而言之,gydF4y2Ba答:芥gydF4y2Ba用20ml 1× SHAPE反应缓冲液(100mm KCl, 40mm HEPES (pH 7.5)和0.5 mM MgCl完全覆盖幼苗gydF4y2Ba2gydF4y2Ba)装在50毫升的Falcon试管中。将NAI添加到1 M的最终浓度,并在振动筛上旋转(1000转/分钟)。这种高浓度的NAI可以穿透植物细胞并在体内修饰RNA。用新制备的二硫苏糖醇(DTT)猝灭反应后,用去离子水清洗幼苗,立即用液氮冷冻并研磨成粉末。总RNA采用热苯酚法提取gydF4y2Ba4 gydF4y2Ba,然后按照制造商的方案进行DNase I处理。对照组使用DMSO(标记为(−)SHAPE)制备,步骤与上述相同。然后,将2µg (+)SHAPE或(−)SHAPE RNA样品添加到包含2µl 0.5µM RNA - dna杂化接头(5 ' - rargrarurcrgrgrararcrarcrgrurcrarcrgrurcrarcrgrurcrargrururgrarargrararcrcrgrcrgrargrargrargrargrargrararcrcrcrcrgrgrargrargrararcrcrcrcrcrccgatctn (N =等摩尔a, T, g, C)和5 ' - gtgactggagttcagacgtgtgctcttccgatctn (2.25 M NaCl, 25 mM MgCl)的19µl缓冲体系中gydF4y2Ba2gydF4y2Ba, 100 mM Tris-HCl, pH 7.5), 2µl 10× DTT (50 mM;新鲜或冷冻原料)和1µl tgurt - iii酶(10µM;InGex)。反应体系在室温下预孵育30分钟,然后1 μ l 25 mM dNTPs (dATP, dCTP, dGTP和dTTP的等摩尔混合物;每个直径25毫米;rna级)。整个反应体系在管中60℃孵育120 min。为了从模板中去除tgurt - iii酶,加入1 μ l 5 M NaOH,样品在95°C下孵育3分钟。样品冷却至室温,用1µl 5 M HCl中和,然后用MinElute Reaction Cleanup Kit (QIAGEN, 28204)清理cdna。捕获类I和类IIgydF4y2BaCOOLAIRgydF4y2Ba与18S rRNA一起,用特异性引物进行10个循环PCR反应(补充表gydF4y2Ba1gydF4y2Ba)使用KOD Xtreme热启动DNA聚合酶(Novagen)。将8个重复PCR反应的扩增DNA片段合并以获得足够的DNA。所得到的DNA样本使用固相可逆固定尺寸选择系统(BECKMAN COULTER)进行尺寸选择。分别为(+)SHAPE和(−)SHAPE smStructure-seq库生成两个独立的生物重复。纯化的DNA样本由华大基因使用PacBio Sequel 3.0构建PacBio文库。gydF4y2Ba
smStructure-seq数据分析gydF4y2BaCOOLAIRgydF4y2Ba亚型gydF4y2Ba
从(+)SHAPE和(−)SHAPE库中的原始读取被转换为HiFi读取(循环一致序列)使用' ccs ' (gydF4y2Bahttps://github.com/PacificBiosciences/ccsgydF4y2Ba),参数为“——minPasses=3”,以达到99.8%左右的预测精度(Q30)gydF4y2Ba14gydF4y2Ba.HiFi读取使用解复用条形码算法Lima v.1.11.0 (gydF4y2Bahttps://github.com/pacificbiosciences/barcodinggydF4y2Ba).导出的HiFi读取被映射到两者gydF4y2BaCOOLAIRgydF4y2Ba参考文献和18S rRNA(补充表gydF4y2Ba1gydF4y2Ba)使用BLASR (v.5.3.3)gydF4y2Ba27gydF4y2Ba——minMatch 10 -m 5——hitPolicy leftmost '。每一次读取都被转换成一个“位向量”。简而言之,每个位向量对应于单个读取,由一系列0(表示匹配)和1(表示不匹配和明确对齐的删除的突变)组成。gydF4y2Ba11gydF4y2Ba.为了生成总体的SHAPE反应性概况,在给定核苷酸上的突变率(MR)就是1的总数除以该位置上的0和1的总数。II类原始形状反应gydF4y2BaCOOLAIRgydF4y2Ba然后使用下面的等式为每个核苷酸生成:gydF4y2Ba
其中(+)SHAPE对应于nai处理的样品,(−)SHAPE指dmso处理的样品。真负率,1−MRgydF4y2Ba(−)形状gydF4y2Ba,表示特定位置的特异性。原始SHAPE反应性(gydF4y2BaRgydF4y2Ba)从数学上估计SHAPE修正的正似然比。原始SHAPE反应性被标准化为一个标准尺度,从0(无反应性)到1左右(高反应性)。gydF4y2Ba28gydF4y2Ba用来显示突变谱。gydF4y2Ba
第II类结构分析gydF4y2BaCOOLAIRgydF4y2Ba达芬奇的异构体gydF4y2Ba
达芬奇的整个管道在扩展数据图中进行了说明。gydF4y2Ba2gydF4y2Ba.对于II类的每个测序读取,由上一步生成的位向量被转换为约束信息(“1”表示单链核苷酸)gydF4y2BaCOOLAIRgydF4y2Ba亚型。单股约束被纳入DaVinci管道的SCFG发动机。SCFG引擎,包括一组用于SCFG的转换规则和每个非终端符号的转换规则的概率分布,由CONTRAfold提供gydF4y2Ba29gydF4y2Ba在CentroidFold中使用扩展函数实用程序gydF4y2Ba30.gydF4y2Ba(——引擎CONTRAfold——采样)。收集由单个位向量衍生的约束生成的RNA结构。由于不同的结构在探测过程中可能具有相同的突变剖面,因此我们使用位向量约束的采样函数来捕获第ii类的多个结构gydF4y2BaCOOLAIRgydF4y2Ba亚型。将收集到的所有RNA结构转化为点括号字符串,然后使用Forgi包中的rnaConvert转化为RNA结构元素gydF4y2Ba31gydF4y2Ba.提取数字化的RNA二级结构元素以创建数字矩阵,并进行降维,如PCA或多维缩放。对降维结果进行聚类gydF4y2BakgydF4y2Ba-表示使用gydF4y2BakgydF4y2Ba-means来自scikit-learn Python包的函数gydF4y2Ba32gydF4y2Ba.的价值gydF4y2BakgydF4y2Ba设置为视觉确定。通过计算每个位置上最常见的RNA结构类型(即最大预期精度)来确定每个簇的代表性结构,并由位于簇中心且与最常见的RNA结构最相似的RNA结构确定。通过计算构象空间中所有碱基对的频率来计算碱基对概率。位置碱基对概率由gydF4y2Ba\ (P {} _ {} = \ mathop{总和\}\ limits_ {j} ^ {j} {P} _ {ij} \)gydF4y2Ba,在那里gydF4y2BaPgydF4y2BaijgydF4y2Ba底的概率是多少gydF4y2Ba我gydF4y2Ba和基底配对gydF4y2BajgydF4y2Ba,超过了它所有的潜力gydF4y2BaJgydF4y2Ba结对伙伴。单链可能性由1−表达式计算gydF4y2BaPgydF4y2Ba我gydF4y2Ba.此外,香农熵的计算为gydF4y2Ba\ ({E} _{我}= \ mathop{总和\}\ limits_ {j} ^ {j} - {P} _ {ij} {\ log} _{10} \离开({P} _ {ij} \) \)gydF4y2Ba.gydF4y2Ba
HIV-1 RRE、RRE61、cspA、TenA结构分析gydF4y2Ba
检测HIV-1 RRE的数据gydF4y2Ba11gydF4y2Ba均来源于RRE-invitroDMS_NL43rna。bam (gydF4y2Bahttps://codeocean.com/capsule/6175523/tree/v1gydF4y2Ba).探测cspA 5’未翻译区域的数据gydF4y2Ba33gydF4y2Ba在37°C和10°C时,从序列读取档案(存取编号gydF4y2BaSRR6123773gydF4y2Ba而且gydF4y2BaSRR6123774gydF4y2Ba).我们在体外进行了包含茎环III、IV和V的折叠HIV-1 RRE61 RNA (3 pmol)的RNA结构探测实验gydF4y2Ba18gydF4y2Ba如前所述gydF4y2Ba11gydF4y2Ba.对3 pmol的TenA rna进行NAI化学处理gydF4y2Ba13gydF4y2Ba,gydF4y2Ba34gydF4y2Ba在存在或不存在1 μ M硫胺焦磷酸(TPP)时。以20:80 (vol/vol)或50:50 (vol/vol)的比例混合nai修饰的RNA样本(tp处理的RNA和未tp处理的RNA)进行文库构建。如上所述,所有测序数据都映射到相应的参考文献。随后的位向量生成,并进行上述的达芬奇分析,包括为数字化RNA结构元素创建数字矩阵,降维,gydF4y2BakgydF4y2Ba-均值确定和代表性结构构造。RRE的硅结构集成分析采用RNAfold进行Boltzmann采样(10,000次)gydF4y2Ba35gydF4y2Ba.硅内结构集成的后续分析与达芬奇分析相同,但仅包括为数字化RNA结构元素创建数字矩阵、降维、gydF4y2BakgydF4y2Ba-均值确定和代表性结构构造。gydF4y2Ba
总RNA提取和RT-qPCR进行基因表达分析gydF4y2Ba
如前所述,提取总RNAgydF4y2Ba36gydF4y2Ba.根据制造商的指南,在进行反转录之前,使用TURBO DNA-free (Ambion TURBO DNase kit, AM1907)消化基因组DNA。使用SuperScript III逆转录酶(ThermoFisher, 18080093)按照制造商的方案使用基因特异性引物进行逆转录。标准内参基因gydF4y2Ba哥伦比亚大学gydF4y2Ba(gydF4y2BaAt5g25760gydF4y2Ba)对基因表达进行归一化。所有引物均列在补充表中gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
染色质结合RNA测定法gydF4y2Ba
染色质结合rna提取如前所述gydF4y2Ba37gydF4y2Ba.简单地说,2克温种或冷种的幼苗在液氮中用砂浆磨成细粉。然后用1%的材料(约200 mg细粉)进行总RNA提取,如上所述。用Honda缓冲液在50 ng μl的存在下制备剩余材料的核gydF4y2Ba−1gydF4y2BatRNA, 20u mlgydF4y2Ba−1gydF4y2BaRNase抑制剂(SUPERase-In;Life Technologies)和1× cOmplete蛋白酶抑制剂(Roche)。核球在等体积的重悬缓冲液(50% (vol/vol)甘油、0.5 mM EDTA、1 mM DTT、100 mM NaCl和25 mM Tris-HCl pH 7.5)中重悬,并用尿素洗涤缓冲液(300 mM NaCl、1 M尿素、0.5 mM EDTA、1 mM DTT和1% twein -20和25 mM Tris-HCl pH 7.5)洗涤两次。将两体积的洗涤缓冲液添加到重悬的核中,并涡旋1秒。用苯酚-氯仿去除染色质和蛋白质。上清中的rna用异丙醇沉淀,溶解并经dna处理。使用SuperScript III逆转录酶(ThermoFisher, 18080093)按照制造商的方案对染色质结合rna进行逆转录。基因特异性引物的混合物(补充表gydF4y2Ba1gydF4y2Ba),gydF4y2BaEF1alphagydF4y2Ba(gydF4y2BaAt5g60390.2gydF4y2Ba)gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,以估计有多少RNA与基因组DNA结合(表达为(染色质结合RNA)/gydF4y2BaEF1alphagydF4y2Ba),都参与了逆转录反应。总rna也按照制造商的方案使用SuperScript III逆转录酶(ThermoFisher, 18080093)进行逆转录。基因特异性引物的混合物(补充表gydF4y2Ba1gydF4y2Ba),gydF4y2BaPP2AgydF4y2Ba(gydF4y2BaAt1g13320gydF4y2Ba)作为对照,加入到反转录反应中,估计II类的总表达水平(表达为(总RNA)/gydF4y2BaPP2AgydF4y2Ba).用以下公式计算色谱结合比:gydF4y2Ba
ChIRP-qPCR化验gydF4y2Ba
ChIRP的执行与前面概述的一样,只是做了一些修改gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba.反义DNA探针设计针对远端外显子序列gydF4y2BaCOOLAIRgydF4y2BaII类,生物素化在3 '端;探针在补充表中列出gydF4y2Ba1gydF4y2Ba.然后,在室温真空条件下,用3% (vol/vol)甲醛交联3 g温生幼苗。交联后用0.125 M甘氨酸淬灭5 min。交联植物研磨成细粉,在50 ml细胞裂解缓冲液(20 mM Tris-HCl pH 7.5, 250 mM蔗糖,25%甘油,20 mM KCl, 2.5 mM MgCl)中裂解gydF4y2Ba2gydF4y2Ba, 0.1% NP-40和5 mM DTT)。裂解液通过两层miraccloth (Merck, D00172956)过滤,并通过离心成粒。用10毫升核洗涤缓冲液(20mm Tris-HCl pH 7.5, 2.5 mM MgCl)洗涤球团两次gydF4y2Ba2gydF4y2Ba, 25%甘油,0.3% Triton X-100和5 mM DTT)。然后将核颗粒重新悬浮在核裂解缓冲液(50 mM Tris-HCl pH 7.5, 10 mM EDTA, 1% SDS, 0.1 mM PMSF和1 mM DTT)中,并使用Bioruptor超声仪(Diagenode)进行超声处理。所有缓冲液均添加0.1 U μlgydF4y2Ba−1gydF4y2BaRNaseOUT (Life Technologies), 1 mM PMSF和Roche cOmplete片剂,以保持任何rna -蛋白质和蛋白质-蛋白质复合物的完整性。按照前面的描述执行以下步骤gydF4y2Ba40gydF4y2Ba.每个反应均使用30 μl预先阻断的Streptavidin C1磁珠(Thermo Fisher Scientific, 65001)。然后,在RNase+反应中加入20 μl的RNase A/T1 Mix (Thermo Fisher Scientific, EN0551),而不是RNaseOUT。gydF4y2Ba4 egydF4y2Ba),在杂交(37°C 4 h)开始前;这些样本被用作背景噪声的对照。RNA用基因特异性引物用SuperScript IV逆转录酶(ThermoFisher, 18090050)洗脱和逆转录。gydF4y2BaCOOLAIRgydF4y2BaRT-qPCR检测富集和DNA洗脱。所有用于逆转录和RT-qPCR的引物均列在补充表中gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
电泳迁移率移位测定gydF4y2Ba
电泳迁移率移位测定(EMSAs)如前所述进行gydF4y2Ba21gydF4y2Ba使用末端标记有Cy5 (DNA)或FAM (RNA)的寡核苷酸。寡核苷酸序列见补充表gydF4y2Ba1gydF4y2Ba.EMSAs使用自制的15%聚丙烯酰胺凝胶、40 mM三乙酸酯(pH 7.4)和10 mM MgCl进行gydF4y2Ba2gydF4y2Ba15伏厘米gydF4y2Ba−1gydF4y2Ba.凝胶图像使用Typhoon FLA 9500荧光阅读器(GE医疗生命科学)拍摄。阳性对照rDNA增强子En3-PAPAS的序列是从以前的研究中获得的gydF4y2Ba21gydF4y2Ba.gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
测序数据已以BioProject ID号保存在Sequence Read Archive (SRA)中gydF4y2BaPRJNA749291gydF4y2Ba.完整的DNA寡聚物,PCR引物和gydF4y2BaCOOLAIRgydF4y2Ba参考序列见补充表gydF4y2Ba1gydF4y2Ba.支持本研究结果的rna表达水平、RT-qPCR和ChIRP-qPCR的原始数据可作为源数据。EMSA和RT-qPCR的未裁剪图像见补充图。gydF4y2Ba1gydF4y2Ba.收录编号(来自拟南芥信息资源(TAIR;gydF4y2Bahttps://www.arabidopsis.org/gydF4y2Ba)),以获取本研究所分析的基因gydF4y2Ba方法gydF4y2Ba(gydF4y2BaAt5g10140gydF4y2Ba),gydF4y2BaCOOLAIRgydF4y2Ba(gydF4y2BaAt5g01675gydF4y2Ba).标准内参基因gydF4y2BaEF1alphagydF4y2Ba(gydF4y2BaAt5g60390gydF4y2Ba),gydF4y2BaPP2AgydF4y2Ba(gydF4y2BaAt1g13320gydF4y2Ba),gydF4y2Ba哥伦比亚大学gydF4y2Ba(gydF4y2BaAt5g25760gydF4y2Ba)对基因表达进行归一化。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
代码可在GitHub (gydF4y2Bahttps://github.com/DingLab-RNAstructure/smStructure-seqgydF4y2Ba).gydF4y2Ba
改变历史gydF4y2Ba
2022年8月31日gydF4y2Ba
在这篇文章最初发布的版本中,参考文献32中列出的第一作者是不正确的,现在已经在文章的HTML和PDF版本中进行了修改。gydF4y2Ba
参考文献gydF4y2Ba
史维泽斯基,刘芳,马古辛,A. & Dean, C.冷诱导沉默的长反义转录本gydF4y2Ba拟南芥gydF4y2BaPolycomb目标。gydF4y2Ba自然gydF4y2Ba462gydF4y2Ba, 799-802(2009)。gydF4y2Ba
徐,C.等。R-loop分辨率促进共转录染色质沉默。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 1790(2021)。gydF4y2Ba
赵阳,等。自然温度波动促进gydF4y2BaCOOLAIRgydF4y2Ba的监管gydF4y2Ba方法gydF4y2Ba.gydF4y2BaDev的基因。gydF4y2Ba35gydF4y2Ba, 888-898(2021)。gydF4y2Ba
Csorba, T., Questa, J. I., Sun, Q. & Dean, C.反义gydF4y2BaCOOLAIRgydF4y2Ba介导染色质状态的协调切换gydF4y2Ba方法gydF4y2Ba在春化处理。gydF4y2Ba国家科学院学报美国gydF4y2Ba111gydF4y2Ba, 16160-16165(2014)。gydF4y2Ba
霍克斯,E. J.等。gydF4y2BaCOOLAIRgydF4y2Ba反义rna形成进化上保守的复杂二级结构。gydF4y2Ba细胞的代表。gydF4y2Ba16gydF4y2Ba, 3087-3096(2016)。gydF4y2Ba
朱平,李斯特,狄恩gydF4y2Ba拟南芥gydF4y2BaFRIGIDA核凝析液gydF4y2Ba方法gydF4y2Ba镇压。gydF4y2Ba自然gydF4y2Ba599gydF4y2Ba, 657-661(2021)。gydF4y2Ba
李鹏,陶志刚,李鹏。非编码RNA剪接变异的表型进化gydF4y2BaCOOLAIRgydF4y2Ba.gydF4y2BaDev的基因。gydF4y2Ba29gydF4y2Ba, 696-701(2015)。gydF4y2Ba
杨霞,杨敏,邓宏,丁勇。植物RNA二级结构及其对基因调控影响研究的新时代。gydF4y2Ba前面。植物科学。gydF4y2Ba9gydF4y2Ba, 671(2018)。gydF4y2Ba
哦,j。g。a。等人。用纳米孔长读法测定异构体特异性RNA结构。gydF4y2Ba生物科技Nat。》。gydF4y2Ba39gydF4y2Ba, 336-346(2021)。gydF4y2Ba
莫兰迪,E.等。RNA结构集合的基因组尺度反褶积。gydF4y2BaNat方法。gydF4y2Ba18gydF4y2Ba, 249-252(2021)。gydF4y2Ba
Tomezsko, P. J.等人。RNA结构多样性的测定及其在HIV-1 RNA剪接中的作用。gydF4y2Ba自然gydF4y2Ba582gydF4y2Ba, 438-442(2020)。gydF4y2Ba
Yang, H., Howard, M. & Dean, C. H3K36me3和H3K27me3在低温诱导表观遗传开关中的拮抗作用gydF4y2Ba拟南芥方法gydF4y2Ba.gydF4y2Ba咕咕叫。医学杂志。gydF4y2Ba24gydF4y2Ba, 1793-1797(2014)。gydF4y2Ba
Spitale, R. C.等。活细胞中的RNA形状分析。gydF4y2BaNat,化学。医学杂志。gydF4y2Ba9gydF4y2Ba, 18-20(2013)。gydF4y2Ba
温格,a.m.等。精确的循环一致性长读测序提高了人类基因组的变异检测和组装。gydF4y2Ba生物科技Nat。》。gydF4y2Ba37gydF4y2Ba, 1155-1162(2019)。gydF4y2Ba
坎农,J. J.等。比较RNA网站(CRW):核糖体、内含子和其他RNA比较序列和结构信息的在线数据库。gydF4y2BaBMC Bioinform。gydF4y2Ba3.gydF4y2Ba, 2(2002)。gydF4y2Ba
Mathews, d.h., Moss, w.n. & Turner, d.h.折叠和发现RNA二级结构。gydF4y2Ba冷泉港。教谕。医学杂志。gydF4y2Ba2gydF4y2Ba, a003665(2010)。gydF4y2Ba
Rouskin, S., Zubradt, M., Washietl, S., Kellis, M. & Weissman, J. S. RNA结构的全基因组探测揭示了体内mRNA结构的主动展开。gydF4y2Ba自然gydF4y2Ba505gydF4y2Ba, 701-705(2014)。gydF4y2Ba
莱杰维奇,等人。对RevM10抑制的抵抗反映了HIV-1 Rev反应元件中的构象开关。gydF4y2Ba国家科学院学报美国gydF4y2Ba105gydF4y2Ba, 14365-14370(2008)。gydF4y2Ba
孙,Q., Csorba, T., Skourti-Stathaki, K., Proudfoot, N. J. & Dean, C. R-loop稳定抑制反义转录gydF4y2Ba拟南芥方法gydF4y2Ba轨迹。gydF4y2Ba科学gydF4y2Ba340gydF4y2Ba, 619-621(2013)。gydF4y2Ba
Zhao, Z., Sentürk, N., Song, C. & Grummt, I.连接到rDNA增强子的lncRNA PAPAS在高温下招募低磷酸化的CHD4/NuRD来抑制rRNA合成。gydF4y2BaDev的基因。gydF4y2Ba32gydF4y2Ba, 836-848(2018)。gydF4y2Ba
Maldonado, R., Filarsky, M., Grummt, I. & Längst, G.嘌呤-和嘧啶-三螺旋形成寡核苷酸在核糖体DNA位点上识别定性不同的靶点。gydF4y2Ba核糖核酸gydF4y2Ba24gydF4y2Ba, 371-380(2018)。gydF4y2Ba
李志强,姜丹丹,何阳。FRIGIDA建立了一种局部染色体环境gydF4y2Ba开花位点cgydF4y2Ba信使rna生产。gydF4y2BaNat。植物gydF4y2Ba4 gydF4y2Ba, 836-846(2018)。gydF4y2Ba
海普沃斯,J.等。秋季表达的自然变异是主要的适应性决定因素gydF4y2Ba拟南芥方法gydF4y2Ba单体型。gydF4y2BaeLifegydF4y2Ba9gydF4y2Ba, e57671(2020)。gydF4y2Ba
钟,b.y.w等。RNA热开关调节白天生长gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2BaNat。植物gydF4y2Ba6gydF4y2Ba, 522-532(2020)。gydF4y2Ba
李,W.等。ein2介导的乙烯信号转译调控gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2Ba细胞gydF4y2Ba163gydF4y2Ba, 670-683(2015)。gydF4y2Ba
琼斯,j.d.g.等。转基因植物中转化、表达外源基因和转座子切除的有效载体。gydF4y2Ba转基因Res。gydF4y2Ba1gydF4y2Ba, 285-297(1992)。gydF4y2Ba
蔡森,M. J. & Tesler, G.利用基本局部比对逐次细化(BLASR)绘制单分子测序reads:应用和理论。gydF4y2BaBMC Bioinform。gydF4y2Ba13gydF4y2Ba, 238 (2012)gydF4y2Ba
Spitale, R. C.等。结构印迹在体内解码RNA调控机制。gydF4y2Ba自然gydF4y2Ba519gydF4y2Ba, 486-490(2015)。gydF4y2Ba
Do, C. B., Woods, D. A. & Batzoglou, S. CONTRAfold:没有基于物理模型的RNA二级结构预测。gydF4y2Ba生物信息学gydF4y2Ba22gydF4y2Ba, e90-e98(2006)。gydF4y2Ba
滨田,M., Kiryu, H., Sato, K., Mituyama, T. & Asai, K.使用广义质心估计器预测RNA二级结构。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 465-473(2009)。gydF4y2Ba
Thiel, b.c., Beckmann, i.c., Kerpedjiev, P. & Hofacker, i.l.基于2D的3D:使用forgi 2.0计算螺旋角和堆叠模式,forgi 2.0是一个以二级结构元素为中心的RNA Python库。gydF4y2BaF1000Res。gydF4y2Ba8 gydF4y2Ba, 287(2019)。gydF4y2Ba
Pedregosa, F.等人。Scikit-Learn: Python中的机器学习。gydF4y2Baj·马赫。学习。Res。gydF4y2Ba12gydF4y2Ba, 2825-2830(2011)。gydF4y2Ba
张杨,等。监测和调节mRNA结构的应激反应是冷休克适应的核心。gydF4y2Ba摩尔。细胞gydF4y2Ba70gydF4y2Ba, 274-286(2018)。gydF4y2Ba
Smola, m.j, Rice, g.m, Busan, S, Siegfried, n.a., Weeks, k.m .通过引物延伸和突变分析(SHAPE-MaP)分析选择性2’-羟基酰化,用于直接、通用和准确的RNA结构分析。gydF4y2BaProtoc Nat。gydF4y2Ba10gydF4y2Ba, 1643-1669(2015)。gydF4y2Ba
Lorenz, R.等人。维也纳娜娜包2.0。gydF4y2Ba算法摩尔生物学。gydF4y2Ba6gydF4y2Ba, 26(2011)。gydF4y2Ba
Box, M. S, Coustham, V., Dean, C. & Mylne, J. S.方案:一种简单的基于苯酚的96孔提取高质量RNA的方法gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2Ba工厂方法gydF4y2Ba7gydF4y2Ba, 7(2011)。gydF4y2Ba
吴,Z.等。定量调控gydF4y2Ba方法gydF4y2Ba通过协调的转录起始和延伸。gydF4y2Ba国家科学院学报美国gydF4y2Ba113gydF4y2Ba, 218-223(2015)。gydF4y2Ba
吴,Z.等。RNA结合蛋白RZ-1B和RZ-1C在发育过程中调控pre-mRNA剪接和基因表达起关键作用gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2Ba植物细胞gydF4y2Ba28gydF4y2Ba, 55-73(2016)。gydF4y2Ba
朱,P.等。gydF4y2Ba拟南芥gydF4y2Ba小核仁RNA监测核糖体生物发生过程中有效的前rrna处理。gydF4y2Ba国家科学院学报美国gydF4y2Ba113gydF4y2Ba, 11967-11972(2016)。gydF4y2Ba
朱春华,张海燕,张海燕。利用RNA纯化技术分离染色质。gydF4y2BaJ. Vis. Exp。gydF4y2Bahttps://doi.org/10.3791/3912gydF4y2Ba(2012)。gydF4y2Ba
Yang, M.等。完整的RNA结构揭示了体内mRNA结构介导的miRNA切割调控。gydF4y2Ba核酸测定。gydF4y2Ba48gydF4y2Ba, 8767-8781(2020)。gydF4y2Ba
Dowell, R. D. & Eddy, S. R.几种用于RNA二级结构预测的轻量级随机上下文无关语法的评估。gydF4y2BaBMC Bioinform。gydF4y2Ba5gydF4y2Ba, 71(2004)。gydF4y2Ba
蒋涛,王丽丽,张凯。树的对齐——一种树编辑的替代方法。gydF4y2Ba定理。第一版。科学。gydF4y2Ba143gydF4y2Ba, 137-148(1995)。gydF4y2Ba
戴根,K. E,李,t.w, Mathews, D. H. & Weeks, K. M.精确的形状定向RNA结构测定。gydF4y2Ba国家科学院学报美国gydF4y2Ba106gydF4y2Ba, 97-102(2009)。gydF4y2Ba
Buske, F. A. Bauer, D. C. Mattick, J. S. & Bailey, T. L.三重器:在基因组和转录组数据中检测核酸三重螺旋。gydF4y2Ba基因组Res。gydF4y2Ba22gydF4y2Ba, 1372-1381(2012)。gydF4y2Ba
确认gydF4y2Ba
这项工作由欧洲研究理事会资助(资助680324;惠康高级研究员(资助210654;C.D.),英国皇家学会教授(RP\R1\180002;生物技术和生物科学研究理事会(BB/L025000/1;Y.D.);及由研究所策略计划GRO (BB/J004588/1)及GEN (BB/P013511/1)发给Y.D.和C.D.gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
M.Y, C.D.和Y.D.构想了这项研究。M.Y, P.Z, C.D.和Y.D.写了论文。Q.L、P.Z.和R.B.进行了SHAPE探针和RNA提取。R.B.生成gydF4y2BaCOOLAIRgydF4y2Ba结构突变构建和转基因植物。P.Z.对结构突变体进行了表型分析、基因表达和遗传研究以及ChIRP分析。M.Y.和Y.Z.构建了RNA结构库。P.M.进行了三层EMSA实验。m.y和J.C.分析了测序数据。C.D.和Y.D.获得了资金。C.D.和Y.D.负责项目管理。C.D.和Y.D.监督了这项研究。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
John Innes Centre已经为本文描述的技术提交了一份名为Y.D, M.Y, J.C.和y.z的专利申请(LU501541)。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢Howard Chang、Chris Helliwell和其他匿名审稿人对本工作的同行评审所作的贡献。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
smStructure-seq可以准确探测全长RNA二级结构gydF4y2Ba在活的有机体内gydF4y2Ba.gydF4y2Ba
一个gydF4y2Ba,主机的HiFi读取百分比gydF4y2BaCOOLAIRgydF4y2Ba计算了我们的smStructure-seq库中的isoforms。~ 273.9亿总碱基(40,123,867原始读取)的gydF4y2BaCOOLAIRsgydF4y2Ba在温暖和两周冷(2W)条件下均获得。gydF4y2BabgydF4y2Ba同时,我们还获得了大约85.8亿碱基(1,317,882个原始reads)的18S rRNA,作为我们smStructure-seq文库的内部控制。完整的18S rRNA(长度1808 nt)系统发育结构根据我们的smStructure-seq生成的SHAPE反应性进行颜色编码(红色标记为SHAPE反应性> = 1;SHAPE反应性0.5-1,黄色标记;形状反应性< = 0.5用灰色标记;5 '端未解决的区域标记为灰色)。该表量化了18S rRNA系统发育结构和SHAPE反应之间的对应关系。在全长18S rRNA中,85.4%的核苷酸显示高gydF4y2Ba在活的有机体内gydF4y2Ba在我们的数据集中,SHAPE反应性对应于系统发育结构中的单链区域(真阳性),而70.1%的核苷酸表现为低gydF4y2Ba在活的有机体内gydF4y2BaSHAPE反应性对应于系统发育结构中的碱基配对区域(真阴性)。真阳性(85.4%)和真阴性(70.1%)信号都比我们之前基于illumina的短读方法高得多gydF4y2Ba41gydF4y2Ba.gydF4y2BacgydF4y2Ba,从突变率谱中得到的归一化SHAPE反应活性被绘制为I类gydF4y2BaCOOLAIRgydF4y2Ba温生长条件下的转录异构体。归一化的SHAPE反应性用颜色编码并显示在Y轴上。gydF4y2Ba
扩展数据图2 DaVinci构象分析管道及其验证。gydF4y2Ba
一个gydF4y2Ba,达芬奇构象分析管道。每一行都指向一个测序读取。红星表示突变,包括错配和缺失。在步骤1中,测序读取遵循以下规则进行位向量化:如果碱基是野生型,则为“0”,如果碱基是突变型,则为“1”。在步骤2中,SCFGgydF4y2Ba42gydF4y2Ba用于推导出最能代表每个突变谱的RNA结构。例如,给定序列“AUGGGAACCAUACCCAAAGGG”,位向量为“00011100001000111000”,生成法则(如步骤2所示)可以推导出步骤2所示的RNA结构,且不依赖于热力学参数。规则中的“|”表示产品规则之间的“或”逻辑。红色的“1”或字母表示突变信息或单链核苷酸。在步骤3中,将收集到的每个突变剖面的RNA结构转化为RNA结构元的数字矩阵并进行降维。然后,确定每个构象簇的代表性RNA结构。详细描述在方法部分。gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BaegydF4y2Ba,gydF4y2Ba在网上gydF4y2Ba(gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba),gydF4y2Ba在体外gydF4y2Ba(gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba)野生型序列(RRE)或突变型RRE61中HIV-1 Rev反应元件(RRE)区域的RNA构象图。gydF4y2BafgydF4y2Ba,由davinco测定的包含和不包含TPP配体的TenA RNA的RNA构象图。对折叠的rna进行了探测gydF4y2Ba在体外gydF4y2Ba并以20 (tp处理的rna):80(未tp处理的rna)的比例进行池化。gydF4y2BaggydF4y2Ba,类似于(gydF4y2BafgydF4y2Ba),但池化率为50 (tp处理的rna):50(未tp处理的rna)。详细讨论(gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BaggydF4y2Ba)在gydF4y2Ba补充讨论gydF4y2Ba.gydF4y2BahgydF4y2Ba, DaVinci检测到的每个集群的比例。比率由(gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba).gydF4y2Ba
扩展数据图3 DaVinci构象分析验证。gydF4y2Ba
cspA RNA在37°C下的davinco -determined RNA结构构象空间gydF4y2Ba33gydF4y2Ba(gydF4y2Ba一个gydF4y2Ba)或10°CgydF4y2Ba33gydF4y2Ba(gydF4y2BabgydF4y2Ba).红色矩形是开始密码子。详细的讨论已经开始gydF4y2Ba补充讨论gydF4y2Ba.gydF4y2Ba
图4高变RNA结构区鉴定。gydF4y2Ba
一个gydF4y2Ba,中温暖构象1的RNA结构模型(图;gydF4y2Ba2gydF4y2Ba).红色三角形表示对应的站点gydF4y2Ba方法gydF4y2BaTSS。该表是warm构象1的各个区域以及整体结构与之前报道的相似度比较gydF4y2Ba在体外gydF4y2Ba5gydF4y2BaRNA结构(gydF4y2BabgydF4y2Ba).拓扑相似度基于树对齐(TA)。gydF4y2Ba43gydF4y2Ba由RNAforester计算gydF4y2Ba35gydF4y2Ba.采用阳性预测值(positive predictive value, PPV)计算碱基配对相似度。gydF4y2Ba44gydF4y2Ba.红色的方块显示了两者之间剧烈的结构变化gydF4y2Ba在体外gydF4y2Ba而且gydF4y2Ba在活的有机体内gydF4y2BaRNA结构。gydF4y2BabgydF4y2Ba,之前报道的RNA结构模型gydF4y2Ba在体外gydF4y2Ba5gydF4y2BaII级。我RNA结构。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,中热构象2和3的RNA结构模型(图;gydF4y2Ba2gydF4y2Ba).TA和PPV的相似性比较分别列于表中。中心3′M结构域的拓扑和碱基配对相似性(粗体和斜体)最低gydF4y2Ba在体外gydF4y2BaII级。我而且warm conformations 1, 2 and 3.egydF4y2Ba,局部结构差异用-log进行测量gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba值)在30 nt滑动窗口中对这三种温构象的单链性进行双向方差分析检验。红色阴影区域表示暖构象1、2和3之间差异最大,即超变区。红色阴影区域对应于(gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba).H4、H5和H6螺旋的序列画为灰色矩形,灰色拱形表示螺旋的形成。gydF4y2BafgydF4y2Ba,局部结构差异用-log进行测量gydF4y2Ba10gydF4y2Ba(gydF4y2BaPgydF4y2Ba为暖比构象(warm conformation 3)与冷比构象(cold - conformation 3)单链性的t检验。红色阴影区域对应于(gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba).gydF4y2Ba
扩展数据图5自然变异增加结构上不同的丰度gydF4y2BaCOOLAIRgydF4y2Ba同种型。gydF4y2Ba
一个gydF4y2Ba,原理图gydF4y2Ba方法gydF4y2Ba而且gydF4y2BaCOOLAIRgydF4y2Ba基因结构。灰色方框为未翻译区域,黑色方框为外显子。II类外显子。iv在Var2-6(参考。gydF4y2Ba7gydF4y2Ba)以绿线表示。kb,千碱基。红色三角形表示gydF4y2Ba方法gydF4y2BaTSS。gydF4y2BabgydF4y2Ba, II类体内结构。iv在Var2-6线。gydF4y2BacgydF4y2Ba,热构象1的RNA结构模型如图所示。gydF4y2Ba4 bgydF4y2Ba第二类。第四从gydF4y2BabgydF4y2Ba.高变量区域显示在黑色方块中。gydF4y2Ba
扩展数据图6gydF4y2BaCOOLAIRgydF4y2BaII级。我我的结构在warm and cold-grown plants.
一个gydF4y2Ba,一类集成结构模型。我我在warm-grown plants generated from CentroidFold (--engine CONTRAfold –sampling) was coloured by the likelihood of single-strandedness. The red triangle indicates the site corresponding to方法gydF4y2BaTSS。数据采用PCA进行可视化处理。四种不同颜色的阴影指的是四个不同的领域。gydF4y2BabgydF4y2Ba,一类集成结构模型。Ii在寒冷生长的植物中。分析结果与(gydF4y2Ba一个gydF4y2Ba).gydF4y2BacgydF4y2Ba,第二类香农熵。我我在warm and cold were calculated from (一个gydF4y2Ba)及(gydF4y2BabgydF4y2Ba分别)。阴影的颜色是根据(gydF4y2Ba一个gydF4y2Ba)及(gydF4y2BabgydF4y2Ba分别)。gydF4y2BadgydF4y2Ba,第二类香农熵。我在warm and cold were calculated from (Fig.2gydF4y2Ba)和(图。gydF4y2Ba3.gydF4y2Ba).阴影根据图中的域进行着色。gydF4y2Ba2gydF4y2Ba)和(图。gydF4y2Ba3.gydF4y2Ba分别)。gydF4y2Ba
图7结构突变体mut-r对gydF4y2BaCOOLAIRgydF4y2Ba结构和gydF4y2Ba方法gydF4y2Ba表达调控。gydF4y2Ba
一个gydF4y2Ba,推导出WT、mut和mut-r中超变区域的原理图。红色三角形表示对应的地点gydF4y2Ba方法gydF4y2BaTSS。小WT和杂种上的突变位点用红色箭头表示。模型由(图。gydF4y2Ba4 gydF4y2Ba).mut-r中的红色阴影区域是插入序列,增加了H4-H6区域的碱基配对。gydF4y2BabgydF4y2Ba,未拼接和拼接的相对表达量gydF4y2Ba方法gydF4y2BaRT-qPCR检测基因型。所有RT-qPCR数据均以均数±标准差表示;N = 3个生物独立实验。独立的结构突变转基因株系被标记为#1和#2。gydF4y2BacgydF4y2Ba,箱形图显示在温暖条件下生长的转基因植物的开花时间,通过莲座叶数测量。中线表示中位数,方框边缘描绘第25和第75百分位,柱状延伸到最小值和最大值,“+”表示平均值。对每个T3混合株系的基因型群体进行分析,从左到右,n = 36、36、35和36。gydF4y2Ba
扩展数据图8gydF4y2BaCOOLAIRgydF4y2BaH4-H6结构上的高变量区域gydF4y2Ba方法gydF4y2Ba表达调控。gydF4y2Ba
一个gydF4y2Ba,类I和类II拼接后的RT-PCRgydF4y2BaCOOLAIRgydF4y2Ba在野生型(mut和WT)或转基因株系中,无论有无结构突变,均存在同种异构体gydF4y2Ba特克斯gydF4y2Ba背景(傻瓜-gydF4y2Ba特克斯gydF4y2Ba和WT -gydF4y2Ba特克斯gydF4y2Ba).gydF4y2Ba哥伦比亚大学gydF4y2Ba作为对照。左图为100bp DNA阶梯图。凝胶源数据参见补充图。gydF4y2Ba1gydF4y2Ba.gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba而且gydF4y2BafgydF4y2Ba,拼接的相对表达水平gydF4y2BaCOOLAIRgydF4y2Ba亚型,gydF4y2Ba方法gydF4y2BaRT-qPCR检测的相关基因型。对不同基因型的混合独立株系群体进行分析。gydF4y2BafgydF4y2Ba).gydF4y2BaegydF4y2Ba, II类染色质结合比例。我在无足轻重的人line under warm conditions relative to WT, assayed by RT-qPCR.ggydF4y2Ba等位基因特异性的相对表达水平gydF4y2Ba方法gydF4y2Ba在F1植物中的转录本来源于WT和结构突变转基因系的杂交。gydF4y2BahgydF4y2Ba,未拼接的相对表达量gydF4y2Ba方法gydF4y2BaRT-qPCR检测WT和结构突变体gydF4y2Ba星期五gydF4y2Ba背景及功能丧失(gydF4y2Ba星期五gydF4y2Ba)的背景。调整后的单因素方差分析gydF4y2BaPgydF4y2Ba在每个比较中表示的值。所有RT-qPCR数据(gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba)表示为平均值±s.d;N = 3个生物独立实验。独立结构突变的转基因株系分别为#1、#2和#3。gydF4y2Ba
扩展数据图9 EMSA测试的三层地层。gydF4y2Ba
一个gydF4y2Ba,嘧啶Y-RNA或嘌呤R-RNA在高变区形成的潜在三联体序列含量。沃森链DNA和克里克链DNA(用红色表示)通过沃森-克里克键相互配对。形成三重结构的低聚RNA (Y-RNA或R-RNA)以绿色显示,可以结合gydF4y2Ba方法gydF4y2Ba通过胡斯坦键的双链DNA。Y-RNA对应于gydF4y2BaCOOLAIRgydF4y2Ba序列含量相同的RNA, R-RNA对应gydF4y2Ba方法gydF4y2Ba序列含量相同的RNA。红色三角形表示对应的站点gydF4y2Ba方法gydF4y2BaTSS。gydF4y2BabgydF4y2Ba图中显示了末端标记为Cy5的DNA信号(红色)和末端标记为FAM的RNA信号(绿色)。DNA/RNA bw,彩色图像黑白投影;gydF4y2Ba方法gydF4y2BadsDNA围绕TSS, triplexator -predictgydF4y2Ba45gydF4y2Ba三层目标位置在gydF4y2Ba方法gydF4y2Ba超变量区域内的TSS(对应于扩展数据图中的红色阴影)。gydF4y2Ba4 e, fgydF4y2Ba和扩展数据图。gydF4y2Ba5度gydF4y2Ba);阴性对照,上游寡核苷酸序列gydF4y2Ba方法gydF4y2BaTSS(星号标记ssDNA寡核苷酸中的杂质);阳性对照,人rDNA增强子En3与lncRNA形成三重寡核苷酸序列gydF4y2Ba’gydF4y2Ba21gydF4y2Ba.当dsDNA:ssRNA的比例为1:1、1:2和1:4时,DNA-RNA三联体样品显示ssRNA浓度增加。对于阳性对照,三倍样品固定在1:4的比例。数据至少代表三个独立的实验。凝胶源数据参见补充图。gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
补充讨论包括关于smStructure-seq的优势和DaVinci方法的验证的详细信息。补充图1包含拼接的I类和II类RT-qPCR原始源图像gydF4y2BaCOOLAIRgydF4y2Ba以及EMSA的三联体形成。gydF4y2Ba
补充表1gydF4y2Ba
引物,适配器和参考序列。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
杨敏,朱鹏,奇马,J。gydF4y2Baet al。gydF4y2Ba体内单分子分析显示gydF4y2BaCOOLAIRgydF4y2BaRNA结构多样性。gydF4y2Ba自然gydF4y2Ba609gydF4y2Ba, 394-399(2022)。https://doi.org/10.1038/s41586-022-05135-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05135-9gydF4y2Ba
这篇文章被引用gydF4y2Ba
对lncRNA生物发生和功能的更高分辨率和体内理解gydF4y2Ba
自然方法gydF4y2Ba(2022)gydF4y2Ba
低温驱动下COOLAIR lncRNA的结构变化gydF4y2Ba
《自然分子细胞生物学gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba