摘要gydF4y2Ba
生长素的极性运输是植物所特有的,并协调植物的生长发育gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2 gydF4y2Ba.PIN- formed (PIN)生长素转运蛋白在质膜上表现出高度不对称的定位,并驱动生长素的极性转运gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba;然而,它们的结构和传输机制在很大程度上仍然未知。在这里,我们报道了三种向内的构象结构gydF4y2Ba拟南芥gydF4y2BaPIN1:载波状态,与天然生长素吲哚-3-乙酸(IAA)结合,并与极性生长素转运抑制剂络合gydF4y2BaNgydF4y2Ba-1-萘酞酸(NPA)。PIN1的跨膜结构域共享一个保守的NhaA折叠gydF4y2Ba5gydF4y2Ba.在衬底结合结构中,IAA由疏水堆积和氢键配合。NPA与IAA在细胞内口袋处竞争同一位点,但具有更高的亲和力。这些发现有助于我们理解pin的底物识别和转运机制,并为未来的定向生长素转运研究建立了框架,定向生长素转运是植物发育中最重要的过程之一。gydF4y2Ba
主要gydF4y2Ba
生长素在调节植物生长发育的几乎每个方面都起着关键作用gydF4y2Ba2 gydF4y2Ba,gydF4y2Ba6克ydF4y2Ba.生长素的一个显著特征是它的细胞间定向运输-生长素极性运输-使植物协调发育和适应外源信号gydF4y2Ba1克ydF4y2Ba.生长素出口商gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba由于它们的极性亚细胞定位决定了生长素的极性运输的方向性,并在生长素的不对称分布和植物发育中起着关键作用,它们对这一过程至关重要并受到了相当大的关注gydF4y2Ba7 gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba.在gydF4y2Ba答:芥gydF4y2BaPIN1到PIN8共有两个保守结构域,n端和c端结构域,并由一个不太保守的亲水环路连接gydF4y2Ba10gydF4y2Ba(扩展数据图。gydF4y2Ba1克ydF4y2Ba).根据亲水环的长度,pin可分为长pin (PIN1-PIN4和PIN7)、短pin (PIN5和PIN8)和中间pingydF4y2Ba8gydF4y2Ba(PIN6)。短的pin通常位于内质网,而长pin位于质膜上,受胞吞作用和循环利用的动态调节gydF4y2Ba8gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba.NPA是生长素输出和生长素极性转运的抑制剂,在建立生长素外排在生长素极性转运中的关键作用方面发挥了重要作用。NPA直接针对pingydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,但其作用机制尚不清楚。gydF4y2Ba
拟南芥gydF4y2BaPIN1和PIN2(也称为EIR1)是第一个被识别的pingydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba.gydF4y2Ba拟南芥gydF4y2BaPIN1广泛表达,在胚胎、根尖分生组织、维管组织以及器官发育中起着重要作用gydF4y2Ba8gydF4y2Ba,gydF4y2Ba19gydF4y2Ba.gydF4y2Bapin1gydF4y2Ba缺乏导致裸露的,针状的花序gydF4y2Ba9gydF4y2Ba.在本研究中,我们开始确定PIN1的结构,揭示底物识别的基础和NPA的抑制机制。gydF4y2Ba
pin1介导的HEK293F细胞生长素外排gydF4y2Ba
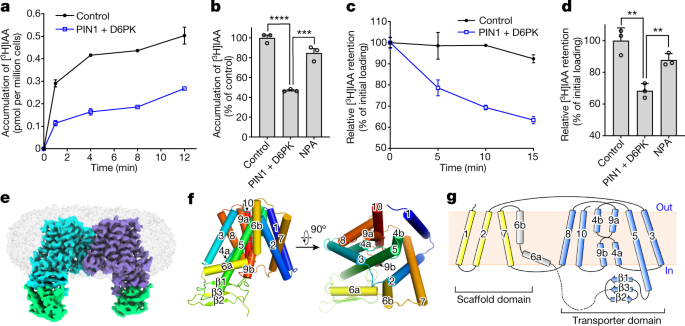
PIN的生长素外排活性已被证明使用gydF4y2Ba拟南芥gydF4y2Ba原生质体,烟草培养细胞,酵母,人类海拉细胞和gydF4y2Ba非洲爪蟾蜍gydF4y2Ba卵母细胞gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba14gydF4y2Ba.值得注意的是,在卵母细胞中,只有当PIN与D6蛋白激酶等激酶共表达时,才检测到生长素外排活性gydF4y2Ba20.gydF4y2Ba(D6PK)。我们表示gydF4y2Ba答:芥gydF4y2BaHEK293F细胞中的PIN1(扩展数据图;gydF4y2Ba2gydF4y2Ba)及其运输活动的特征。在负载试验中,共表达PIN1和D6PK的细胞积累的放射性同位素标记[gydF4y2Ba3.gydF4y2BaH]IAA与单独转染D6PK的细胞相比(图5)。gydF4y2Ba1 a, bgydF4y2Ba).在外排试验中,在将生长素转移到无同位素缓冲液后,随着时间的推移测量放射性标记的生长素,残留[gydF4y2Ba3.gydF4y2BaH]IAA在对照条件下基本保持不变,但在共表达PIN1和D6PK的细胞中IAA显著降低(图6)。gydF4y2Ba1 c, dgydF4y2Ba).值得注意的是,仅表达PIN1会导致[gydF4y2Ba3.gydF4y2BaH]与对照细胞相比,负载和流出实验中的IAA,但低于共表达D6PK的细胞(扩展数据图)。gydF4y2Ba2 b, cgydF4y2Ba).这些结果表明,PIN1在HEK293F细胞中表达时可介导生长素的主动外排,并进一步被D6PK激活。pin1介导的生长素外排被NPA抑制(图。gydF4y2Ba1 b, dgydF4y2Ba),与卵母细胞的结果相似gydF4y2Ba16gydF4y2Ba.gydF4y2Ba
一个gydF4y2Ba,负荷试验显示[gydF4y2Ba3.gydF4y2BaH]与只表达D6PK的对照细胞相比,同时表达PIN1和D6PK的细胞IAA降低。gydF4y2BabgydF4y2Ba,相对[gydF4y2Ba3.gydF4y2BaH]在无NPA或存在NPA的情况下,测定5分钟内IAA的积累。gydF4y2BacgydF4y2Ba,流出试验显示[gydF4y2Ba3.gydF4y2BaH]与对照细胞相比,共表达PIN1和D6PK的细胞IAA保留减少。gydF4y2BadgydF4y2Ba, (gydF4y2Ba3.gydF4y2BaH]在无NPA或存在NPA的情况下,IAA相对于初始负载的保留率,测量时间为10分钟。为每个点或结构进行三个独立的实验gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba.双尾未配对gydF4y2BatgydF4y2Ba以及。**gydF4y2BaPgydF4y2Ba对照与PIN1 + D6PK = 0.0041, **gydF4y2BaPgydF4y2Ba有NPA与无NPA = 0.0052, ***gydF4y2BaPgydF4y2Ba= 0.001, ****gydF4y2BaPgydF4y2Ba< 0.0001。数据为均数±标准差。gydF4y2BaegydF4y2Ba, PIN1的电子密度概述。PIN1原聚体对应的密度分别用青色和紫色表示。感染者被标为绿色。gydF4y2BafgydF4y2BaPIN1单体的整体结构。gydF4y2BaggydF4y2Ba, PIN1拓扑图。gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
通过磷酸化调节PIN1gydF4y2Ba
PIN极性和转运活性受多种蛋白激酶调控gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba.在长pin的亲水环中已经发现了几个磷酸化位点,包括PIN1中的三个苏氨酸残基(T227、T248和T286)和四个丝氨酸残基(S231、S252、S271和S290)gydF4y2Ba14gydF4y2Ba,gydF4y2Ba22gydF4y2Ba.我们生成了三种苏氨酸(分别为T3A和T3E)或四种丝氨酸(分别为S4A和S4D)的不可磷酸化和拟磷突变体,并检查了它们的转运活性(扩展数据图)。gydF4y2Ba二维gydF4y2Ba).与D6PK共表达时,非磷酸化T3A和S4A突变体的转运活性与野生型相比略有下降(扩展数据图)。gydF4y2Ba2 egydF4y2Ba).值得注意的是,在单独表达的拟磷突变体中,外排活性没有进一步增强(扩展数据图。gydF4y2Ba2 egydF4y2Ba),提示HEK293F细胞中D6PK对PIN1的激活可能是由于其他位点的磷酸化gydF4y2Ba20.gydF4y2Ba.质谱分析HEK293F细胞中与D6PK共表达的纯化野生型PIN1,鉴定出14个磷酸位点,包括已知的S252和S290位点(扩展数据表)gydF4y2Ba1克ydF4y2Ba).仅对PIN1表达的分析也鉴定出10个磷酸基,这可能有助于其微弱的外排活性(扩展数据表)gydF4y2Ba1克ydF4y2Ba).gydF4y2Ba
PIN1的结构测定gydF4y2Ba
纯化后的PIN1蛋白表现出良好的溶解行为(扩展数据图。gydF4y2Ba2 ggydF4y2Ba).为了便于冷冻电子显微镜(cro - em)分析,我们使用体外纳米体合成平台生成了合成纳米体(sybodies)gydF4y2Ba25gydF4y2Ba.在微尺度荧光实验中使用能增加PIN1热稳定性的联体gydF4y2Ba26gydF4y2Ba,我们制备了复杂样品,并进行了结构测定(扩展数据图。gydF4y2Ba3得了gydF4y2Ba).使用一个选定的sybody (Sybody-21)(扩展数据图。gydF4y2Ba2 hgydF4y2Ba),我们确定了PIN1的二聚体结构(图。gydF4y2Ba1 egydF4y2Ba和扩展数据图。gydF4y2Ba3 d jgydF4y2Ba).地图密度是高质量的,能够从头建立模型(扩展数据图。gydF4y2Ba3比gydF4y2Ba而且gydF4y2Ba4gydF4y2Ba).大多数亲水环缺失——可能是由于其固有的灵活性——并且没有报道的磷酸基可见,排除了磷酸化对PIN1调控的进一步结构分析。体结合在亲水环的近膜区(图。gydF4y2Ba1 egydF4y2Ba).我们还在3D分类过程中发现了一个单体PIN1(扩展数据图。gydF4y2Ba3 jgydF4y2Ba),然而,这类课程并没有产生高分辨率的地图。因此,我们主要对二聚PIN1结构进行了分析。gydF4y2Ba
PIN1的整体架构gydF4y2Ba
PIN1的跨膜结构域有十个跨膜片段(TM1到TM10),如先前报道的,N端和C端均位于细胞外gydF4y2Ba10gydF4y2Ba(无花果。gydF4y2Ba1 f, ggydF4y2Ba).PIN1采用NhaA折叠;这种褶皱首先在细菌钠中观察到gydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba逆向转运gydF4y2Ba27gydF4y2Ba和哺乳动物钠共有gydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba换热器gydF4y2Ba28gydF4y2Ba和根尖钠依赖性胆汁酸转运蛋白(ASBT)同源物,如ASBTgydF4y2Ba纳米gydF4y2Ba和ASBTgydF4y2BaYFgydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba.TM1到TM5和TM6到TM10形成两个倒置重复序列,可以很好地叠加(扩展数据图)。gydF4y2Ba5gydF4y2Ba).TM1、TM2、TM6和TM7形成支架结构域,TM3到TM5和TM8到TM10形成转运体结构域(图2)。gydF4y2Ba1克gydF4y2Ba).TM1, TM2和TM7组成二聚体界面,主要由疏水残基组成(扩展数据图。gydF4y2Ba5 b, cgydF4y2Ba).TM6是不连续的,其前半部分倾斜,几乎与膜表面平行。gydF4y2Ba1 fgydF4y2Ba).gydF4y2Ba
两个不连续的倒置螺旋——tm4和tm9在跨膜结构域的中心交叉;这种特征螺旋在asbt和Na的底物配位和输运中是必不可少的gydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba转运蛋白gydF4y2Ba28gydF4y2Ba(无花果。gydF4y2Ba2gydF4y2Ba).两个脯氨酸,TM4的P111和TM9的P579,分别位于交叉处,然后分别是一个天冬酰胺(N112)和谷氨酰胺(Q580)。gydF4y2Ba2gydF4y2Ba).值得注意的是,P579L突变gydF4y2Bapin1-4gydF4y2Ba等位基因被报道破坏PIN1的功能,证实了这些脯氨酸的重要作用gydF4y2Ba29gydF4y2Ba.在ASBT的内向结构中gydF4y2Ba纳米gydF4y2Ba,二纳gydF4y2Ba+gydF4y2Ba鉴定了两个结合位点,一个(Na1)位于交叉中心附近,另一个(Na2)位于细胞质附近gydF4y2Ba30.gydF4y2Ba(扩展数据图。gydF4y2Ba5 dgydF4y2Ba).NagydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba转运体在相似的位置上有带正电的残基,模仿钠离子的作用gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba(扩展数据图。gydF4y2Ba5 dgydF4y2Ba).在PIN1中,Na1和Na2位置没有密度,周围的残渣组成不同。一个带正电的残基R547存在于Na1位置,并与S108、A576和L578的骨架相互作用,从而稳定了TM4和TM9的交叉结构(图5)。gydF4y2Ba2 bgydF4y2Ba).Na2位点的中性电荷残基Q580与S106和L110的骨架相互作用(图5)。gydF4y2Ba2 bgydF4y2Ba).PIN1也不太可能在Na2位置结合钠,因为大多数极性或带负电荷的残基都存在于ASBT的相同位置gydF4y2Ba纳米gydF4y2Ba被疏水或中性带电残基所取代。gydF4y2Ba
TM5之后的一小段(残基165-211)形成了由三个β-链组成的细胞质结构域(图。gydF4y2Ba2摄氏度gydF4y2Ba).该结构域与邻近的跨膜片段紧密结合,包括TM5、TM6a和TM9b。β链主要通过疏水相互作用与TM5和TM6a结合,β1 -β2连接子通过盐桥与TM5和TM9b相互作用。TM5中的R471与β1 -β2环中的V177、D178和I181的主干相互作用。TM5中的K472与S183侧链相互作用,TM9b中的E590与R187相互作用(图。gydF4y2Ba2摄氏度gydF4y2Ba).gydF4y2Ba
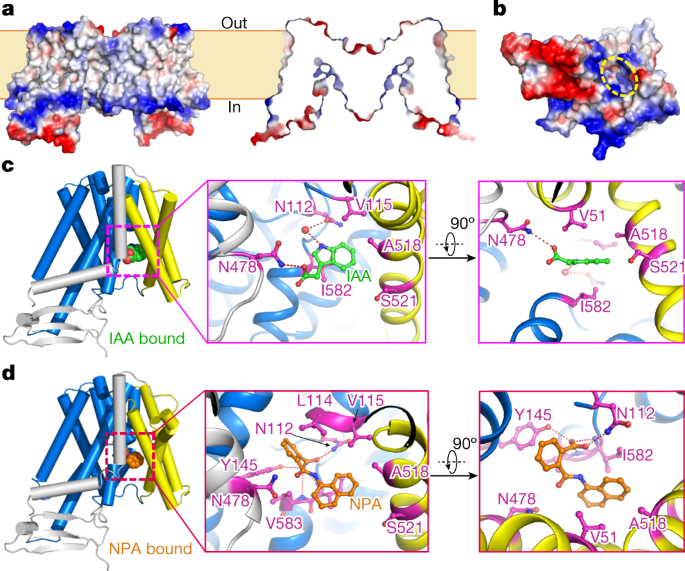
每个PIN1单体都有一个溶剂可达的腔体,该腔体向细胞内一侧具有弱正电位开口(图2)。gydF4y2Ba3 a, bgydF4y2Ba),暗示一种向内的状态。因此,PIN1更好地对准ASBT的内向结构gydF4y2Ba纳米gydF4y2Ba和ASBTgydF4y2BaYFgydF4y2Ba(扩展数据图。gydF4y2Ba5 egydF4y2Ba),而支架区域显示出较大的变化(扩展数据图。gydF4y2Ba5 egydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba, apo状态下PIN1表面静电势的侧视图和剖面图。负电荷和正电荷分别用红色和蓝色表示。gydF4y2BabgydF4y2Ba, PIN1单体胞内侧表面静电势概述。空腔用黄色虚线表示。gydF4y2BacgydF4y2Ba, PIN1协调IAA。IAA在整体视图中显示为球体(左侧)。TM1、TM2和TM7为黄色;TM6呈灰色,转运体结构域的跨膜段呈海蓝色。IAA分子和相互作用残基在放大图中显示为棒状(中间和右边)。氢键用红色虚线表示。gydF4y2BadgydF4y2Ba, PIN1协调NPA。在侧视图(左)和放大视图(中、右)中,NPA分别以球体和棒状显示。gydF4y2Ba
IAA映射到PIN1的胞内口袋gydF4y2Ba
在整个纯化过程和冷冻样品制备之前,由于IAA的存在,我们获得了总体分辨率为3.2的电子显微镜图Å(扩展数据图)。gydF4y2Ba3 h, jgydF4y2Ba).在细胞腔底部附近出现了一个额外的电镜密度,可以与IAA对接(扩展数据图)。gydF4y2Ba4 b, cgydF4y2Ba).在IAA和TM4的交叉区域之间也观察到一个小的密度,我们将其建模为水分子(扩展数据图)。gydF4y2Ba4 bgydF4y2Ba).整体的iaa结合结构与载脂蛋白状态基本相同(扩展数据图。gydF4y2Ba6gydF4y2Ba).IAA结合位点周围的残基在两种结构中几乎相同(扩展数据图)。gydF4y2Ba6 bgydF4y2Ba).在结构中,IAA通过氢键和疏水相互作用进行协调(图2)。gydF4y2Ba3 cgydF4y2Ba).IAA的羧基在TM6b中与N478相互作用。水分子是IAA咪唑环酰胺基与N112相互作用的桥梁。咪唑环被包裹在V51和I582之间,两者都位于环平面的4 Å范围内(图5)。gydF4y2Ba3 cgydF4y2Ba).所有相互作用的残基(V51, N112, N478和I582)在pin中高度保守(扩展数据图。gydF4y2Ba1克ydF4y2Ba).N112, N478和I582是不变的,而V51在PIN5和PIN8分别被亮氨酸和异亮氨酸取代。gydF4y2Ba
NPA映射到PIN1的胞内口袋gydF4y2Ba
我们还确定了PIN1的npa结合结构(扩展数据图。gydF4y2Ba3 i, jgydF4y2Ba).在细胞内袋的底部,我们发现了一个与NPA完全吻合的密度,它与IAA结合位点的位置相似(图2)。gydF4y2Ba3 dgydF4y2Ba和扩展数据图。gydF4y2Ba4 b、dgydF4y2Ba而且gydF4y2Ba6 fgydF4y2Ba).NPA结合结构与载脂蛋白结构基本相同,尽管NPA周围的残基略有改变,提供了更好的配位(扩展数据图)。gydF4y2Ba6 c, dgydF4y2Ba).NPA紧密协调(图;gydF4y2Ba3 dgydF4y2Ba):萘基排在V51和I582之间,羧基与N112和Y145的侧链以及I582和V583的主链原子形成氢键,苯环排在Y145、L114和V583之间。因此NPA保留了IAA与PIN1的结合模式,因为V51、N112和I582都参与了两个配体的结合。事实上,NPA和IAA具有化学相似性,都包含一个疏水环和一个带负电荷的基团(扩展数据图。gydF4y2Ba6 egydF4y2Ba).而NPA与PIN1之间的相互作用由于NPA中多了一个苯甲酸基团而包含了更多的氢键。值得注意的是,这一组突出到由TM5和交叉区域形成的口袋中(扩展数据图。gydF4y2Ba6克gydF4y2Ba).对于NhaA褶皱转运体,交叉区域被认为经历了局部重排,导致了完成运输周期的构象变化gydF4y2Ba28gydF4y2Ba.NPA的苯甲酸基团可能降低了交叉区迁移能力,从而使PIN1停留在向内构象。gydF4y2Ba
IAA和NPA结合PIN1gydF4y2Ba
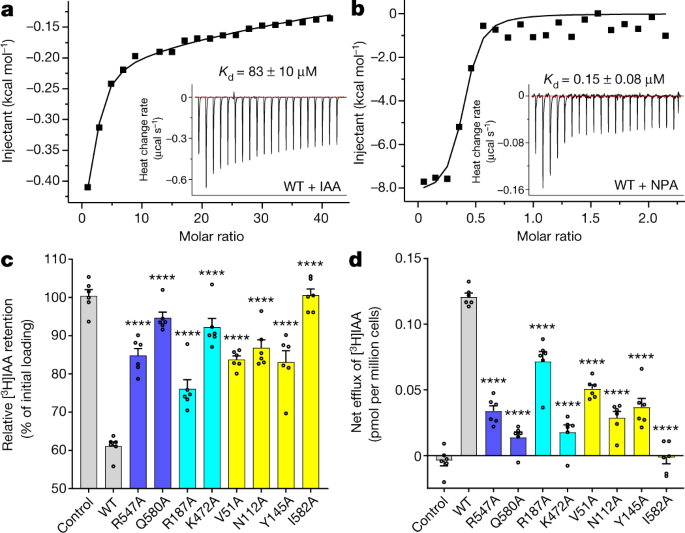
我们首先在生理细胞质pH (pH 7.0)下测试了IAA与PIN1的结合亲和力。我们确定了解离常数(gydF4y2BaKgydF4y2BadgydF4y2Ba等温滴定量热法(ITC) (83 μM)和表面等离子体共振法(SPR) (186 μM)(图2)gydF4y2Ba4gydF4y2Ba和扩展数据图。gydF4y2Ba7一个gydF4y2Ba).pH值为5.5时gydF4y2BaKgydF4y2BadgydF4y2BaITC和SPR分别为168 μM和268 μM(扩展数据图)。gydF4y2Ba7 a、bgydF4y2Ba),均在相似的范围内,且仅在pH 5.5时较pH 7.0时增加了约两倍,说明PIN1与IAA的结合受pH影响不大。PIN1还能结合其他天然生长素,包括吲哚-3-丁酸、吲哚-3-丙酸和4-氯吲哚-3-乙酸(扩展数据图)。gydF4y2Ba7一个gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba, ITC结果显示IAA在pH 7.0时与野生型PIN1结合。插图显示了原始的热变化记录。gydF4y2BabgydF4y2Ba, ITC结果表明NPA在pH 7.0时与野生型PIN1结合。gydF4y2BacgydF4y2Ba, HEK293F细胞野生型(WT)和突变PIN1的生长素转运特性研究。gydF4y2BadgydF4y2Ba, PIN1野生型和突变型IAA净外排。对每个结构进行6个独立实验gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba.Dunnett多重比较检验的单因素方差分析。****gydF4y2BaPgydF4y2Ba突变体与野生型的差异< 0.0001。数据为均数±s.e.m。gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
为了确认PIN1结构中底物结合位点的位置,我们生成了几个变体,并使用ITC检查了IAA结合(扩展数据图)。gydF4y2Ba7汉英gydF4y2Ba).N112和I582突变到丙氨酸阻止了IAA的结合(扩展数据图)。gydF4y2Ba7 d, egydF4y2Ba).的gydF4y2BaKgydF4y2BadgydF4y2BaV51A突变体为1.39 mM,约为野生型的17倍,表明结合亲和力明显降低(扩展数据图)。gydF4y2Ba7 cgydF4y2Ba).N478A突变对凝胶过滤中的表达水平和溶液行为有不利影响,阻碍了进一步的分析,表明N478在结构稳定方面有功能。gydF4y2Ba
我们还研究了NPA与野生型PIN1的结合。与IAA相比,NPA具有较高的亲和力gydF4y2BaKgydF4y2BadgydF4y2Ba约0.15 μM(图;gydF4y2Ba4 bgydF4y2Ba),与先前的迹象一致,NPA是一种有效的抑制剂,并表现出对植物膜整体成分的高亲和力结合gydF4y2Ba36gydF4y2Ba.NPA与N112A突变体之间的结合在很大程度上受损,这与N112参与IAA和NPA识别的结构观察相一致(扩展数据图)。gydF4y2Ba7 ggydF4y2Ba).NPA与Y145A突变体之间未观察到结合(扩展数据图)。gydF4y2Ba7小时gydF4y2Ba).值得注意的是,IAA与Y145A之间的结合也受到了损害(扩展数据图。gydF4y2Ba7 fgydF4y2Ba).Y145在运输过程中是否参与了IAA在其他状态下的直接结合,还是在维持关键结合位点上发挥了结构作用,还有待进一步研究。gydF4y2Ba
这些结合实验支持了结构研究预测的生长素结合位点,并证实了NPA在同一位点具有较高的亲和力。gydF4y2Ba
的IAA外排活动gydF4y2Bapin1gydF4y2Ba突变体gydF4y2Ba
我们的结构分析确定了可能对PIN1的稳定性和转运活性至关重要的残基,包括与Na位置相似的R547和Q580gydF4y2Ba+gydF4y2BaASBT中的结合位点gydF4y2Ba纳米gydF4y2Ba其中,R187、R471和K472带电残基介导胞质β-片结构域与跨膜段之间的相互作用,以及iaa配位残基V51、N112、N478和I582。我们生成了这些残基的丙氨酸突变体,并测量了它们的生长素外排活性。R547A和Q580A突变体明显增加[gydF4y2Ba3.gydF4y2BaH]IAA保留和净流出减少,表明运输活动严重受损(图。gydF4y2Ba4 c, dgydF4y2Ba和扩展数据图。gydF4y2Ba2gydF4y2Ba).这表明这些残基对PIN1的转运活动至关重要,可能是通过模仿Na的作用gydF4y2Ba+gydF4y2Ba在ASBTgydF4y2Ba纳米gydF4y2Ba稳定交叉螺旋。gydF4y2Ba
接下来,我们分析了介导胞质β-片和跨膜段之间相互作用的残基。突变R471到丙氨酸导致几乎检测不到表达(扩展数据图。gydF4y2Ba2gydF4y2Ba).R187A突变体表现出轻微的外排活性下降,而K472A表现出相当大的损伤(图。gydF4y2Ba4 c, dgydF4y2Ba).综上所述,这些结果表明该区域的结构完整性对运移活动至关重要。值得注意的是,该段的序列仅在长pin之间是保守的(扩展数据图。gydF4y2Ba1克ydF4y2Ba).其具体功能有待进一步研究。gydF4y2Ba
我们还利用V51A、N112A和I582A突变体表征了iaa配位残基的转运活性,但没有使用N478A突变体,后者表现出蛋白表达受损。V51A、N112A和I582A突变导致外排活性降低,尤其是I582A(图2)。gydF4y2Ba4 c, dgydF4y2Ba).与ITC的结果一致,Y145A突变体的IAA转运活性受到很大程度的损害(图1)。gydF4y2Ba4 c, dgydF4y2Ba),表明其参与了NPA抑制和IAA外排。与ITC的结果一起,观察到的结合袋代表了PIN1中一个重要的iaa协调位点。具体来说,PIN1的N112在asbt中是保守的,而在NagydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba转运蛋白,它被一个带负电荷的天冬氨酸或谷氨酸残渣所取代(扩展数据图。gydF4y2Ba5 dgydF4y2Ba).这加强了交叉螺旋在衬底识别中的关键作用,并为我们的PIN1结构提供了强大的功能支持。gydF4y2Ba
讨论gydF4y2Ba
在这里,我们描述了PIN1的结构和底物结合剖面,PIN1是中国主要的PIN生长素输出体gydF4y2Ba拟南芥gydF4y2Ba.在现有的向外和向内结构以及相应的功能分析的基础上,提出了一种具有特征的NhaA折叠运输机的升降式交替存取机构gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba.从这个角度来看,转运蛋白结构域在膜的一侧拾起底物,沿着支架结构域滑动发生构象变化,并将底物释放到膜的另一侧(扩展数据图)。gydF4y2Ba7我gydF4y2Ba).虽然类似的模型对于基于pin1的IAA传输是合理的,但确认该机制需要在传输周期中捕获更多的状态,特别是面向外部的结构。NPA和IAA直接竞争PIN1的胞内结合袋。由于NPA与PIN1之间具有较高的亲和力,一旦NPA被结合,即使在更高的浓度下,IAA也被禁止进入结合口袋(扩展数据图)。gydF4y2Ba7我gydF4y2Ba).gydF4y2Ba
pin1介导的生长素转运的能量来源仍然未知。由于IAA作为弱酸的化学性质,在不同的pH值下,其质子化或去质子化形式的比例有所不同。在生长素转运的化学渗透假说中,外质体中质子化的疏水IAAH容易通过脂质双分子层扩散。一旦进入中性或碱性的细胞内pH环境,它主要存在于脱质子的IAA中gydF4y2Ba−gydF4y2Ba形式和由pin等运输工具促进的输出是必不可少的gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba.我们的体内实验结果支持这一模型,表明没有PIN1表达,几乎不会发生IAA外排(图2)。gydF4y2Ba1 c, dgydF4y2Ba).质膜上相同的pH梯度被认为为生长素的输出提供了能量。然而,我们没有发现pH 5.5和6.5之间IAA的相对保留率有任何显著差异(扩展数据图。gydF4y2Ba2 fgydF4y2Ba).同样,我们没有检测到pH值对IAA与PIN1结合有太大影响(扩展数据图。gydF4y2Ba7 a、bgydF4y2Ba).这两个发现都表明PIN的转运可能不利用pH梯度。尽管如此,这需要进一步的验证,最好使用体外运输系统。gydF4y2Ba
总之,我们的研究为生长素外排和生长素极性转运机制提供了结构性的见解。他们为未来基于结构的pin功能分析和用于农业用途的生长素类似物设计建立了一个框架。gydF4y2Ba
方法gydF4y2Ba
蛋白表达及纯化gydF4y2Ba
全长的DNA序列gydF4y2Ba答:芥gydF4y2BaPIN1可在Uniprot (gydF4y2Bahttps://www.uniprot.orggydF4y2Ba),其登录码为Q9C6B8,并被克隆到pCAG载体(Invitrogen)中,带有n端Flag标签用于低温电镜样品制备或额外的c端AVI标签用于体选择。所有PIN1变体都是用N-terminal Flag标记以相同的方式生成的。HEK293F细胞(信和生物)转染PIN1质粒,转染密度为3 × 10gydF4y2Ba6克ydF4y2Ba所使用的HEK293F细胞系尚未经过验证,支原体污染检测为阴性。对于1-l细胞培养,1.5 mg质粒与4 mg线性聚乙烯亚胺(PEI) (polyciences)在50 ml培养基中预混合30分钟。然后将混合物添加到细胞培养中,孵育15分钟。转染细胞在37°C下培养60 h,然后以3000转/分的速度离心收集10 min。将微球重悬在含有25 mM HEPES、pH 7.4、150 mM NaCl、1.5% (w/v) DDM (Anatrace)和含有1mm苯甲基磺酰氟、抑肽酶(1.3 mg ml)的蛋白酶抑制剂鸡尾酒的裂解缓冲液中gydF4y2Ba−1gydF4y2Ba)、蛋白酶抑素3 (0.7 mg mlgydF4y2Ba−1gydF4y2Ba)和leupeptin (5 mg mlgydF4y2Ba−1gydF4y2Ba).4℃孵育2 h后,用14000转/分离心分离上清液1 h,用抗flag M2亲和凝胶(Sigma)在4℃孵育45 min。树脂用10 ml缓冲液冲洗3次,每次含有25 mM HEPES, pH 7.4, 150 mM NaCl, 0.06% (w/v)甘薯蓣皂苷元(Anatrace)。用洗涤缓冲液加200 μg ml洗脱蛋白gydF4y2Ba−1gydF4y2Ba标志肽。洗脱液用100 kda截断的Centricon (Millipore)浓缩,并使用Superose 6 Increase柱(GE Healthcare)在含有25 mM HEPES、pH 7.4、150 mM NaCl和0.02% (w/v) GDN的缓冲液中通过尺寸排除层析进一步纯化。为了测定IAA结合结构,从洗涤剂提取蛋白开始,整个过程中加入10mm IAA钠盐(Sigma)。凝胶过滤缓冲液含有10 mM IAA钠盐,25 mM HEPES, pH 7.4, 150 mM NaCl和0.02% (w/v) GDN。gydF4y2Ba
细胞生长素运输试验gydF4y2Ba
答:芥gydF4y2Ba将PIN1的野生型或突变体D6PK (Uniprot登录码:Q9FG74)分别亚克隆到pCAG载体中。HEK293F细胞密度为1.5 × 10gydF4y2Ba6克ydF4y2Ba每毫升细胞均转染空载体或PIN1构建物,或以3:1的质量比共转染PIN1和D6PK。转染24小时后,离心收集细胞,重悬进行负载试验或流出试验。对于PIN1突变体的所有特征,除非另有说明,细胞均与D6PK共转染。细胞计数用Coulter计数和显微镜观察确定。对于所有检测系统,500 μl不等的细胞悬液含有3 × 10gydF4y2Ba6克ydF4y2Ba细胞。对于[gydF4y2Ba3.gydF4y2BaH]IAA积累试验,细胞重悬并在37°C下与PBS柠檬酸缓冲液孵育,pH 5.5 (10 mM NagydF4y2Ba2 gydF4y2BaHPOgydF4y2Ba4gydF4y2Ba, 1.8 mM KHgydF4y2Ba2 gydF4y2Ba阿宝gydF4y2Ba4gydF4y2Ba, 2.7 mM KCl, 137 mM NaCl, pH由无水柠檬酸调整),含16 nM [gydF4y2Ba3.gydF4y2BaH]IAA(比活性25 Ci mmolgydF4y2Ba−1gydF4y2Ba美国放射性标记化学品)。在指定的时间点通过离心停止加载过程。然后用pH为7.4的冰冷PBS缓冲液清洗细胞两次,并用相同的缓冲液加1% Triton X-100重悬以进行细胞裂解。用液体闪烁计数法(Tri-Carb 2910TR, PerkinElmer)测定细胞裂解过程中的放射性。为了进行生长素外排实验,细胞首先被加载在pH为5.5的PBS柠檬酸缓冲液中,外加40 nM [gydF4y2Ba3.gydF4y2BaH]IAA浸泡5分钟,然后用[gydF4y2Ba3.gydF4y2BaH]不含iaa的PBS柠檬酸缓冲液。重新悬浮后(定义为零时间点)或在其他指定时间点立即服用500 μl。将细胞离心,用1ml冰冷的PBS缓冲液(pH值7.4)洗涤两次,用相同的缓冲液加1% Triton X-100重悬进行闪烁计数。对于PIN1突变体,[gydF4y2Ba3.gydF4y2BaH]IAA保留率表现为相对于0分钟测量的10分钟剩余放射性,净流出量表示为放射性标记的量[gydF4y2Ba3.gydF4y2BaH]IAA测量在0分钟减去保留[gydF4y2Ba3.gydF4y2BaH]IAA在10分钟测量。对于NPA抑制实验,细胞用[gydF4y2Ba3.gydF4y2BaH] 10 μM NPA存在时的IAA (Sigma)。gydF4y2Ba
PIN1的生物素化用于体选择gydF4y2Ba
纯化PIN1 (1.3 mg mlgydF4y2Ba−1gydF4y2Ba用BirA生物素化(66 μg ml)gydF4y2Ba−1gydF4y2Ba),在缓冲液中加入150 mM NaCl, 0.02% (w/v) GDN, 10 mM乙酸镁,5 mM ATP, 28 μM生物素和25 mM HEPES pH 7.4,温度为4℃。反应16 h后,加入1 mM ATP和13 μg ml继续进行生物素化gydF4y2Ba−1gydF4y2Ba再注射5小时。将反应混合物与Ni-NTA树脂孵育以去除his标记的BirA。通过凝胶过滤在Superose 6 Increase 10/300 GL色谱柱上进行分离(GE Healthcare)。含有PIN1的馏分浓缩并以小比例在液氮中快速冷冻,并在−80°C保存后使用。gydF4y2Ba
杂交体的体外转译和选择gydF4y2Ba
在体外翻译sybodies时,9.3 μl的混合物(PUREfrex 2.1 kit, Genefrontier)含有1.8 μl无核酸酶水,4 μl溶液I, 0.5 μl溶液II, 1 μl溶液III, 0.5 μl 10 mM半胱氨酸,0.5 μl 80 mM还原性谷胱甘肽,0.5 μl 60 mM氧化性谷胱甘肽,0.5 μl 1.875 mg mlgydF4y2Ba−1gydF4y2Ba二硫异构酶DsbC (Genefrontier)首先在PCR循环器中37°C孵育5分钟。加入0.7 μl的sybody mRNA文库启动反应。37°C孵育30 min后,加入100 μl陈冰洗净液(0.02% GDN, 150 mM NaCl, 50 mM醋酸镁,0.5% BSA, 0.5%肝素,1 μl RnaseIn, 50 mM Tris-acetate pH 7.4), 20000离心gydF4y2BaggydF4y2Ba在4°C下加热5分钟。将上清液与50 nM生物素化的PIN1混合,在冰上孵育20分钟,进行溶液筛选。将混合物加入12 μl链霉亲和素珠(Dynabeads Myone Streptavidin T1)中,与WTB缓冲液(50 mM Tris-acetate pH 7.4, 150 mM NaCl, 50 mM acetate镁)预平衡,并添加0.5% BSA孵育10 min。用WTB- d缓冲液(WTB缓冲液中GDN为0.008%)洗涤三次,再用RD洗脱液(100 μg ml)洗脱gydF4y2Ba−1gydF4y2Ba酵母RNA, 150 mM NaCl, 50 mM EDTA, 50 mM Tris-acetate pH 7.4)。用Rneasy Kit (Qiagen)纯化洗脱后的mRNA,用引物(5’-CTTCAGTTGCCGCTTTCTTTCTTG-3’)和2 μl RNA转录酶(Agilent)在40 μl的反应中进行逆转录。cDNA纯化(machery - nagal)并使用引物对进行pcr扩增(5 ' -ATATGCTCTTCTAGTCAGGTTCAGCTGGTTGAGAGCG-3 '和5 ' - tatagctcttcatgcgctcacagtcacttgggttac -3 '用于凹库和环库,5 ' -ATATGCTCTTCTAGTCAAGTCCAGCTGGTGGAATCG-3 '和5 ' -TATAGCTCTTCATGCAGAAACGGTAACTTGGGTGCCC-3 '用于凸库。扩增后的DNA产物用IIs型酶切酶BspQI纯化消化,并连接到用同样酶处理过的载体pDX_init上。将结扎产品改造成gydF4y2Ba大肠杆菌gydF4y2Ba电穿孔SS320感受态细胞生成噬菌体展示库。gydF4y2Ba
在越来越严格的条件下,用交替珠进行了三轮噬菌体展示。第一轮在96孔镀67 nM neutravidin(赛默飞世尔科学公司)中进行。纯化后的噬菌体与50 nM生物素化PIN1在室温(20 - 22°C)下孵育20分钟,然后加入96孔板。用TBS-D缓冲液(0.01% GDN, 150 mM NaCl, 20 mM Tris-HCl pH 7.4)洗涤三次后,用0.25 mg ml洗脱噬菌体颗粒gydF4y2Ba−1gydF4y2Ba胰蛋白酶。洗脱后的噬菌体颗粒被扩增并应用于第二轮噬菌体展示。将12 μl MyOne Streptavidin C1珠与50 nM生物素化的PIN1和噬菌体颗粒在100 μl摇盘液中孵育10 min。用5 μM的非生物素化PIN1在室温下孵育3分钟,以挑战较弱的粘结剂。噬菌体展示过程以与第二轮相同的方式重复,除了生物素化和非生物素化PIN1的浓度降低了10倍。利用片段交换(FX)克隆技术将噬菌体中的符号编码片段交换到pSb_init载体中,进行进一步的酶联免疫吸附试验gydF4y2Ba42gydF4y2Ba.gydF4y2Ba
酶联免疫吸附试验gydF4y2Ba
将上述得到的pSb_init质粒转化为gydF4y2Ba大肠杆菌gydF4y2BaMC1061活性细胞。单菌落接种,在96孔板中37°C培养4 h。然后将预培养物播种到1 ml Terrific肉汤中(1.2%色氨酸,2.4%酵母提取物,1% NaCl, 4 ml甘油,0.231% KH)gydF4y2Ba2 gydF4y2Ba阿宝gydF4y2Ba4gydF4y2Ba, k为1.254%gydF4y2Ba2 gydF4y2BaHPOgydF4y2Ba4gydF4y2Ba),添加25 μg mlgydF4y2Ba−1gydF4y2Ba氯霉素,37℃培养。2 h后,温度降至22℃。再用0.02% (w/v) L-(+)-阿拉伯糖诱导半体表达17 h。收集细胞微球,用TES缓冲液(0.5 μg ml)裂解gydF4y2Ba−1gydF4y2Ba溶菌酶,0.5 mM EDTA, 20% (w/v)蔗糖,50 mM Tris-HCl pH 8.0)。在TBS缓冲液(150 mM NaCl, 20 mM Tris-HCl pH 7.4)中进一步培养,并添加1mm MgClgydF4y2Ba2 gydF4y2Ba在4°C下,以3220 g离心30 min。用含联体的上清部分进行ELISA检测。gydF4y2Ba
将max - sorp培养皿(Thermo Fisher Scientific)与蛋白A在PBS中4°C孵育16小时。去除多余的蛋白A,用TBS缓冲液清洗一次,用添加0.5% BSA的TBS堵塞,用TBS清洗三次。在TBS-BSA- d缓冲液(TBS中添加0.5% (w/v) BSA和0.02% GDN)中以1:2 000稀释的抗myc抗体(Sigma)加入96孔板与蛋白a结合,多余的抗体用TBS- d缓冲液(TBS中添加0.01% GDN)洗涤三次。将上述10微升的周浆提取物加入96孔板,室温孵育20分钟。然后用TBS-D缓冲液清洗三次。在培养皿中加入50纳摩尔生物素化的PIN1或对照蛋白。室温孵育20 min后,用TBS-D缓冲液洗涤三次。将链霉亲和素与辣根过氧化物酶(HRP)结合添加到每个孔中,在室温下孵育20分钟。用TBS-D缓冲液洗涤三次,然后用ELISA显影缓冲液(51 mM Na)显影gydF4y2Ba2 gydF4y2BaHPOgydF4y2Ba4gydF4y2Ba, 24 mM柠檬酸,0.006% (v/v) HgydF4y2Ba2 gydF4y2BaOgydF4y2Ba2 gydF4y2Ba, 0.1 mg mlgydF4y2Ba−1gydF4y2Ba3, 3, 5, 5 ' -tetramethylbenzidine)。ELISA信号在平板阅读器650 nm处用吸光度法检测。gydF4y2Ba
生物层干涉测定法gydF4y2Ba
PIN1与sybody的结合亲和力在30°C下使用HIS1K生物传感器用Octet RED96系统(ForteBio)进行BLI测量。传感器首先在动能缓冲液(25 mM HEPES pH 7.4, 150 mM NaCl, 0.01% GDN)中平衡10分钟。对于BLI测量,记录基线相位120 s。将传感器浸泡在2 μg ml中监测加载相gydF4y2Ba−1gydF4y2Ba在动力缓冲液中标记了他的躯体。通过将传感器浸泡在不同浓度的PIN1分析物中360秒来监测结合,并通过将传感器浸泡在无PIN1的缓冲液中600秒来记录解离相。数据拟合为1:1的化学计量平衡解离常数gydF4y2BaKgydF4y2BaDgydF4y2Ba,关联速率常数gydF4y2BaKgydF4y2Ba在gydF4y2Ba,解离速率常数gydF4y2BaKgydF4y2Ba从gydF4y2Ba使用Octet数据分析软件9.0版本进行计算。gydF4y2Ba
微尺度荧光热稳定性试验gydF4y2Ba
NgydF4y2Ba-(4-(7-二乙基氨基-4-甲基-3-香豆素基)苯基)马来酰亚胺(CPM, Sigma)法测定PIN1与不同联体的热稳定性。将CPM染料以4mg ml溶解在二甲基亚砜(DMSO)中gydF4y2Ba−1gydF4y2Ba使用前将其稀释40倍至含有25 mM HEPES pH 7.4, 150 mM NaCl, 0.025% DDM的缓冲液中。将两微克纯化的PIN1蛋白与子体(1:4摩尔比)混合,在冰上孵育30分钟。将稀释后的CPM (1.5 μl)加入蛋白中,置于冰中,在黑暗中充分混合。反应混合物(共20 μ l)受控加热,升温速率为3°C mingydF4y2Ba−1gydF4y2Ba在LightCycler480 (Roche)实时PCR仪中。激发波长设置为440 nm,发射波长设置为488 nm,连续测量荧光强度。在从室温到90°C的温度范围内进行测定。gydF4y2Ba
低温电镜样品制备和数据采集gydF4y2Ba
为了制备PIN1和sybody (sybody-21用于最终结构测定)配合物,将纯化后的PIN1和sybody-21以~1:3的摩尔比混合后注入凝胶过滤。未对纯化的体进行稀释。对于apo或iaa结合的PIN1冷冻- em样品制备,将等分(4 μ l)纯化的蛋白质添加到辉光放电的孔碳网格(Quantifoil Au R1.2/1.3, 300目)中,用Vitrobot Mark IV (Thermo Fisher Scientific)在8°C下100%湿度下用4 s时间进行印迹,并浸入液氮冷却的液态乙烷中。为了制备NPA结合PIN1的低温电镜样品,首先将纯化后的PIN1与终浓度为100 μM的NPA (Sigma)在室温下孵育30 min,然后应用于网格。栅格被加载到300kv的Titan Krios (FEI)电子显微镜中,配备了生物量子能量过滤器和K2或K3直接电子探测器(Gatan)。图像记录采用EPU或SerialEM超分辨率模式。离焦值范围为−1.5 ~−2.3 μm。图像堆栈的曝光时间为3秒,剂量分为32帧,总剂量为50 egydF4y2Ba−gydF4y2Ba一个gydF4y2Ba−2gydF4y2Ba.gydF4y2Ba
图像处理gydF4y2Ba
在扩展数据图中给出了数据处理的流程图。gydF4y2Ba3 jgydF4y2Ba.使用RELION 3.1实现的MotionCor2进行运动校正和剂量加权gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba.用CTFFIND4估计离焦值gydF4y2Ba45gydF4y2Ba.在人工检查后,使用RELION选择良好的显微照片进行自动颗粒挑选。提取采摘颗粒并应用于二维分类。然后从好的级别的粒子导入到cryoSPARC (v.3.2.0)gydF4y2Ba46gydF4y2Ba用于进一步的数据处理。在载脂蛋白结构分析中,通过从头算三维重建将粒子分为五类。两类分别代表二聚体和单体PIN1,被选择进行非均匀细化。经过非均匀细化,在3.7 Å获得了含有919,145个粒子的二聚体形式的EM图。使用三个手动生成的低通过滤参考的异构细化和随后的非均匀细化,得到了一个总体分辨率为3.3 Å的映射,包含302,862个粒子。进一步的对比度传递函数细化,从头分类和非均匀细化使用cryoSPARC进一步提高了分辨率。最后,从210,597个粒子中获得了PIN1载apo二聚态的总体分辨率为3.1 Å的地图,这是用金标准傅里叶壳相关在0.143准则下的高分辨率噪声替代方法估计的gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba.使用ResMap估计局部分辨率变化gydF4y2Ba49gydF4y2Ba.对于单体状态,从257,731个粒子中获得了总体分辨率为5.9 Å的最终EM地图。对于iaa结合的二聚态的数据处理,进行了类似的程序,并使用250,009个粒子获得了总体分辨率为3.2 Å的EM图。从头开始三维重建时未观察到PIN1单体状态对应的类。对于npa结合的二聚态的数据处理,进行了类似的程序,并使用215,094个粒子获得了总体分辨率为3.2 Å的EM图。gydF4y2Ba
模型构建和细化gydF4y2Ba
载脂蛋白、二聚体PIN1的3.1 Å图用于COOT从头建立模型gydF4y2Ba50gydF4y2Ba.以Phe、Tyr、Trp和Arg等大残基为指导序列分配,并考虑氨基酸的化学性质,便于建模。通过PHENIX在真实空间中进行结构细化gydF4y2Ba51gydF4y2Ba.模型的过拟合是通过在两个独立地图中的一个中根据金标准的细化方法进行细化,并将细化后的模型与另一个地图进行测试来监测的。改进后的模型分别停靠到IAA或npa结合的二聚物PIN1的地图中,并由PHENIX进一步改进。三维重建和模型细化的统计数据可以在扩展数据表中找到gydF4y2Ba2 gydF4y2Ba.gydF4y2Ba
等温滴定量热法gydF4y2Ba
用MicroCal iTC200微热量计(MicroCal)测定IAA和PIN1变体之间的结合亲和力。在含有0.02% GDN、25 mM HEPES pH 7.0(或MES pH 5.5)和150 mM NaCl的缓冲液中纯化的野生型或突变型PIN1浓缩至0.05 mM左右进行等温滴定量热法滴定。蛋白用溶解在与25°C粒径排除层析所用缓冲液相同的5 mM IAA滴定。为了测定NPA与PIN1的结合亲和力,在含有1% DMSO、0.02% GDN、25 mM HEPES pH 7.0和150 mM NaCl的缓冲液中,用100 μM NPA滴定至0.01 mM的纯化蛋白。数据采用Origin 7.0 (MicroCal)软件拟合。gydF4y2Ba
表面等离子体共振gydF4y2Ba
在Biacore 8K体系(Cytiva)上进行IAA、吲哚-3-丁酸(IBA)、吲哚-3-丙酸(IPA)和4-氯吲哚-3-乙酸(4-Cl-IAA)的SPR实验,温度为25℃,流速为30 μl mingydF4y2Ba−1gydF4y2Ba.通过胺偶联化学将纯化的野生型PIN1蛋白固定在系列S CM5传感器芯片(Cytiva)上。不同浓度的配体在pH 7.0缓冲液(25 mM HEPES, pH 7.0, 150 mM NaCl, 0.01% GDN, 2% DMSO)或pH 5.5缓冲液(25 mM MES, pH 5.5, 150 mM NaCl, 0.01% GDN, 2% DMSO)中流过芯片表面。数据采用Biacore Insight Evaluation Software Version 3.0.12,采用稳态亲和绑定模型进行分析。gydF4y2Ba
蛋白质序列和坐标gydF4y2Ba
序列为8引脚和D6PK在gydF4y2Ba答:芥gydF4y2Ba均可在Uniprot (gydF4y2Bahttps://www.uniprot.orggydF4y2BaPIN1: Q9C6B8、PIN2: Q9LU77、PIN3: Q9S7Z8、PIN4: Q8RWZ6、PIN5: Q9FFD0、PIN6: Q9SQH6、PIN7: Q940Y5、PIN8: Q9LFP6、D6PK: Q9FG74。ASBT和NapA的坐标可在蛋白质数据库(PDB)中公开获取,并具有以下登录代码gydF4y2Ba纳米gydF4y2Ba: 3zux, asbtgydF4y2BaYFgydF4y2Ba: 4N7W和NapA: 5BZ2。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
apo, iaa结合和npa结合的二聚态的三维冷冻- em密度图gydF4y2Ba答:芥gydF4y2BaPIN1已按登录号存入电子显微镜数据库gydF4y2Baemd - 33691gydF4y2Ba,gydF4y2Baemd - 33693gydF4y2Ba而且gydF4y2Baemd - 33692gydF4y2Ba,分别。apo、iaa结合和npa结合结构模型的坐标已根据登录代码存入蛋白质数据库(PDB)gydF4y2Ba7 y9tgydF4y2Ba,gydF4y2Ba7 y9vgydF4y2Ba而且gydF4y2Ba7 y9ugydF4y2Ba,分别。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
参考文献gydF4y2Ba
范内斯特,S. &弗里ml, J.生长素:植物发育变化的触发器。gydF4y2Ba细胞gydF4y2Ba136gydF4y2Ba, 1005-1016(2009)。gydF4y2Ba
植物生长素运输的回溯视角。gydF4y2Ba前面。植物科学。gydF4y2Ba12gydF4y2Ba, 756968(2021)。gydF4y2Ba
Petrasek, J.等。PIN蛋白在细胞生长素外排中起限速作用。gydF4y2Ba科学gydF4y2Ba312gydF4y2Ba, 914-918(2006)。gydF4y2Ba
周建军,罗建军。植物中PIN-FORMED生长素外排载体的研究。gydF4y2BaInt。理学。gydF4y2Ba19gydF4y2Ba, 2759(2018)。gydF4y2Ba
石勇。次生主动转运体的常见褶皱及转运机制。gydF4y2Ba为基础。启Biophys。gydF4y2Ba42gydF4y2Ba, 51-72(2013)。gydF4y2Ba
十四站生长素。gydF4y2Ba冷泉港。教谕。医学杂志。gydF4y2Ba14gydF4y2Ba, a039859(2022)。gydF4y2Ba
Wisniewska, J.等人。极性PIN定位指导植物生长素的流动。gydF4y2Ba科学gydF4y2Ba312gydF4y2Ba, 883(2006)。gydF4y2Ba
Krecek, P.等人。生长素转运蛋白的PIN- formed (PIN)蛋白家族。gydF4y2Ba基因组医学杂志。gydF4y2Ba10gydF4y2Ba, 249(2009)。gydF4y2Ba
Adamowski, M. & Friml, J. pin依赖的生长素运输:作用,调控和进化。gydF4y2Ba植物细胞gydF4y2Ba27gydF4y2Ba, 20-32(2015)。gydF4y2Ba
Nodzynski, T.等人。等离子膜定位PIN生长素转运组分的拓扑结构探讨。gydF4y2Ba摩尔。植物gydF4y2Ba9gydF4y2Ba, 1504-1519(2016)。gydF4y2Ba
Mravec, J.等人。植物生长素的亚细胞稳态是由er定位的PIN5转运体介导的。gydF4y2Ba自然gydF4y2Ba459gydF4y2Ba, 1136-1140(2009)。gydF4y2Ba
西蒙,S.等。内质网和质膜上的PIN6生长素转运体介导植物生长素的稳态和器官发生gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2Ba新植醇。gydF4y2Ba211gydF4y2Ba, 65-74(2016)。gydF4y2Ba
Kleine-Vehn, J.等人。植物中极性PIN靶向和转胞作用的细胞和分子要求。gydF4y2Ba摩尔。植物gydF4y2Ba1克ydF4y2Ba, 1056-1066(2008)。gydF4y2Ba
Barbosa, i.c.r, Hammes, U. Z. & Schwechheimer, C. PIN-FORMED生长素转运蛋白的磷酸化激活和极性控制。gydF4y2Ba植物科学gydF4y2Ba23gydF4y2Ba, 523-538(2018)。gydF4y2Ba
蒂尔,W. D.等人。黄酮醇介导的PIN外排复合物的稳定调节生长素的极性运输。gydF4y2BaEMBO J。gydF4y2Ba40gydF4y2Ba, e104416(2021)。gydF4y2Ba
阿巴斯,L.等。萘酞酸与生长素转运蛋白结合并抑制。gydF4y2Ba国家科学院学报美国gydF4y2Ba118gydF4y2Ba, e2020857118(2021)。gydF4y2Ba
Luschnig, C., Gaxiola, R. A., Grisafi, P. & Fink, G. R. EIR1,一种参与生长素运输的根特异性蛋白,是植物向地性所必需的gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2BaDev的基因。gydF4y2Ba12gydF4y2Ba, 2175-2187(1998)。gydF4y2Ba
Galweiler, L.等。调节生长素的极性运输gydF4y2Ba在gydF4y2BaPIN1在gydF4y2Ba拟南芥gydF4y2Ba维管组织。gydF4y2Ba科学gydF4y2Ba282gydF4y2Ba, 2226-2230(1998)。gydF4y2Ba
Omelyanchuk, n.a.等人。PIN1生长素转运体的详细表达图gydF4y2Ba拟南芥gydF4y2Ba根。gydF4y2BaBMC植物生物学。gydF4y2Ba16gydF4y2Ba, 5(2016)。gydF4y2Ba
Zourelidou, M.等。pinin - shaped蛋白的生长素外排由两种不同的蛋白激酶D6蛋白激酶和PINOID蛋白激活。gydF4y2BaeLifegydF4y2Ba3.gydF4y2Ba, e02860(2014)。gydF4y2Ba
多莉,M.等人。共同进化的MAPK和PID磷酸化位点表明,陆地植物中PIN生长素转运蛋白存在古老的环境控制。gydF4y2Ba2月。gydF4y2Ba592gydF4y2Ba, 89-102(2018)。gydF4y2Ba
谭淑娟,吕希尼格,陈志勇,李志刚。植物生长素生物合成、转运和信号通路中蛋白磷酸化的研究进展。gydF4y2Ba摩尔。植物gydF4y2Ba14gydF4y2Ba, 151-165(2021)。gydF4y2Ba
Friml, J.等。根尖-基底PIN极性靶向中一个依赖于pinoid的二元开关引导生长素外排。gydF4y2Ba科学gydF4y2Ba306gydF4y2Ba, 862-865(2004)。gydF4y2Ba
Hajny, J.等。受体激酶模块在通道化过程中靶向pin依赖的生长素运输。gydF4y2Ba科学gydF4y2Ba370gydF4y2Ba, 550-557(2020)。gydF4y2Ba
齐默尔曼等人。合成用于膜蛋白构象捕获的单结构域抗体。gydF4y2BaeLifegydF4y2Ba7 gydF4y2Ba, e34317(2018)。gydF4y2Ba
科托夫,V.等。洗涤剂溶膜蛋白的高通量稳定性筛选。gydF4y2BaSci代表。gydF4y2Ba9gydF4y2Ba, 10379(2019)。gydF4y2Ba
亨特,C.等。a Na的结构gydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba反转运蛋白,并深入了解pH的作用机制和调控。gydF4y2Ba自然gydF4y2Ba435gydF4y2Ba, 1197-1202(2005)。gydF4y2Ba
Padan, E. & Michel, H. NhaA:次级主动转运蛋白的独特结构褶皱。gydF4y2BaIsr。j .化学。gydF4y2Ba55gydF4y2Ba, 1233-1239(2015)。gydF4y2Ba
本内特,S. R. M.,阿尔瓦雷斯,J.,博辛格,G. & Smyth, D.gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2Ba植物J。gydF4y2Ba8gydF4y2Ba, 505-520(1995)。gydF4y2Ba
胡南杰,岩田,S,卡梅隆,a . D.和德鲁,D.胆汁酸钠转运体ASBT的细菌同源物的晶体结构。gydF4y2Ba自然gydF4y2Ba478gydF4y2Ba, 408-411(2011)。gydF4y2Ba
李,C.等。钠-质子反转运体NhaA二聚体的晶体结构及机理新知。gydF4y2BaJ. Gen. Physiol。gydF4y2Ba144gydF4y2Ba, 529-544(2014)。gydF4y2Ba
李,C.等。钠/质子反口的双畴提升机构。gydF4y2Ba自然gydF4y2Ba501gydF4y2Ba, 573-577(2013)。gydF4y2Ba
董,杨,等。人NHE1-CHP1复合物的结构与机制。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 3474(2021)。gydF4y2Ba
温克曼,我等人。哺乳动物钠/质子交换器NHE9的结构与提升机制。gydF4y2BaEMBO J。gydF4y2Ba39gydF4y2Ba, e105908(2020)。gydF4y2Ba
松冈,R.等人。哺乳动物钠的结构、机制及脂质介导的重构gydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba换热器NHA2。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba29gydF4y2Ba, 108-120(2022)。gydF4y2Ba
贝纳斯科尼,P.,帕特尔,B. C.,里根,J. D. &萨勃拉曼尼亚,M. V. ThegydF4y2BaNgydF4y2Ba-1-萘酞酸结合蛋白是一种完整的膜蛋白。gydF4y2Ba植物杂志。gydF4y2Ba111gydF4y2Ba, 427-432(1996)。gydF4y2Ba
Drew, D. & Boudker, O.膜转运蛋白共享的分子机制。gydF4y2Ba为基础。学生物化学启。gydF4y2Ba85gydF4y2Ba, 543-572(2016)。gydF4y2Ba
加拉耶娃,A. A. &斯洛布姆,D. J.膜运输升降机式机制。gydF4y2Ba物化学。Soc。反式。gydF4y2Ba48gydF4y2Ba, 1227-1241(2020)。gydF4y2Ba
鲁伯,P. H. &谢尔德雷克,A. R.载体介导的生长素转运。gydF4y2Ba足底gydF4y2Ba118gydF4y2Ba, 101-121(1974)。gydF4y2Ba
植物细胞中吲哚乙酸的转运与pH值和电势梯度的关系,及其对IAA极性转运的意义。gydF4y2Ba新植醇。gydF4y2Ba74gydF4y2Ba, 163-172(1975)。gydF4y2Ba
去了,F. W.沉思和猜测。gydF4y2Ba为基础。Rev.植物物理学。gydF4y2Ba25gydF4y2Ba, 1-27(1974)。gydF4y2Ba
齐默尔曼等人。合成纳米体对抗脆弱的蛋白质。gydF4y2BaProtoc Nat。gydF4y2Ba15gydF4y2Ba1707-1741(2020)。gydF4y2Ba
郑世强等。MotionCor2:改进冷冻电子显微镜光束诱导运动的各向异性校正。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 331-332(2017)。gydF4y2Ba
Zivanov, J.等人。在RELION-3中自动高分辨率冷冻- em结构测定的新工具。gydF4y2BaeLifegydF4y2Ba7 gydF4y2Ba, e42166(2018)。gydF4y2Ba
Rohou, A. & Grigorieff, N. CTFFIND4:快速准确的电子显微图像离焦估计。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba192gydF4y2Ba, 216-221(2015)。gydF4y2Ba
Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A. cryoSPARC:快速无监督冷冻em结构确定的算法。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 290-296(2017)。gydF4y2Ba
陈,S.等。高分辨率噪声替代测量过拟合和验证分辨率在三维结构确定单粒子电子冷冻显微镜。gydF4y2BaUltramicroscopygydF4y2Ba135gydF4y2Ba, 24-35(2013)。gydF4y2Ba
罗森塔尔,P. B. & Henderson, R.单粒子电子冷冻显微镜中粒子取向、绝对手和对比度损失的最佳测定。gydF4y2BaJ. Mol.生物学。gydF4y2Ba333gydF4y2Ba, 721-745(2003)。gydF4y2Ba
Kucukelbir, A., Sigworth, F. J. & Tagare, H. D.对低温电磁密度图的局部分辨率进行量化。gydF4y2BaNat方法。gydF4y2Ba11gydF4y2Ba, 63-65(2014)。gydF4y2Ba
Emsley, P., Lohkamp, B., Scott, W. G. & Cowtan, K.白骨顶的特征与发育。gydF4y2BaActa Crystallogr。DgydF4y2Ba66gydF4y2Ba, 486-501(2010)。gydF4y2Ba
亚当斯,P. D.等。PHENIX:一个全面的基于python的大分子结构解决方案系统。gydF4y2BaActa Crystallogr。DgydF4y2Ba66gydF4y2Ba, 213-221(2010)。gydF4y2Ba
确认gydF4y2Ba
感谢中国科学技术大学低温电子显微镜中心和中国科学院生物物理研究所生物成像中心对电子显微镜设备的支持;我们感谢B. Zhu、X. Huang和所有其他工作人员在低温电子显微镜数据收集方面的技术支持。我们感谢J. Ren在传输分析方面的技术支持和M. Seeger提供的密码体库。国家自然科学基金项目(82151215、31870726资助D.L., 31900885资助X.L., 31870732资助孙林峰),安徽省自然科学基金项目(2008085MC90资助X.L., 2008085J15资助孙林峰),中央高校基本科研业务费专项(WK9100000031资助孙林峰),中国科技大学“双一流”科研经费(YD9100002004,孙林峰)。孙林峰获邱氏科技基金会杰出青年学者奖和唐居士基金会青年学者奖资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
孙林峰构想了这个项目。X.L.和孙林峰设计了实验。Z.Y.和J.X.完成了大部分的分子克隆、蛋白纯化和结构测定工作。C.Z.和C.S.进行了部分分子克隆和蛋白纯化。Z.Y.进行了ITC检测。J.X.进行了SPR测试。Z.Y., J.X., H.W.和W.Y.进行生长素转运试验。z。y。g。负责低温电子显微镜数据收集。J.H.在D.L的监督下制造了针对PIN1的同伙。孙良汉孝、Y.M.和S.T.协助进行了转运实验。 All authors contributed to data analysis. S.T. and J.F. contributed to results interpretation and manuscript preparation. X.L. and Linfeng Sun wrote the paper.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢Dolf Weijers和其他匿名审稿人对这项工作的同行评审所做的贡献。可以获得同行评审报告。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1 8个引脚的序列比对gydF4y2Ba拟南芥gydF4y2Ba.gydF4y2Ba
8个pin的序列可在Uniprot (gydF4y2Bahttps://www.uniprot.orggydF4y2BaPIN1: Q9C6B8, PIN2: Q9LU77, PIN3: Q9S7Z8, PIN4: Q8RWZ6, PIN5: Q9FFD0, PIN6: Q9SQH6, PIN7: Q940Y5, PIN8: Q9LFP6。使用ClustalW对序列进行对齐。PIN1的二级结构元件在序列上方。不变氨基酸用红色阴影表示。IAA相互作用残基以橙色五角形表示。gydF4y2Ba
图2 pin1介导的HEK293F细胞生长素转运。gydF4y2Ba
一个gydF4y2Ba抗flag标签抗体全细胞western blot检测PIN1表达。独立实验重复了三次,得到了相似的结果。gydF4y2BabgydF4y2Ba, PIN1和D6PK的共表达进一步降低[gydF4y2Ba3.gydF4y2BaH]-IAA在负载试验中的积累。gydF4y2BacgydF4y2Ba, PIN1和D6PK的共表达进一步降低[gydF4y2Ba3.gydF4y2Ba生长素外排试验中-IAA的保留。独立实验重复了三次gydF4y2Ba(bgydF4y2Ba,gydF4y2Bac)gydF4y2Ba.显著性用双尾未配对法测定gydF4y2BatgydF4y2Ba以及。***gydF4y2BaPgydF4y2Ba= 0.0004 forgydF4y2Ba在gydF4y2BaPIN1 vs PIN1 + D6PK ingydF4y2Ba(b)gydF4y2Ba, * * *gydF4y2BaPgydF4y2Ba= 0.0005 PIN1与PIN1 + D6PK ingydF4y2Ba(c)gydF4y2Ba.数据以均数±标准差表示。gydF4y2BadgydF4y2Bawestern blot检测PIN1磷酸化突变体的表达。T3E突变体的表达水平相对较低,而其他所有结构的表达水平相似。gydF4y2BaegydF4y2Ba,相对[gydF4y2Ba3.gydF4y2BaH]-IAA保留的磷酸盐突变体流出试验。每个结构都重复独立实验三次。显著性用双尾未配对法测定gydF4y2BatgydF4y2Ba以及。*gydF4y2BaPgydF4y2Ba= 0.0182, ns =不显著。数据以均数±标准差表示。gydF4y2BafgydF4y2Ba, IAA射流在不同ph后[gydF4y2Ba3.gydF4y2BaH]-IAA加载在pH 5.5的缓冲液中,细胞分别转移到pH为5.5或6.5的缓冲液中。每组N = 3个生物独立实验。显著性用双尾未配对法测定gydF4y2BatgydF4y2Ba以及。Ns =不显著。数据以均数±标准差表示。gydF4y2BaggydF4y2Ba, WT PIN1单独的典型凝胶过滤和考马斯蓝染色SDS-PAGE结果。独立实验重复了三次,得到了相似的结果。gydF4y2BahgydF4y2Ba, PIN1蛋白单独或与Sybody-21结合的微尺度荧光热稳定性分析结果。在Sybody-21的存在下,PIN1的热稳定性提高了~5度。所有凝胶和印迹的完整版本在补充图中提供。gydF4y2Ba1克ydF4y2Ba.gydF4y2Ba
图3 PIN1与Sybody-21的Cryo-EM分析gydF4y2Ba
一个gydF4y2BaWT PIN1结合Sybody-21的典型凝胶过滤和考马斯蓝染色SDS-PAGE结果。PIN1 + Sybody-21和冗余Sybody-21对应的峰分别用红色箭头和粉色箭头表示。独立实验已经重复了五次,得到了相似的结果。gydF4y2BabgydF4y2Ba, apo PIN1 + Sybody-21的典型冷冻- em图像。gydF4y2BacgydF4y2Ba,载脂蛋白PIN1 + Sybody-21的典型二维分类图像。gydF4y2BadgydF4y2Ba,载波PIN1 + Sybody-21整体图谱的金标准傅里叶壳层相关曲线。gydF4y2BaegydF4y2Ba, apo PIN1 + Sybody-21样本的局部分辨率图采用ResMap计算。gydF4y2BafgydF4y2Ba, apo PIN1 + Sybody-21图的欧拉角分布。gydF4y2BaggydF4y2Ba,载脂蛋白PIN1 + Sybody-21图的FSC模型曲线。gydF4y2BahgydF4y2BaPIN1 + IAA和Sybody-21样本整体图谱的金标准傅里叶壳层相关曲线。gydF4y2Ba我gydF4y2Ba, PIN1 + NPA和Sybody-21样本整体图谱的金标准傅里叶壳层相关曲线。gydF4y2BajgydF4y2Ba,低温电磁数据处理流程图。所有凝胶和印迹的完整版本在补充图中提供。gydF4y2Ba1克ydF4y2Ba.gydF4y2Ba
扩展数据图4 PIN1图的代表性电磁密度。gydF4y2Ba
一个gydF4y2Ba, apo PIN1图十个跨膜螺旋的EM密度。具有显著侧链的残基被标记到一边。gydF4y2BabgydF4y2Ba, apo PIN1图中配体结合口袋的EM密度(左),iaa结合图(中),npa结合图(右)。在PIN1 + IAA或NPA样品的EM图中识别出清晰的密度,分别作为IAA或NPA分子停靠。在IAA和N112残渣之间也观察到类似水的密度。gydF4y2BacgydF4y2Ba, IAA的电磁密度放大图。gydF4y2BadgydF4y2Ba, NPA电磁密度的放大视图。gydF4y2Ba
扩展数据图5 PIN1共享一个nhaa折叠。gydF4y2Ba
一个gydF4y2BaPIN1的前5个和后5个TMs为结构倒置重复序列。经两次旋转手术,TM6-10与TM1-5合并良好。gydF4y2BabgydF4y2BaPIN1的二聚体界面由每个单体的TM1、2、7组成。gydF4y2BacgydF4y2Ba二聚体界面主要由疏水残基组成。gydF4y2BadgydF4y2Ba, ASBT的两个钠结合位点gydF4y2Ba纳米gydF4y2Ba(左)与NagydF4y2Ba+gydF4y2Ba/小时gydF4y2Ba+gydF4y2Ba转运蛋白NapA(右)。左边,钠离子以紫色球体表示。N265和底物牛磺胆酸盐以棒状表示。介导N265和牛磺胆酸盐相互作用的水分子显示在红色球体中。对,在NapA的Na1位点存在赖氨酸残基,并通过离子相互作用稳定交叉螺旋。如棒状图所示,NapA的Na2位点主要由疏水残基组成。gydF4y2BaegydF4y2Ba, PIN1与ASBT内向结构之间的结构对齐gydF4y2BaYFgydF4y2Ba(PDB代码:4N7W)和ASBTgydF4y2Ba纳米gydF4y2Ba(PDB代码:3ZUX)。gydF4y2Ba
图6 PIN1的IAA和NPA协调。gydF4y2Ba
一个gydF4y2BaPIN1的apo和iaa结合结构的结构比较。gydF4y2BabgydF4y2Ba在载脂蛋白和IAA结合结构中,IAA结合位点周围的残基几乎相同。gydF4y2BacgydF4y2Ba, PIN1的apo和npa结合结构的结构比较。gydF4y2BadgydF4y2BaNPA结合位点周围的残基发生微妙的重排。gydF4y2BaegydF4y2Ba, IAA和NPA的化学结构。gydF4y2BafgydF4y2Ba, PIN1的iaa结合结构和npa结合结构的叠加。gydF4y2BaggydF4y2BaNPA的苯甲酸基团与TM5和TM4、TM9的交叉区域形成的口袋结合。为PIN1的表面静电势。gydF4y2Ba
扩展数据图7 PIN1与IAA、NPA或其他天然生长素的结合gydF4y2Ba
一个gydF4y2Ba, SPR分别检测ph7.0和5.5条件下PIN1与IBA、IPA和4-Cl-IAA的结合亲和力。在酸性pH下,IBA和IPA的结合亲和性显著降低,而4-Cl-IAA的结合亲和性显著降低gydF4y2BaKgydF4y2BadgydF4y2Ba大约增加了两倍。这些结果表明,pin可以结合一系列天然生长素,但PIN1是否可以运输这些生长素有待进一步研究。gydF4y2BabgydF4y2Ba, pH 5.5时WT PIN1的IAA-ITC结果。gydF4y2Ba氟gydF4y2Ba, PIN1 V51A、N112A、I582A和Y145A突变体在pH 7.0时的IAA-ITC结果。N112A、I582A和Y145A突变体未获得明显结合。V51A拟合的结合亲和力为1.39±0.16 mM,比WT PIN1高17倍。gydF4y2BaggydF4y2Ba,gydF4y2BahgydF4y2BaPIN1 N112A和Y145A突变体的NPA-ITC结果。gydF4y2Ba我gydF4y2Ba, PIN1转运IAA与NPA抑制IAA交替通路机制的卡通模型。gydF4y2Ba
补充信息gydF4y2Ba
补充图1gydF4y2Ba
未裁剪的western blot或考马斯蓝染色SDS-PAGE凝胶扫描。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
杨震,夏杰,洪杰,杨志强。gydF4y2Baet al。gydF4y2Ba生长素识别和外排的结构见解gydF4y2Ba拟南芥gydF4y2BaPIN1。gydF4y2Ba自然gydF4y2Ba609gydF4y2Ba, 611-615(2022)。https://doi.org/10.1038/s41586-022-05143-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05143-9gydF4y2Ba
这篇文章被引用gydF4y2Ba
n -1-萘酞酸的分子机制,植物生物学和农业的化学工具gydF4y2Ba
分子园艺gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba