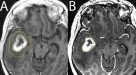
摘要
本研究旨在测试传统MRI获得的t1加权与T2加权信号强度之比(T1W/T2W比值:rT1/T2)是否可以作为松弛时间的替代预测IDH组织学上低级别胶质瘤的突变状态。8名被试的rT1/T2与T1、T2弛豫时间均呈指数相关性(rT1/T2 = 1.63exp)−0.0005 t1-relax+ 0.30, rT1/T2 = 1.27exp−0.0081 t2-relax+ 0.48;R2= 0.64和0.59)。在25例患者的测试队列中,IDHwt肿瘤的平均rT1/T2 (mrT1/T2)显著高于IDHmt肿瘤(p< 0.05), mrT1/T2鉴别IDHmt的最佳截止值为0.666 ~ 0.677,(AUC = 0.75,p< 0.05),在29例患者(AUC = 0.75)的外部国内队列中得到验证。p= 0.02)。然而,这一结果并未在TCIA/TCGA的外部国际队列中得到验证(AUC = 0.63,p= 0.08)。t分布随机邻居嵌入分析显示,TCIA/TCGA队列中图像特征的多样性大于两个国内队列。外部验证在TCIA/TCGA队列中的失败可归因于其更广泛的原始成像特征。
介绍
遗传特征是神经胶质瘤患者必不可少的诊断工作,作为手术前预测IDH突变状态有助于选择最合适的治疗策略。对于预后不良的idh -野生型(IDHwt)胶质瘤,应寻求快速和积极的手术干预1.相比之下,对于idh突变(IDHmt)世界卫生组织(WHO) 2级和3级胶质瘤(低级别胶质瘤:LrGGs)来说,在最大切除和保持日常生活质量之间找到最佳平衡是至关重要的,因为这些胶质瘤的预后更好1,2,3.,4,5.研究团体一直在努力开发能够预测疾病的非侵入性成像技术IDH胶质瘤的突变状态,包括磁共振波谱(MRS)6,7,8灌注和扩散加权磁共振成像(MRI)9,10以及利用传统核磁共振成像进行机器学习5,11,12.尽管这些技术主要依赖于定量或半定量的MRI测量,定性成像特征,如“T2流体衰减反转恢复(FLAIR)不匹配标志”已被证明是预测的强大成像替代标记IDH在组织学上表现为lrgg的肿瘤中的突变状态13,14,15.这些定性成像特征来自放射学定量测量,如T1和t2弛豫时间15,16.理想情况下,组织松弛时间应直接通过MR松弛仪测量;然而,由于需要额外的扫描时间,这尚未纳入常规成像方案。
传统MRI的t1加权(T1W)与T2加权(T2W)信号强度之比(T1W/T2W之比:rT1/T2)已被报道为检测脑髓磷脂完整性的定量成像技术17,18,19并且可能被用作组织松弛时间的成像替代物。本研究的目的是检验rT1/T2是预测的假设IDH组织学上lrgg的突变状态。
材料与方法
病人群
该研究得到了旭川医科大学医院机构审查委员会(批准号21041)和大阪国际癌症研究所(批准号1612065191)的批准。对于回顾性收集的数据,免除了书面知情同意的要求。为前瞻性招募队列中的患者提供了详细的解释,并获得了每位患者或其家属的书面知情同意。调查是按照所有有关的当地准则和条例进行的,并遵守《赫尔辛基宣言》的原则。
我们首先重新分析了先前发表的组织学LrGGs的T1和t2弛豫测量数据16并将这些T1和T2弛豫测量数据与rT1/T2进行比较(再分析队列,图2)。1).使用MP2RAGE和多回声t2加权图像对9例胶质瘤患者进行T1和t2弛缓测量。因此,我们可以获得这9例患者的T1和T2松弛测量以及rT1/T2数据,并在排除1例K27M突变肿瘤患者后将其中8例患者的数据纳入我们的分析16.
总体研究队列。首先,通过重新分析参考文献的原始数据,研究了rT1/T2与T1和T2弛豫时间的相关性。16.然后,本研究分两个阶段进行,首先是探索性队列研究,然后是验证性队列研究,研究rT1/T2与IDH组织学证实的lrgg的突变状态。IDHmt IDH-mutant;IDHwt IDH-wildtype;旭川医科大学附属医院AMUH;大阪国际癌症研究所;TCIA/TCGA,癌症影像档案/癌症基因组图谱。
然后,我们准备了一组新的三个队列,并进行了两个阶段的研究(图2)。1).第一队列(探索性队列)包括25例组织学和分子组织学证实的LrGGs (IDHwt: 8, IDHmt: 17),在旭川医科大学医院(AMUH)接受治疗。第二和第三队列被用作验证队列。验证队列1包括来自大阪国际癌症研究所(OICI)的29名患者(IDHwt: 13, IDHmt: 16),验证队列2包括来自癌症影像档案(TCIA)/癌症基因组图谱(TCGA)低级别胶质瘤收集数据集的101名患者(IDHwt: 19, IDHmt: 82),于2020年2月1日访问20.,21.验证队列1可视为“国内”验证队列,验证队列2可视为“国际”验证队列。
病理诊断依据2016年WHO中枢神经系统肿瘤分类22.根据2021年世卫组织分类系统,目前队列中的IDHwt肿瘤无法完全表征,如分子分析叔启动子突变,表皮生长因子受体没有进行基因扩增和+ 7/−10染色体拷贝数改变。纳入标准为T1WI和T2WI的可用性。排除了图像配准失败或图像不充分或不典型(如肿瘤出血)的患者。表格S1提供所有三个队列的详细信息。
基因分析
两个实验室对神经胶质瘤组织进行了遗传分析:日本旭川医科大学病理学系,用于探索性队列;以及日本大阪大阪国立医院临床研究所生物医学研究与创新部,作为验证队列1。免疫生物学检测IDH1对探索性队列进行Sanger测序,检测热点突变IDH1/2的密码子132IDH1密码子172IDH2)验证队列13..的IDH肿瘤在TCIA/TCGA数据集中的突变状态从Ceccarelli等人的报告中获得。23.
T1w/T2w (rT1/T2)图像重建
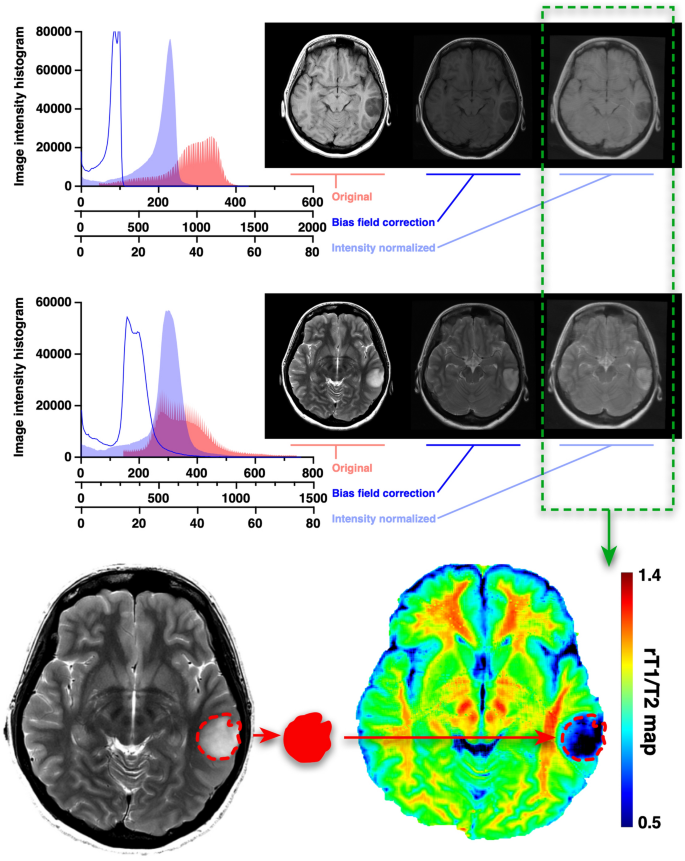
表格S1详细列出了MRI采集参数。探索性队列中的大多数图像使用通用电气3t扫描仪(芝加哥,伊利诺伊州,美国)获得,验证队列1中的图像使用西门子3t扫描仪(德国埃尔兰根)获得。TCIA队列(验证队列2)的图像由不同MRI供应商的1.5和3t扫描仪获得。使用Mango软件(版本4.0.1;德克萨斯大学健康科学中心,https://ric.uthscsa.edu/mango/mango.html(于2022年3月6日访问)。我们使用了一种内部成像软件,该软件包含了从T1WI和T2WI重建rT1/T2图像的算法24.计算rT1/T2的算法和MATLAB代码可以从Ganzetti等人开发的SPM12开源工具箱中获得(https://www.nitrc.org/projects/mrtool/(于2022年3月6日访问)18.Ganzetti等人之前报道了重建分析的细节。18.首先使用SPM12对原始T1WI和T2WI进行偏置场校正(https://www.fil.ion.ucl.ac.uk/spm/(于2022年3月6日访问)。然后根据从脑脊液、骨骼和软组织等非脑组织中提取的强度调整强度直方图。最后,使用NIfTI“扫描仪-解剖”坐标系,对处理后的T2WI进行共配准并除以处理后的T1WI生成rT1/T2图像(图2)。2).
MP2RAGE和多回波T2WI的T1、t2弛豫测量
在3t MR扫描仪(Prisma;西门子医疗,埃尔兰根,德国)。将MP2RAGE图像转换为t1松弛时间图,进行t1松弛测量。通过将多回波t2加权图像转换为t1松弛时间图进行t2松弛测量。在这两种情况下,松弛测量通过贝叶斯推理建模(Olea Nova+;佳能医疗系统,枥木,日本)。进一步的技术细节已在之前报道过16.
兴趣体素(VOIs)分割及平均rT1/T2计算
作者1具有6年神经外科经验,在ITK-SNAP软件(版本3.8.0,http://www.itksnap.org(于2022年3月6日访问)。在t2wi上设计voi,以视觉识别病理高强度区域,尽可能避免模糊和模糊异常病变(图2)。S1).最后一位作者,有22年的神经外科经验,然后评估了voi并确定了它们的位置或要求修改(发生在5个voi中)(表1)S1).这些voi (TCIA-00067、TCIA-00071、TCIA-00110、TCIA-00111、TCIA-00113)的Dice相似系数在0.52 ~ 0.81之间。使用NIfTI“一般仿射变换”坐标系对T2WI进行该操作。
在NIfTI“扫描-解剖”坐标系下,每张rT1/T2图像与NIfTI“一般仿射变换”坐标系下的T2WI图像使用神经学研究中的体积成像进行共配准,包括共配准和roi (VINCI;德国科隆马克斯·普朗克神经学研究所,http://www.nf.mpg.de/vinci3/,于2022年3月6日访问),以确保使用NIfTI“一般仿射变换”坐标系进行进一步分析(图2)。S2).然后将三维voi应用于rT1/T2图像,计算voi内的平均rT1/T2 (mrT1/T2)(图2)。2).当数据可用时,将VOIs应用于T1和t2弛豫时间图。
图像特征提取
根据上述方法提取T1WI和T2WI图像特征25.T1WI和T2WI在截断上0.1%的信号后转换为256级灰度图像。由于rT1/T2图像本质上是定量的,因此没有对rT1/T2图像执行此程序。基于T1WI、T2WI和rT1/T2图像的voi内256级灰度直方图计算一阶纹理特征。在灰度共生矩阵(GLGM)和灰度运行长度矩阵(GLRLM)分析中测量二阶纹理特征。每张图像共提取了49个成像特征(表1)S2).前面已经提供了提取的成像特征的详细信息25.纹理特征仅用于比较队列之间的图像特征,而不用于预测IDH突变状态。这样的多参数变量需要庞大的训练数据集来建立可靠的预测模型。
统计和t分布随机邻居嵌入(t-SNE)分析
使用Prism 9 for macOS (GraphPad Software, San Diego, CA, USA)进行统计分析。mrT1/T2与IDH采用Mann-Whitney U检验和受试者工作特征(ROC)曲线分析研究突变状态。一个p-value小于0.05被认为是显著的。采用t分布随机邻域嵌入(t-SNE)分析研究三个队列之间MRI质量和特征的差异。本分析使用了带有默认参数的0.15版本的Rtsne包(表1)S3,S4,S5)26.
结果
松弛测量与rT1/T2的相关性
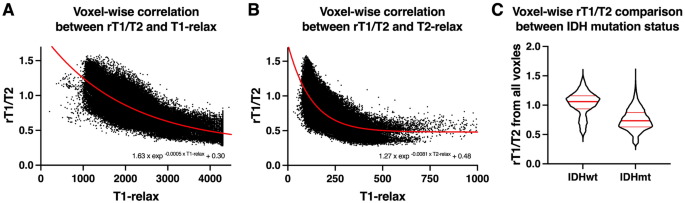
在LrGG组织学组织中T1-松弛时间和T2-松弛时间(分别缩写为T1-松弛和T2-松弛)与rT1/T2相关性的初步研究中,共对114,465个体素进行了体素分析。T1-和T2-弛豫与rT1/T2呈指数相关性(rT1/T2 = 1.63exp)−0.0005 t1-relax+ 0.30, rT1/T2 = 1.27exp−0.0081 t2-relax+ 0.48;R2= 0.64, 0.59)。3.A、B)。进一步比较IDHwt和IDHmt肿瘤体素的rT1/T2差异,发现IDHwt肿瘤体素的rT1/T2显著高于IDHmt肿瘤体素(中位数= 1.06 vs. 0.73);p< 0.0001)。3.C)。这些发现支持了进一步的研究,以验证rT1/T2作为预测肿瘤的影像学替代指标的假设IDH组织学上lrgg的突变状态。
测定rT1/T2截断值,区分LrGG的IDH突变状态
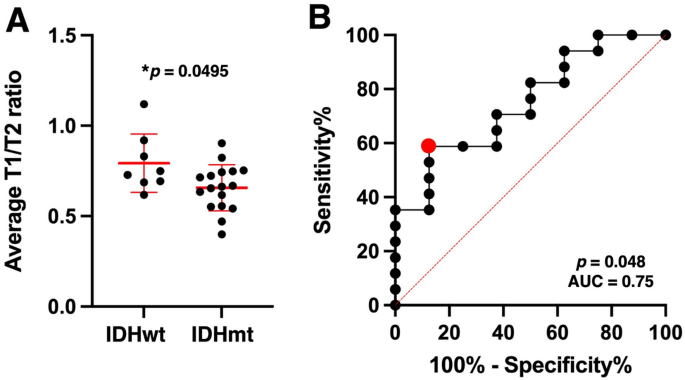
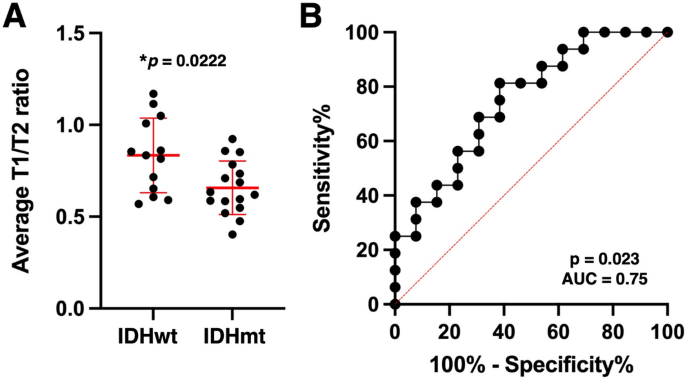
使用AMUH的探索性队列来探索和确定用于区分IDHwt和IDHmt肿瘤的平均rT1/T2 (mrT1/T2)的临界值。如图所示。4A, mrT1/T2在IDHwt肿瘤中显著高于IDHmt肿瘤(中位数= 0.74 vs. 0.66);p< 0.05)。ROC曲线分析显示,mrT1/T2鉴别IDHmt的最佳临界值为0.666 ~ 0.677,敏感性58.8%,特异性87.5% (AUC = 0.75);p< 0.05)。4B)。
rT1/T2截断值在国内和TCIA/TCGA数据集的验证
然后使用国内队列(来自OICI的验证队列1)和国际公共数据集(TCIA/TCGA队列)验证确定的mrT1/T2截断值。OICI队列验证了IDHwt和IDHmt肿瘤之间mrT1/T2的统计学差异(中位数= 0.83 vs. 0.63)。p= 0.02)。5A)。OICI队列的ROC分析显示AUC为0.75 (p= 0.02),灵敏度为56.3%,特异性为69.2%,使用先前获得的检测IDHmt肿瘤的截止值(图2)。5B)。
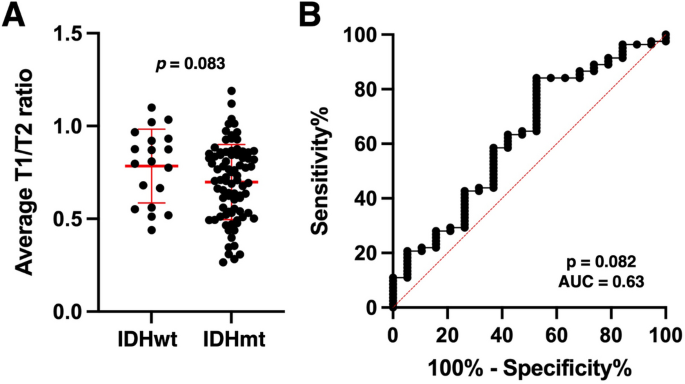
在验证队列2 (TCIA/TCGA队列)中,IDHwt和IDHmt肿瘤之间的mrT1/T2无显著差异(p= 0.08)。6A)。验证队列2 (TCIA/TCGA队列)的ROC分析显示,使用先前获得的检测IDHmt LrGGs的截止值,灵敏度为42.7%,特异性为73.7% (AUC = 0.63,p= 0.08)。6B)。
原始图像特征对rT1/T2重构的影响
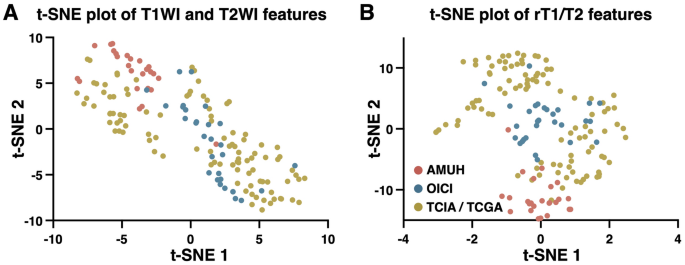
我们进一步研究了原始T1WI和T2WI图像特征对重建rT1/T2图像的影响。原始T1WI和T2WI的t-SNE分析显示探索性队列(AMUH队列)和验证队列(OICI队列)中的图像特征聚类,而验证队列2 (TCIA/TCGA队列)中的图像特征分散在t-SNE1和t-SNE轴上(图2)。7A).从T1WI和T2WI重建rT1/T2后,所有数据点的聚类更加密集(图2)。7B),使AMUH和OICI队列的数据点更接近。然而,与两个国内队列相比,从TCIA/TCGA队列获得的数据点仍然分散。进一步分析发现,TCIA/TCGA队列数据点的分散部分归因于组成整个队列的机构的多样性(图2)。S3A)。从T1WI和T2WI重建rT1/T2不足以聚类所有数据点,这在亨利福特医院的数据中尤为明显(图2)。S3B)。
对整个队列的原始T1WI和T2WI特征以及rT1/T2特征的t-SNE分析无法区分IDH突变状态(图2)S4).
讨论
与其他研究人员一起,我们报道了肿瘤组织的T1和t2弛豫特性与肿瘤组织的T1和t2弛豫特性密切相关IDHlrgg的突变状态16,27.这些观察结果是通过采用传统的磁共振弛豫技术获得的16,28或者通过新开发的磁共振指纹识别技术29.然而,由于扫描时间方面的额外要求,这两种技术都尚未被纳入常规的LrGG成像研究16或序列和后处理优化30.,31.相比之下,传统的结构MRI(即T1WI和T2WI)可以部分表征组织在射频诱导的核磁共振自旋极化后的弛豫特性。例如,T2-FLAIR错配标志利用了定量组织松弛特性所代表的常规结构MRI的定性成像特征,从而能够检测idh突变1p/19q完整肿瘤13,14,15,16.
本研究旨在探索利用rT1/T2从定性常规结构MRI中检索定量数据的可能性,rT1/T2最初是作为脑内髓磷脂含量的成像替代方法提出的17.虽然确切的病理机制尚不清楚19,32, rT1/T2检测多发性硬化症的相对较高的灵敏度表明,它可能适用于表征大脑内的组织微观结构33.如图所示。3.rT1/T2与组织T1、T2弛豫时间呈良好的相关性。此外,在国内两组组织学LrGGs患者队列中,我们探索并验证了病变内rT1/T2均值可以区分IDHwt和IDHmt肿瘤,其敏感性为56%-59%,特异性为69-88%(图2)。4和5).这些令人鼓舞的发现支持了rT1/T2可用于手术前测定的假设IDH组织学上LrGGs的突变状态,不进行组织松弛特性的直接测量。
然而,在国内队列(验证队列1)中获得的结果并没有在TCIA/TCGA队列(验证队列2)中得到重复(图2)。6).虽然IDHmt肿瘤的rT1/T2低于IDHwt肿瘤的趋势得到了再现,但与国内队列相比,差异要小得多。我们认为这种差异是由于国内队列(探索性和验证性队列1)和TCIA/TCGA队列(验证性队列2)在图像质量和图像特征上的差异造成的。正如预期的那样,TCIA/TCGA队列比国内队列具有更大的图像特征多样性,可能是因为TCIA/TCGA队列包含来自美国四个不同医疗机构的图像(图2)。7和无花果。S3)34.表格S1表明每个机构对MRI采集参数有不同的偏好,这可能会影响最终的分析。最近一项针对多发性硬化症患者rT1/T2的多中心临床研究结果证实了这一观点,该研究使用了不同场强的磁共振扫描仪。在整个队列中观察到rT1/T2的广泛变化,可能是由于输入数据的异质性35.因此,使用rT1/T2进行预测IDH突变状态时,可能需要使用预设的MRI采集方案或机构相关的截止值。
必须解决本研究的几个局限性。首先,国内两个队列的样本量相对较小,IDHwt和IDHmt在三个队列中的分布存在差异。这种分布差异可能是TCIA/TCGA队列中截断值无法验证的原因。理想情况下,一个具有较大样本量和肿瘤均匀分布的队列IDH突变状态是验证我们发现的必要条件。二是总体敏感性和特异性不高,临床常规应用不能接受。由于数据不足,目前的回顾性研究无法整合其他成像方式进行分析;然而,将rT1/T2与扩散和灌注MRI相结合可能会提高诊断的准确性,这方面的研究应在未来进行。例如,最近的一项研究提出了弥散和灌注MRI相结合的概念IDH突变检测,但需要外部验证36.第三,如Ganzetti等人所述,rT1/T2重建过程中的一些手术非常复杂。18,尤其是图像强度归一化。本研究中使用的归一化方法使用非脑组织(包括脑脊液、骨骼和软组织)校准图像强度;然而,深度学习等其他方法可以为改善rT1/T2分析提供潜在的解决方案37.最后,最近深度学习和人工智能的快速发展提高了诊断的准确性IDH胶质瘤的突变状态,报道的敏感性和特异性分别为0.88和0.8612,这比本研究的结果要高得多12.最合适的机器学习算法还有待确定。因此,仅通过T1WI和T2WI重建rT1/T2的简单性仍然具有吸引力,尽管需要进一步改进和验证。
结论
本研究结果表明,组织学上LrGGs的rT1/T2与T1和T2松弛时间密切相关。国内两个队列rT1/T2均值能够区分IDHwt和IDHmt肿瘤,差异均有统计学意义。然而,由于原始TCIA/TCGA队列中影像学特征的多样性,这一结果并未在原始TCIA/TCGA队列中得到验证。
数据可用性
本研究中使用和分析的数据集可应通讯作者的合理要求向其提供。
参考文献
埃克尔-帕索,j.e。et al。基于肿瘤中1p/19q、IDH和TERT启动子突变的胶质瘤组。心血管病。j .地中海。372中文信息学报,2499-2508(2015)。
铃木H。et al。II级和III级胶质瘤的突变景观和克隆结构。Nat,麝猫。47, 458-468(2015)。
Arita, H。et al。TERT启动子突变和MGMT甲基化状态的结合可以预测新诊断的胶质母细胞瘤的临床相关亚群。Acta Neuropathol。Commun。4, 79(2016)。
Arita, H。et al。胶质瘤中IDH1/2突变检测。脑肿瘤病理32, 79-89(2015)。
Arita, H。et al。病变定位采用磁共振成像放射组学预测II/III级胶质瘤中IDH和TERT启动子突变。科学。Rep.-UK8, 11773(2018)。
崔C。et al。idh突变胶质瘤患者2-羟戊二酸的磁共振波谱检测。Nat,地中海。18, 624-629(2012)。
安德洛内西,o.c.。et al。2-羟戊二酸的检测IDH-突变胶质瘤患者的体内光谱编辑和二维相关磁共振波谱。科学。Transl。地中海。4[j] .浙江大学学报(自然科学版),2012。
安德洛内西,o.c.。et al。2-羟戊二酸的磁共振波谱检测致癌性IDH1突变。j .中国。投资。123, 3659-3663(2013)。
低浓缩铀,K。et al。WHO分级II-III级弥漫性胶质瘤组织学和遗传亚型的灌注和扩散MRI特征。j . Neuro-oncol。134, 177-188(2017)。
兴,Z。et al。采用DWI和DSC-PWI结合常规MR成像无创评估世界卫生组织II级和III级星形细胞瘤中IDH突变状态。点。j . Neuroradiol。38科学通报,1138-1144(2017)。
赵,J。et al。机器学习识别神经胶质瘤患者IDH突变的诊断准确性和潜在协变量:来自荟萃分析的证据。欧元。Radiol。30.中文信息学报,4664-4674(2020)。
剑,一个。et al。机器学习预测脑胶质瘤分子标记的磁共振成像:系统回顾和荟萃分析。神经外科89, 31-44(2021)。
帕特尔,s.h.。et al。T2-FLAIR不匹配,低级别胶质瘤中IDH和1p/19q状态的成像生物标志物:TCGA/TCIA项目中国。癌症Res。23中文信息学报,6078-6085(2017)。
布朗,m.p. G。et al。T2-FLAIR不匹配标志作为非增强idh突变,1p/19q完整的低级别胶质瘤的成像标记:一项验证研究神经肿瘤防治杂志。20., 1393-1399(2018)。
木下光男,M。et al。获取FLAIR反转时间对idh突变、非codel星形细胞瘤T2-FLAIR错配可检测性的影响。前面。肿瘤防治杂志。10, 596448(2021)。
木下光男,M。et al。T2- flair错配标志是由idh突变型1p19q非编码星形细胞瘤的T1和T2过长引起的。粉剂。的原因。医学科学。20., 119-123(2021)。
基于T1和t2加权MRI显示的髓磷脂含量在体内绘制人类皮层区域。j . >。31中文信息学报,1197 - 1116(2011)。
Ganzetti, M., Wenderoth, N.和Mantini, D.使用T1和t2加权磁共振成像数据绘制全脑髓鞘。前面。嗡嗡声。>。8, 671(2014)。
帕累托,D。et al。衡量组织完整性的t1加权与t2加权信号强度之比:与多发性硬化症患者磁化转移比的比较点。j . Neuroradiol。41, 461-463(2020)。
The_Cancer_Genome_Atlas。https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga(2020)。2020年2月1日发布。
The_Cancer_Imaging_Archive。https://public.cancerimagingarchive.net/ncia/login.jsf.(2020)。2020年2月1日发布。
International_Agency_for_Research_on_Cancer。WHO对中枢神经系统肿瘤的分类(国际癌症研究机构,2016)。
切,M。et al。分子图谱揭示了弥漫性胶质瘤的生物学离散亚群和进展途径。细胞164, 550-563(2016)。
山本,S。et al。T1w/ t2w比值图对胶质母细胞瘤非增强性肿瘤的预测与可视化。大脑科学。12, 99(2022)。
佐佐木,T。et al。放射组学和MGMT启动子甲基化对新诊断的胶质母细胞瘤的预后。科学。Rep.-UK9, 14435(2019)。
van der Maaten, L. J. P. & Hinton, G. E.使用t-SNE可视化数据。j·马赫。学习Res。9中文信息学报,2579-2605(2008)。
施普林格,E。et al。MR指纹-弥漫性胶质瘤的放射基因组标记。癌症14, 723(2022)。
木下光男,M。et al。磁共振松弛法在胶质瘤肿瘤细胞密度成像中的应用:11c -蛋氨酸PET的探索性研究及其通过立体定向组织取样的验证。癌症13, 4067(2021)。
妈,D。et al。磁共振指纹识别。自然495, 187-192(2013)。
可怜的人,m.e.。et al。磁共振指纹识别第1部分:潜在用途、当前挑战和建议。j .增效。的原因。成像51, 675-692(2019)。
麦吉夫尼博士。et al。磁共振指纹鉴定复习第2部分:技术和方向。j .增效。的原因。成像51中文信息学报,993-1007(2019)。
Preziosa, P。et al。神经突密度解释多发性硬化症皮质t1加权/ t2加权比值。j .神经。Neurosurg。精神病学。JNNP。https://doi.org/10.1136/jnnp-2020-324391(2021)。
Boaventura, M。et al。多发性硬化症T1/ t2加权比值:一项与临床相关的纵向研究杂志中国。34, 102967(2022)。
崔玉生。et al。通过深度学习和放射组学预测胶质瘤IDH突变状态的全自动混合方法。神经肿瘤防治杂志。23中文信息学报,304-313(2020)。
Margoni, M。et al。通过T1w/T2w比值探讨多发性硬化症脑微结构损伤:一项多中心研究。j .神经。Neurosurg。精神病学93, 741-752(2022)。
普鲁伊斯。et al。利用MRI灌注和扩散参数对成人非增强胶质瘤分子亚型进行无创分化。Neuro-oncol。睡觉。4, vdac023(2022)。
胡,Z。et al。MIL归一化-准确的MRI放射组学分析的先决条件。第一版。医学杂志。地中海。133, 104403(2021)。
致谢
本研究由日本科学促进会(no . 19K09526, 22K09200),日本医学研究与开发机构(日本癌症研究项目,no . 19188187和21459042),武田科学基金会,MSD生命科学基金会和大川信息与电信基金会资助。所有的科学拨款都是给MK的。
作者信息
作者及单位
贡献
M.K.构思了这个实验;理科硕士t。,h。新墨西哥州S.H, r.a., Y.K, M.T。和K.N.收集数据;t . S.Y。,T.U, Y.K, M.T。K.N.抗议进行实验;T.S.和M.K.写了手稿;H.K.和M.K.监督了这个研究项目。所有作者都审阅了稿件。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益冲突。
额外的信息
出版商的注意
b施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议,该协议允许以任何媒介或格式使用、共享、改编、分发和复制,只要您适当地注明原作者和来源,提供知识共享许可协议的链接,并注明是否进行了更改。本文中的图像或其他第三方材料包含在文章的知识共享许可协议中,除非在材料的署名中另有说明。如果材料未包含在文章的知识共享许可中,并且您的预期用途不被法律法规允许或超过允许的用途,您将需要直接获得版权所有者的许可。如欲查阅本许可证副本,请浏览http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
Sanada, T., Yamamoto, S., Sakai, M.;et al。T1- t2加权信号强度比与T1-和t2 -松弛时间的相关性IDH胶质瘤中的突变状态。Sci代表12, 18801(2022)。https://doi.org/10.1038/s41598-022-23527-9
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41598-022-23527-9