摘要
涎管癌(SDC)预后的分子靶点和预测性生物标志物尚未完全确定。我们进行了全面的分子分析,以发现SDC的新型生物标志物。共对67例SDC样本进行了464个基因的DNA测序,并结合个体的临床病理特征进行了转录组分析。利用来自27例患者的mRNA表达数据,探索了与联合雄激素阻断(CAB)治疗反应相关的预后生物标志物。55例(82.1%)发现受体酪氨酸激酶(RTK)基因或MAPK通路基因发生致癌突变。38例患者(56.7%)发现磷脂酰肌醇3-激酶(PI3K)/AKT信号通路发生改变。有趣的是,患者的预后可以用mRNA表达谱来预测,但不能用基因突变谱来预测。风险评分生成的表达数据的四基因集,其中包括ADAMTS1,DSC1,RNF39,IGLL5在队列中,基因是总生存期的重要预后标志物(HR = 5.99, 95%可信区间(CI) = 2.73-13.1,p= 7.8 × 10−6).的表达式构造出另一个风险评分CD3E而且LDB3是CAB治疗无进展生存期的一个强有力的预后标志(p= 0.03)。RTK基因、MAPK通路基因和PI3K/AKT通路基因的突变可能是SDC肿瘤发生的关键突变。本研究中确定的基因表达谱可能有助于对CAB治疗的良好候选者进行分层,并可能受益于额外的全身治疗。
简介
涎管癌(SDC)是一种侵袭性恶性肿瘤,类似于高级乳腺导管癌1,2,3..SDC曾被认为占所有唾液腺癌的1-4%。4,5,6尽管最近的研究报告的发病率高于以前的记录3.,7.SDC通常发生在主要的唾液腺,如腮腺,多见于50岁以上的男性8,9.SDC可以从头发生,也可以作为癌外多形性腺瘤(PA)的恶性成分发生。1,2,10.
目前SDC的治疗方法包括完全手术切除原发部位,然后辅助化疗/放疗2,3.,8,9.然而,复发和远处转移频繁发生,导致预后不良。在常规治疗下,超过50%的SDC患者在3-5年内死于该疾病,即使在完全手术切除后,他们仍有很大的复发风险。5年总生存率(OS)为20% ~ 30%2,3.,6,8,9,11.对于切除术后晚期SGC患者的全身治疗或放射治疗,疗效数据有限1,2,8,9.目前迫切需要可靠的预后生物标志物来选择高危复发患者以及可能受益于额外的全身或放射治疗的患者1,2,3..
与乳腺癌相似,常规免疫组化发现SDC常表现出雄激素受体(AR)、HER2(编码为ERBB2基因)和表皮生长因子受体(EGFR)8,12,13,14,15,16,17.然而,SDC通常缺乏雌激素受体或孕激素受体的表达,这在乳腺癌中经常观察到12,18.联合雄激素阻断(CAB)与LHRH类似物和比卡鲁胺是一种常见的雄激素剥夺疗法(ADT),具有相对轻微的毒性,被认为是不可切除的ar阳性SGC患者的一种有前途的治疗选择。一些临床试验已经证明ADT治疗对患者有良好的反应8,19,20.,21,22.在前列腺癌中,生成的AR-V7编码的剪接变异基于“增大化现实”技术可能对雄激素抑制剂治疗产生耐药性23,而在SDC中很少发现预测CAB治疗疗效的特异性生物标志物24,25,26,27,28.对于her2靶向治疗,曲妥珠单抗联合化疗在临床试验中产生了令人印象深刻的反应率29,30..
由于新一代测序技术的发展,SDC研究了基因、转录组和蛋白质组谱31,32,33,34,35,36.肿瘤突变负担高于其他肿瘤类型的SGC,其中磷脂酰肌醇3-激酶(PI3K)/AKT/mTOR通路是最常改变的通路37,38.其他突变常在TP53,极品/国家管制当局方面,ERBB2,表皮生长因子受体,BRAFCCND1/CDK通路基因31,39,40,41,42,43.有报道描述了SDC临床结果的预后因素31,39.肿瘤微环境中免疫细胞的浸润程度是免疫检查点抑制剂的预测指标34,36.然而,尚未有研究将全面的分子分析数据与SDC患者详细的临床数据相结合,进行丰富的综合分析。因此,我们对一组SDC患者进行了综合癌症基因组和RNA测序(RNA-seq)的综合分析,并结合详细的临床特征分析这些数据。
结果
病人的特点
该研究队列包括76名SDC患者,他们于2005年至2017年在参与日本SDC联盟的医院接受了手术切除。9例因从标本中分离出的DNA质量较差而被排除。其余67例患者(8例女性,59例男性)的人口统计学和临床资料汇总于表中1.中位年龄为62.1岁,女性和男性患者的中位年龄相似(平均59.4岁vs. 62.6岁;p= 0.5)。原发肿瘤部位以腮腺最多见(76.1%),其次为颌下腺(20.9%)。12例(17.9%)SDC是PA外癌的恶性组成部分。超过50%的患者为IV期癌症(62.7%),并接受了手术和化疗/放疗(56.7%)。在40个月的中位随访时间内,73.1%的患者在初始治疗后观察到癌症复发。共有27例患者(40.3%)接受了CAB治疗。
肿瘤基因组测序SDC突变谱
我们使用了TOP癌症基因组44评估小组中464个基因的体细胞单核苷酸变异(SNVs)和插入/缺失(indels),并确定肿瘤突变负担(TMB)和拷贝数变异。我们在受体酪氨酸激酶(RTK)/MAPK信号通路的基因中发现了频繁的致癌突变,包括24例极品激活突变7例ERBB2突变5例BRAF突变4例MAP2K4突变13例NF1突变(图。1).TP53在大约一半的病例中发生了改变。拷贝数分析进一步鉴定出6例带有ERBB2放大。总的来说,55例(82.1%)存在RTK基因或MAPK通路基因的致癌突变。在我们队列中发现的所有突变的列表显示为补充数据1.我们纳入了Dalin等人之前的研究结果。31表示单个基因的突变频率(补充图;1).
一个经常突变的基因,用颜色编码它们对每个肿瘤的改变状态。顶部显示性别、年龄、分期、原发部位、肿瘤来源(新生或非多形性腺瘤)、肿瘤突变负荷(TMB)。b描述酪氨酸激酶融合的示意图。FGFR2,受潮湿腐烂,NTRK3RNA-seq识别的融合显示了它们的功能域。的FGFR2基因(NM_000141)在17外显子下游被破坏,随后在2外显子上游被连接CCDC6的外显子3 (NM_005436)KIAA1217(NM_019590),或的外显子10NOL4(NM_003787)。外显子15NTRK3基因(NM_002530)连接于基因的5外显子上ETV6(NM_001987)。酪氨酸激酶结构域(TK)在所有鉴定的融合中都保持不变。CC,线圈域;HLH,螺旋-环-螺旋结构域。c在腮腺和颌下腺肿瘤中发现驱动突变。d驾驶员突变阳性和阴性病例的平均年龄(左)和最高变异等位基因频率(右)的比较。箱线图的元素定义如下:中心线、中位数;框的下界,下四分位数;方框的上界,上四分位数;下须,最小值;上须,最大值。
此外,38例肿瘤中PI3K通路出现异常(56.7%)PIK3CA(29例),PIK3R1(5例),PTEN(7例)和AKT1(1例)以相互排斥的方式发现。极品突变显著同时发生PIK3CA变异(83.3%)。在多个病例中发现的其他基因突变ZFHX3(8例),FOXA1(6例),ARID1A(6例)KMT2A / C / D(分别5例、9例、3例)。平均TMB为2.3 mut/Mb, 2例(2.7%)出现高突变(>10 mut/Mb)。
RNA-seq法检测SDC融合基因
RNA-seq检测出4例酪氨酸激酶融合基因(FGFR2-CCDC6,FGFR2-KIAA1217,FGFR2-NOL4,ETV6-NTRK3)及六宗其他融合个案(CHCHD7-PLAG1,FGFR1-PLAG1,MAPK14-ZFAND3,NFIX-MAST1,SLC45A3-ELK4,TMCC1-PLXND1,ERBB2-CTTN)(图。1 b和补充图。2).
总的来说,在55例(82.1%)病例中发现了驱动癌基因,我们将其定义为受体酪氨酸激酶(RTK)基因或MAPK通路基因中的致癌突变。
SDC中mRNA表达分析
的表达式配置文件ERBB2将RNA-seq建立的拷贝数数据与TOP组、免疫组化法测定的蛋白表达以及FISH法测定的拷贝数进行比较,四种测定结果一致(补充图1)。3.).因此,RNA-seq分析可能是稳健的评估ERBB2畸变。类似地,的表达式基于“增大化现实”技术(p= 7 × 10−4,低vs高,学生的t测试),表皮生长因子受体(p= 0.02, 3+与其他,学生的t试验)均与免疫组化分析结果相关。相比之下,CD274与驱动因子阳性肿瘤相比,驱动因子阴性肿瘤中编码PD-L1的表达更丰富(p= 0.01,学生的t测试)。
遗传特征与临床病理特征的关联
大约40%的肿瘤在腮腺中隐藏极品突变和40%的下颌下肿瘤呈阳性ERBB2基因改变(图;1 c).所有极品-突变瘤(23例)为新生PDC,而50%(6/12)为前PA肿瘤ERBB2基因改变。
为评估驱动突变是否为主干突变,将驱动基因的变异等位频率(VAF)与所有突变基因中的最高VAF进行比较。对于snv和indels驱动突变阳性的病例,驱动VAF与最高VAF的平均比值为75.5 (95% CI = 12.6-134),表明驱动突变是SDC的基础突变(补充图)。4).
驾驶员阳性和阴性病例的最高VAF无显著差异(平均分别为36.4%和44.8%)。p= 0.05,学生t测试)(图1 d).驾驶员阳性病例的平均年龄略高于驾驶员阴性病例(分别为63.5岁和55.2岁)。p< 0.05,学生t测试)(图1 d).所有临床病理特征见补充资料2.
SDC的转录谱
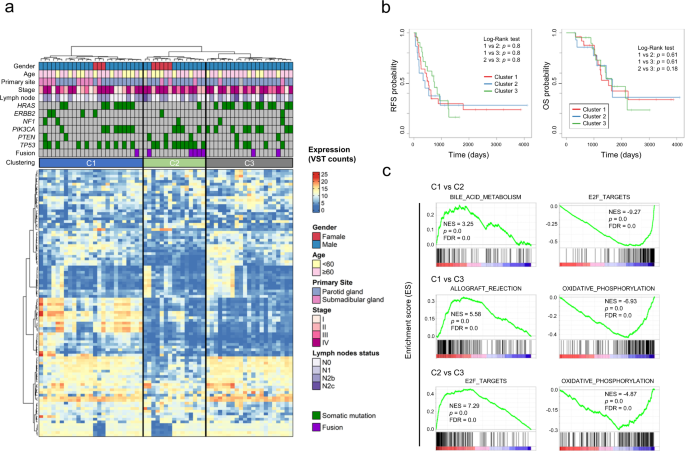
我们根据RNA-seq数据的基因表达谱对肿瘤进行k-means聚类分析,并将聚类与临床病理特征或基因突变谱进行比较(图2)。2).使用表达变异最大的前100个基因进行聚类,将队列分为三组:聚类1(图中左边的聚类)。2)、簇2(中)和簇3(右)(补充数据3.).女性患者在聚类2 (p< 0.01,聚类2 vs其他,Fisher精确检验),而男性患者在聚类3中富集(p< 0.05,聚类3 vs.其他,Fisher精确检验)。各组间无进展生存期(PFS)无显著差异(图2)。2 b).中位随访时间为40.3个月(范围2.4-135个月),1-3组的中位PFS分别为16.4、11、23.6个月(95% CI = 16-42.8个月、6.6-41.1个月和17.7-30.3个月)。1-3组的中位OS分别为40.5、36.5和46.5个月(95% CI = 38.7-64.2个月、26.2-57.8个月和37.5-55.6个月)。
一个利用RNA-seq数据进行K-means聚类分析。上半部分显示临床信息(性别、年龄、原发部位、淋巴结受累、突变情况)。Fisher检验通过k-means聚类确定了与两组相关的因素。b通过k-means聚类划分为1-3组的队列无复发生存Kaplan-Meier曲线。cGSEA结果显示,在k-means聚类定义的聚类中,所示基因集差异富集。完整的GSEA结果可在补充数据中找到4.
基因集富集分析(GSEA)显示,3个聚类中2、9和12个基因在聚类1-3位点分别上调(问< 0.01,一个聚类比其他聚类)。其中,“E2F_TARGETS”和“HALLMARK_INTERFERON_ RESPONSE”富集在簇2中,“HALLMARK_OXIDATIVE_PHOSPHORYLATION”富集在簇3中,“hallmark_epithelial - mesenchymal_transition”富集在簇1中。这些数据是大量的转录组,这三组可能很好地反映了微环境的组成(图。2摄氏度及补充资料4).唾液腺标记基因的表达包括HTN1/3而且PRB1/2/3/4编码唾液糖蛋白在聚类1中特异性升高,提示聚类1肿瘤中的唾液分化(补充数据)3.).
SDC治疗后生存的风险评估
使用来自61例病例的RNA-seq数据进行风险评估。中位随访时间为40.5个月(范围:10-135个月),中位OS为40.5个月(95%置信区间(CI) = 40.3-54.6个月)。OS分析选择满足以下条件的基因:总样本中超过80%(49/61)的基因表达为>0,sd为>1。单因素Cox比例风险回归分析显示9个基因与OS显著相关(p≤2 × 10−3).
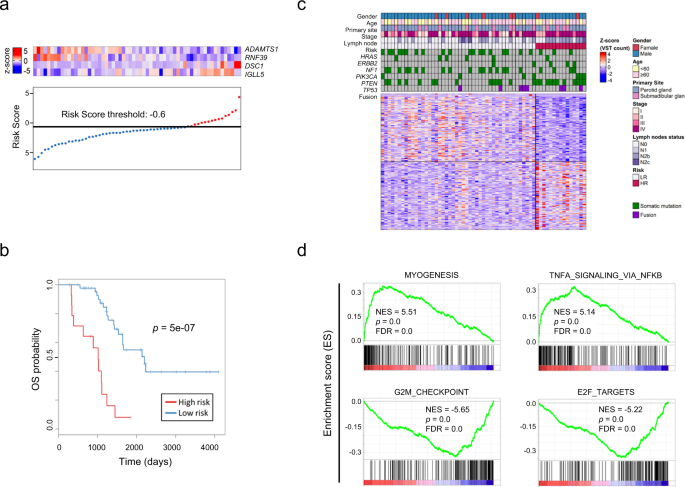
为了生成OS预后特征,在队列中使用这9个基因进行正向条件逐步回归和多变量Cox分析。该程序建立了包含四个基因的预后模型,包括ADAMTS1,DSC1,RNF39,IGLL5.我们利用该模型的回归系数构建了一个风险评分,并在第75百分位进行了人工选择,选择了一个合适的阈值,即−0.6(图6)。3).高危患者(由基于四基因特征的风险评分定义)OS明显较差(p= 5 × 10−7, log-rank检验)。3 b).
一个四基因表达和风险评分在队列中的分布z分数。红色和浅蓝色分别表示高表达和低表达。所有患者的风险评分按升序绘制,并按阈值(垂直黑线)划分为低风险(蓝色)或高风险(红色)。风险评分阈值为−0.6。b高危组和低危组按四基因预后特征分层的队列总生存Kaplan-Meier曲线。采用log-rank检验进行评价p值。c高危和低危患者前200个差异表达基因的热图,红色和蓝色分别表示高表达和低表达。d在高危肿瘤中,GSEA鉴定出显著不同的基因集为差异过表达或低表达。补充数据4展示了完整的GSEA结果。NES归一化富集评分,FDR错误发现率。
为了了解高危肿瘤的生物学基础,我们确定了高危肿瘤中前100个显著过表达基因和前100个低表达基因(图2)。3 c及补充资料3.).在低高危组和高危组之间没有不同的临床特征。然而,TP53根据Fisher精确测试,高危组的突变显著增加(p< 0.01)(补充图。5).与细胞增殖相关的基因集,如“G2M_CHECKPOINT”和“E2F_TARGETS”在高风险肿瘤中显著富集,而“MYOGENESIS”和“TNFA_SIGNALING_VIA_NFKB”等基因集在低风险组中富集(图2)。3 d及补充资料4).
使用多变量Cox分析,OS风险评分仍然显著(HR = 5.62,p= 7.7 × 10−3)在不受性别、年龄、原发部位、对一线治疗的反应和基因突变影响的队列中进行2).通过单因素分析,OS的其他危险因素包括对一线治疗的反应(SD;hr = 17.2,p= 6.5 × 10−4帕金森病;hr = 7.94,p= 2.1 × 10−4),PTEN突变(HR = 2.51,p= 0.02),TP53突变(HR = 2.74,p= 3 × 10−3),融合基因的存在(HR = 2.6,p= 0.04),淋巴结状态N2b/N2c (HR = 2.31,p= 0.01);然而,这些因素在多变量分析中均无显著差异。
CAB治疗的风险评估
与CAB反应相关的生物标志物来源于27例CAB治疗患者的mRNA表达数据。中位随访时间为22.2个月(范围4.2-61.4个月),CAB治疗的中位PFS为9.9个月(95% CI = 6.7-13.0个月)。选择满足以下条件的基因进行分析:样本中基因表达量最高的为>50。单因素Cox比例风险回归分析显示14个基因与PFS显著相关(p≤2 × 10−3).
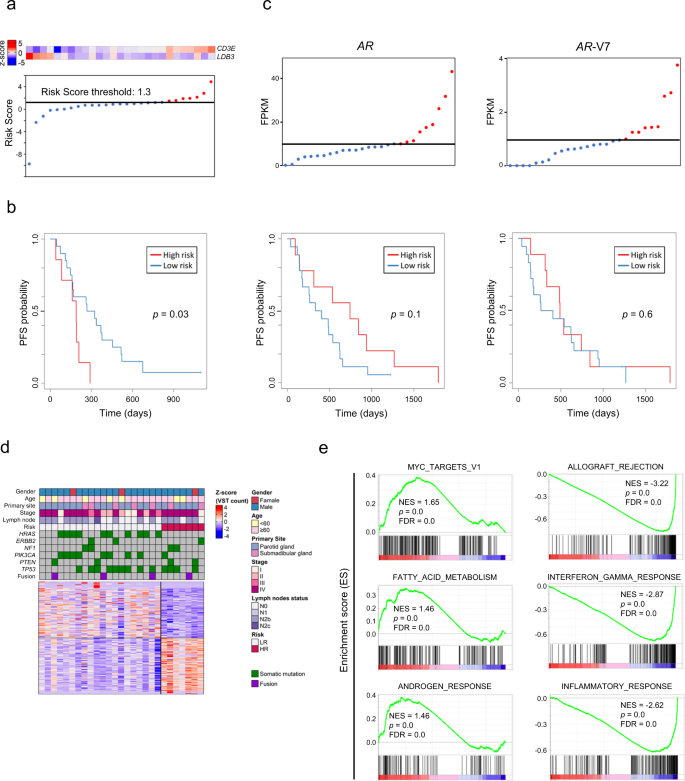
采用前向条件逐步回归和多变量Cox分析,使用14个基因生成预后特征。这个程序建立了一个有两个基因的预后模型,CD3E而且LDB3.我们利用该模型的回归系数构建了一个风险评分,并在第75百分位手动选择了一个合适的阈值(图2)。4).根据基于两基因特征的风险评分定义的高危患者PFS明显更差(p= 0.03, log-rank检验)。4 b).无论是AR-V7变体表达或总数基于“增大化现实”技术表达与患者对CAB治疗的反应相关(图;4摄氏度和补充图。6).
一个双基因表达和风险评分在队列中的分布z分数。红色和浅蓝色分别表示高表达和低表达。所有患者的风险评分按升序绘制,并按阈值(垂直黑线)划分为低风险(蓝色)或高风险(红色)。风险评分阈值为−4.0。b在高危或低危患者中,根据两基因预测特征进行分层的队列中无复发生存的Kaplan-Meier曲线。采用log-rank检验进行评价p值。cKaplan-Meier曲线表示按AR和AR- v7表达分层的队列中的无进展生存期(PFS)。d高危和低危患者前200个差异表达基因的热图,红色和蓝色分别表示高表达和低表达。e在高危肿瘤中,GSEA鉴定出显著不同的基因集为差异过表达或低表达。补充数据4展示了完整的GSEA结果。NES归一化富集评分,FDR错误发现率。
我们使用由双基因标记定义的患者组重复了差异表达基因的分析,以进一步了解高危肿瘤的生物学(图。4 d及补充资料3.).与OS预后特征分析相似,包括“MYC_TARGETS_V1”、“脂肪酸代谢”和“雄激素反应”在内的基因集在高风险肿瘤中显著富集,而包括“同种异体排斥反应”、“干扰素γ反应”和“炎症反应”在内的基因集在低风险组中富集(图2)。4 e及补充资料4).
采用免疫组化法检测ADAMTS1、DSC1、RNF39、CD3和LDB3在肿瘤细胞中的表达(补充图。7).在各自基因中未观察到FPKM与蛋白表达强度或蛋白表达阳性细胞比例的一致性(补充数据5).
讨论
这是第一个利用新一代测序结合临床病理数据的分子分析来确定CAB治疗的预测性生物标志物和SDC的预后生物标志物的研究。我们的分析在67个肿瘤中的55个(82.1%)中确定了RTK/MAPK信号通路的驱动突变。考虑到驱动基因突变是截断型的,SDC可能依赖于这些驱动基因来生长。驱动突变阳性和阴性病例基因突变的最高VAF无差异;因此,后一种情况是真阴性,而不是肿瘤含量低导致的假阴性。先前对31例SDC病例的综合基因组分析也显示~74%的病例中存在驱动突变31.我们的RNA-seq分析确定了RTK融合和ERBB2在多种情况下过度表达。此外,我们精确地描述了涉及组蛋白修饰的基因,如KMT2A / C / D,以及转录因子,ZFHX3而且FOXA1,其表达在我们之前的研究中被确定为良好的预后45.SDC的TMB低于其他实体肿瘤,这与之前的研究一致31.全基因组测序和长读测序的进一步分析可能有助于揭示驱动阴性SDC的转化机制。
RNA-seq的实用性是通过检测融合基因和通过表达分析。融合基因的存在是不良OS的独立标志(HR = 2.6,p= 0.04)。特别是,我们确定了目标FGFR1/2而且NTRK3多种情况下的融合。这是有希望的,有最近的发展和批准的靶向治疗胆管癌FGFR2融合和实体瘤NTRK融合46,47,48,49.RNA-seq对mRNA表达的评估与HER2、AR和EGFR的免疫组化染色密切相关。的表达式CD274在驱动阴性组中上调,这表明PD-L1抑制剂可能是这些患者可行的治疗选择。
双基因特征和表达CD3E而且LDB3,但不是基于“增大化现实”技术,成功地将患者按CAB治疗结果进行分层。CD3E编码CD3-epsilon多肽,形成t细胞受体- cd3复合物,并在偶联抗原识别到几种细胞内信号转导途径中发挥重要作用50.CD3免疫组化证实淋巴细胞中CD3表达,而肿瘤细胞中CD3E RNA表达可能反映了肿瘤免疫环境。浸润的CD8 +细胞毒性T细胞和CD3 + T细胞与良好预后的相关性已在原发性和转移性的不同组织学特征和解剖位置的癌症中得到广泛证实,包括黑色素瘤、大多数鳞状细胞癌、大细胞肺癌和几种类型的腺癌51,52,53,54.epsilon多肽也参与了t细胞的发育。LDB3编码横纹肌中含有PDZ结构域的适配蛋白,通过其LIM结构域将蛋白激酶c介导的信号偶联到细胞骨架55.我们的分析表明,t细胞浸润可能是CAB治疗的预测因子,这与之前研究SDC免疫微环境和新抗原景观的研究一致34,36.
既不CD3E而且LDB3已被报道参与AR信号通路或二氢睾酮的代谢,二氢睾酮是一种有效的AR激动剂。我们的GSEA分析比较了高风险组和低风险组对CAB治疗的影响,揭示了低风险组中雄激素通路相关基因的富集。这表明激素信号依赖可能是治疗的另一个预测性生物标志物。
我们还定义了一个四基因集(ADAMTS1,DSC1,RNF39,IGLL5)用于预测侵袭性SDC。该基因集可以作为一种有价值的预测性生物标志物,用于对可能受益于额外的全身或放射治疗的患者进行分层。这四个基因与多种过程有关,如炎症和细胞粘附。
ADAMTS1是一种表征良好的基质金属蛋白酶相关酶。然而,哺乳动物细胞研究报告了ADAMTS1在癌症发展中的相互矛盾的作用56不同的机制与它的原质/抗肿瘤活性有关,包括调节血管生成、淋巴管生成、细胞增殖、粘附、迁移、降解或与细胞外基质成分的相互作用。例如,ADAMTS1前列腺癌和结直肠癌中启动子高甲基化是否下调57,但在胰腺癌中过表达58,纤维肉瘤59,以及肾细胞癌60.目前,ADAMTS1在SDC进展中的作用尚不清楚。
DSC1在细胞-细胞粘附中起重要作用的是一种桥粒钙粘蛋白吗61.这些钙粘蛋白包括DSC1-3和四种纤凝蛋白。据报道,DSC1参与乳腺癌的进展、转移潜能和细胞粘附过程62.目前,很少有人知道涉及RNF39而且IGLL5在肿瘤发生。
GSEA分析发现高危组中G2/M检查点、E2F转录因子的细胞周期相关靶点和上皮-间充质转化(epithelial-mesenchymal transition, EMT)相关基因表达上调,而低风险组中基因表达上调涉及肌源性分化。在EMT过程中,上皮细胞失去细胞极性和细胞间的黏附,获得迁移和侵袭性,成为间充质干细胞。与细胞周期和EMT相关的富集基因集可能表明侵袭性SDC具有快速细胞生长、高侵袭性和迁移能力。调节ADAMTS1而且DSC1表达可能在SDC细胞的存活和侵袭中起重要作用,并可能解释其与预后不良患者的关系。考虑到基因突变TP53或PTEN也是预后不良的独立标记物,这种遗传和表观遗传变化的组合可能促进SDC的进展和复发。
蛋白质表达与RNA-seq数据之间的不一致是因为RNA-seq量化RNA表达的敏感性可能比免疫组化量化蛋白质表达的敏感性更高。事实上,大多数样本中DSC1、LDB3和RNF39蛋白表达水平过低,免疫组化检测不到,而ADAMTS1在所有样本中表达均为中阳性。CD3蛋白在淋巴细胞中表达,但肿瘤细胞中CD3E RNA的表达可能反映了肿瘤的免疫环境。虽然在这些基因中没有观察到一致性,但我们有信心,我们的RNA-seq适用于RNA表达的评估,因为在HER2、AR和EGFR表达中观察到良好的相关性(补充图。3.).RNA-seq可能是评价蛋白水平低或免疫组化分析不完善的基因表达的较好方法。
本研究的一个主要局限性是,这是一项回顾性研究,没有曲线下面积评估支持生物标志物特征的特异性。因此,应进行独立队列的前瞻性研究,以验证概念的证明。TOP检测的另一个局限性是它不能检测出癌症中存在的所有突变,也不一定能阐明所有的肿瘤特征。因此,需要全基因组测序和全基因组甲基化分析等综合分析来识别更多的新型生物标志物。
总之,我们的发现可能有应用于临床的潜力。表达谱分析可以直接预测CAB的治疗效果和SDC的预后。我们的基因组和转录组分析强调了精确的肿瘤分析对为SDC患者提供更多治疗选择的重要性。
方法
研究设计和患者标本
研究队列包括76名SDC患者,他们于2005年10月至2017年9月期间在日本SDC财团的医院接受了手术切除,包括国际卫生福利大学、三田医院、东京医科大学医院、东京医科大学八公治医学中心、北海道大学、新泻癌症中心医院、庆应义塾大学和东海大学。9例患者因DNA质量差而被排除在外,其余67例患者进行了分析。Otsuka等人此前报道了几个病例。9, Takase等。16, Masubuchi等。17,志村等。41, Fushimi等。20.高桥等。29, Okada等人。63.一位专门研究唾液腺肿瘤的认证病理学家(TN)根据当前世界卫生组织的分类标准回顾了SDC的组织学特征。所有患者均获得手术切除肿瘤的新鲜冷冻标本或福尔马林固定石蜡包埋(FFPE)。本研究获得了国立癌症中心伦理委员会(No. 2019-271)、国际卫生福利大学、三田医院(No. 5-19-6)、东京医科大学(No. 2019-271)的批准。北海道大学医学院和医学研究生院(No. 017-0487),庆应义塾大学医学院(No. 20120083),东海大学医学院(No. 20R-204),东京医科大学八公町医学中心(No. 20R-204)。SH2563)和新泻癌症中心医院(No. 2021-300)。除因失去随访或注册时死亡而无法联系到的受试者外,所有受试者都提供了书面知情同意书。在这些情况下,每个参与机构的机构审查委员会批准将现有的组织样本用于研究目的。本研究没有使用来自选择不参与的患者的样本。
免疫组织化学和荧光原位杂交(FISH)
如前所述,进行免疫组化和FISH16,41.简单地说,将FFPE肿瘤组织切成3 μm厚的切片,利用基于聚合物的热介导抗原提取检测系统,使用1:400稀释的抗HER2 (Polyclonal, cat# A0485, Agilent Technologies, Santa Clara, CA)的一抗,AR(克隆AR441, cat# PM109AA, BIOCARE Medical LLC, CA),未稀释的EGFR(克隆31G7, cat# 423701, NICHIREI BIOSCIENCES INC, Tokyo, Japan), 1:50稀释的PD-L1(克隆22C3, cat# M3653, Agilent Technologies),ADAMTS1 (cat# 12749-1-AP, Proteintech, Rosemont, IL) 1:50稀释,DSC1(克隆A-4, cat# sc-398590, Santa Cruz Biotechnology, Dallas, TX) 1:100稀释,RNF39 (cat# HPA047115, Atlas Antibodies, Bromma,瑞典)1:200稀释,CD3(克隆SP-7, cat# 413601, Nichirei Biosciences,东京,日本)1:100稀释,LDB3 (cat# 11004-1-AP, Proteintech) 1:100稀释。二氨基联苯胺用于检测抗原-抗体反应。所有条件均采用适当的阳性和阴性对照。ERBB2使用PathVysion HER-2 DNA探针试剂盒(cat# 02J01-031, Abbott Molecular, Des Plaines, IL)进行扩增评估。对于PD-L1 IHC 22C3 pharmDx检测,使用Dako Autostainer Link 48平台和自动染色方案对载玻片进行染色,病理学家a . U.和T. H.使用光学显微镜对每个样本中阳性肿瘤细胞的百分比进行评分。
HER2状态评估
HER2阳性定义为免疫组化3+或ERBB2根据美国临床肿瘤学会/美国病理学家学院乳腺癌指南进行放大64.在Her2免疫组化中,3+染色定义为环状膜染色,完整而强烈,肿瘤细胞出现频率为>10%。为ERBB2用FISH分析预测拷贝数,对100个不重叠、完整的间期肿瘤核进行4′,6-二氨基氨基-2-苯基吲哚(DAPI)染色。的ERBB2基因(红色信号)和CEP17(绿色信号)评估每个细胞核的拷贝数。ERBB2放大时是加注平均ERBB2/CEP17在所有核中比值≥2.0ERBB2信号形成了紧密的基因簇。
AR、EGFR、ADAMTS1、DSC1、RNF39、CD3和LDB3的免疫组化评估
当≥20%的肿瘤细胞核呈强染色时,1例被认为是AR阳性。EGFR免疫染色细胞百分比从0到3+进行评分:0,0%;1 + 1 - 10%;2 + 11 - 30%;3+ >30%EGFR得分3+为阳性。对ADAMTS1、DSC1、RNF39、CD3、LDB3染色强度及阳性细胞比例进行评估。染色强度从0到3+分为:0,阴性;1+,轻度阳性;2+,中度阳性; 3+, strongly positive.
DNA测序与TOP癌症基因面板突变呼叫和拷贝数分析
新鲜冷冻或FFPE SDC标本用TOP panel version 3进行分析44.该方法评估了464个基因的核苷酸变异和插入/缺失,以计算TMB并推断拷贝数变异。使用GeneRead DNA FFPE试剂盒(Qiagen, Hilden, Germany)从FFPE样本中提取基因组DNA,每个DNA样本使用Agilent Kit (v6) (Agilent Technologies)进行目标片段富集。使用HiSeq2500 (Illumina)进行了下一代测序,使用配对端选项,测序reads使用BWA独立地与人类参考基因组(hg38)对齐65,鲍蒂http://bowtie-bio.sourceforge.net/bowtie2/index.shtml),以及NovoAlign (http://www.novocraft.com/products/novoalign/).体细胞突变使用MuTect (http://www.broadinstitute.org/cancer/cga/mutect), SomaticIndelDetector (http://www.broadinstitute.org/cancer/cga/node/87)和VarScan (http://varscan.sourceforge.net).突变分析的排除标准为(i)读取深度<20或VAF <0.1;(ii)突变仅由一条基因组链支持;或(iii)它们是1000个基因组计划数据集(http://www.internationalgenome.org/)或我们的内部资料库。基因突变用SnpEff标记(http://snpeff.sourceforge.net).使用我们内部的管道评估拷贝数,计算logR比(LRR),如下所示:(i)使用1000基因组计划数据库中选择的纯合(VAF,≤0.05或≥0.95)或杂合(VAF, 0.4-0.6) SNP;(ii)所选SNP位置的正常和肿瘤读数深度根据距离该位置约100 bp的GþC百分比进行计数和调整66;(iii)计算LRR = log2(t我/n我),n我而且t我为SNP位置的正常深度和肿瘤调节深度;(iv)每个代表性LRR由SNP位置周围的移动窗口(1 Mb)的中位数确定。对整个基因组的每个区域分别测定主等位基因和小等位基因拷贝数的LRR值。的p根据GISTIC所使用的算法,通过置换试验(10万次迭代)从lrr中确定各自基因组区域的增益或损失值67,问数值使用R包计算问值(http://github.com/jdstorey/qvalue).
转录组测序,表达分析,融合基因和外显子跳跃检测
使用RNA- bee (Tel-Test Inc., Gainesville, FL)从新鲜、冷冻或FFPE样品中提取总RNA,并用DNase I (Thermo Fisher Scientific, Waltham, MA)处理。RNA-seq文库使用TruSeq RNA Access文库Prep Kit (Illumina, San Diego, CA)制备。使用HiSeq 2500 (Illumina)从每个簇的两端进行测序。RNA-seq使用TopHat (v2.0.9;https://ccb.jhu.edu/software/tophat/index.shtml).基因表达用Cufflinks (v2.1.1;http://cole-trapnell-lab.github.io/cufflinks)和基因融合的研究使用了化解管道(https://bitbucket.org/dranew/defuse).选择符合以下标准的融合基因:COSMIC中报道的融合基因或RTK/MAPK信号通路中激酶的融合基因,融合分裂reads为>10。外显子跳跃性分析使用了之前研究中开发的内部管道68(i)使用Burrows-Wheeler Aligner和Bowtie2将RNA-seq读取到hg38和NCBI参考序列(RefSeq);(ii)在映射的RefSeq数据中检测到跳过的外显子;动态创建虚拟转录组序列;(iv)对齐RNA-seq读取候选转录组序列;并且(v)基于带断点的读取确定了外显子跳过候选。
桑格测序
使用3130xl遗传分析仪(Thermo Fisher Scientific)进行毛细管测序时,使用10 ng模板cDNA制备的PCR产物进行扩增FGFR2-CCDC6,FGFR2-KIAA1217,FGFR2-NOL4,ETV6-NTRK3,MAPK14-ZFAND3,NFIX-MAST1,SLC45A3-ELK4,TMCC1-PLXND1,ERBB2-CTTN使用GoTaq G2热启动主混合绿色(Promega, Madison, WI),按照制造商的说明,使用以下底漆:5 ' -GCAGTTGGTAGAAGACTTGGATCG-3 '和5 ' -GCAAGGGTTTCTTTCTCCTTCTGC-3 '用于FGFR2-CCDC6, 5 ' -GCAGTTGGTAGAAGACTTGGATCG-3 '和5 ' - gacgtctctgataaatgctcacc -3 '为FGFR2-KIAA1217, 5 ' -GCAGTTGGTAGAAGACTTGGATCG-3 '和5 ' -ACAGCTGATGTTGAGTAAGTGGCC-3 '为FGFR2-NOL4, 5 ' -GAACCACATCATGGTCTCTGTCTC-3 '和5 ' -CAGGAAGACCTTTCCAAAGGCTC-3 'ETV6-NTRK3, 5 ' -GAACCTACAGAGAACTGCGGTTAC-3 '和5 ' -TACTTGGAGCGGAATCATCGTCTG-3 'MAPK14-ZFAND3, 5 ' -GCCACATCACATTGGAGTCACAATC-3 '和5 ' - cagcgctccaatattcttcagcagc -3 'NFIX-MAST1, 5 ' -TTCACCTTCTCAGCCCTGCAGATC-3 '和5 ' -TTCTGCAGGAGCTGAAGAAGGAAC-3 'SLC45A3-ELK4, 5 ' -TGTTCCATCCACAGAGTCTGTGTG-3 '和5 ' -ACTCCTGTGTTTCCAACCAGTCTC-3 'TMCC1-PLXND1, 5 ' -ATAACACCCACCTCTGCTTCGTG-3 '和5 ' -CATACTTCCCGCCGAATCCTTTG-3 'ERBB2-CTTN.
签名生成和统计分析
生存风险评估的OS时间定义为从开始治疗到因任何原因死亡或最后一次随访日期之间的时间。OS分析选择满足以下条件的基因:总样本中超过80%(49/61)的基因表达为>0,sd为>1。在这个初始过滤之后,p计算值,9个基因p≤0.002,并进一步分析队列中多变量Cox分析的正向条件逐步回归。用于CAB风险评估的PFS时间定义为从开始CAB治疗之日起至疾病进展或死亡之日。CAB处理的PFS分析选择了满足以下条件的基因:样本中表达量最高的基因为>50。在这个初始过滤之后,p计算了14个基因p≤0.002,进一步用于构建预测模型。候选基因在逐步多元Cox回归分析中进行拟合,以评估它们对队列生存预测的相对贡献。与生存相关的基因被包括在预后特征中。根据多元Cox回归分析中估计的回归系数,计算预测OS和PFS的预后风险评分,与以往研究相似66.所有统计分析均采用R软件(3.5.1版本;https://www.r-project.org/)及相关套装。使用“Survival”(v2.44.1.1)包进行生存分析和Cox回归分析。使用Kaplan-Meier方法分析OS和PFS,根据风险评分或驱动突变亚型使用log-rank检验评估曲线差异。GSEA采用Java GSEA软件(http://software.broadinstitute.org/gsea/index.jsp) (v2.2.4)。
报告总结
有关研究设计的进一步资料,请参阅自然研究报告摘要链接到这篇文章。
数据可用性
我们已将本文中所有新的和未发表的DNA和RNA测序数据的原始测序数据存入日本DNA数据库的日本基因型-表型档案,登录号为hum0094,登录号为JGAS000534。
代码的可用性
没有未报告的自定义计算机代码或算法用于生成本文的结果。所有源代码的引用都包含在材料和方法中。
参考文献
Nagao, T., Licitra, L., Loening, T., Vielh, P. & Williams, M.D.《WHO头颈部肿瘤分类》第4版(编辑L - naggar, a.k., Chan, J.K.C, Grandis, j.r., Takata, T. & slootg, P.J.) 173-174(IARC, 2017)。
Nakaguro, M.等。涎管癌:组织学、细胞学、分子生物学和治疗的最新进展。癌症Cytopathol。128, 693-703(2020)。
D 'Heygere, E., Meulemans, J. & Vander Poorten, V.涎管癌。咕咕叫。当今。Otolaryngol。头。脖子杂志。26, 142-151(2018)。
Boukheris, H., Curtis, R. E., Land, C. E. & Dores, g.m. 1992 - 2006年根据世界卫生组织分类的大唾液腺癌发病率:美国一项基于人群的研究。癌症的论文。Biomark。上一页。18, 2899-2906(2009)。
Tamaki, T.等人。日本罕见癌症的负担:“罕见治疗”定义的应用癌症的论文。38, 490-495(2014)。
Jayaprakash, V.等。浸润性涎管癌的生存率和预后因素:来自监测、流行病学和最终结果数据库的228例分析头。脖子36, 694-701(2014)。
王志强,王志强,等(编)。涎管癌,见WHO头颈部肿瘤分类(第4版). 173 (IARC, Lyon, France, 2017)。
布恩等人。雄激素受体阳性晚期涎管癌的雄激素剥夺治疗:荷兰全国35例患者的病例系列。头。脖子40, 605-613(2018)。
大冢,K.等。涎管癌的临床结果和预后因素:141例患者的多机构分析。安。Surg.杂志。23, 2038-2045(2016)。
吉尔伯特,m.r.等人。75例涎管癌20年临床分析。JAMA Otolaryngol。头。脖子杂志。142, 489-495(2016)。
奥斯本,V.等。使用国家癌症数据库的涎管癌的特征、治疗和结果。口服。肿瘤防治杂志。71, 41-46(2017)。
Williams, m.d.等。激素和生长因子受体在涎管癌中的差异表达:生物学意义和在患者治疗分层中的潜在作用。点。J.外科。31, 1645-1652(2007)。
Hosal, a.s., Fan, C, Barnes, L. & Myers, e.n.涎管癌。Otolaryngol。头。脖子杂志。129, 720-725(2003)。
Nasser, S. M., Faquin, W. C. & Dayal .涎腺肿瘤中雄激素、雌激素和孕激素受体的表达。恶性唾液腺肿瘤中雄激素受体的频繁表达。点。j .中国。病理学研究。119, 801-806(2003)。
范春燕,范志强,陈志强,陈志强,陈志强。涎管癌组织中雄激素受体、表皮生长因子受体和转化生长因子α的表达。拱门。Otolaryngol。头。脖子杂志。127, 1075-1079(2001)。
Takase, S.等。涎管癌的生物标志物免疫谱:临床病理和预后意义与修订分类的评估。Oncotarget8, 59023-59035(2017)。
Masubuchi, T.等人。雄激素受体、HER2、Ki-67、EGFR在涎管癌中的表达及其临床病理意义J. clinin。肿瘤防治杂志。20., 35-44(2015)。
Lewis, J. E, McKinney, b.c., Weiland, L. H., Ferreiro, J. A. & Olsen, K. D.涎管癌。对26例患者的临床病理及免疫组化进行了分析。癌症77, 223-230(1996)。
维斯库斯,P. V., Price, K. A., Garcia, J. J., schembrii - wismayer, D. J. & Chintakuntlawar, A. V.雄激素受体阳性复发或转移性唾液腺癌患者一线雄激素剥夺治疗与化疗的比较——一项回顾性研究。肿瘤防治杂志。9, 701(2019)。
Fushimi, C.等。联合雄激素阻断治疗雄激素受体阳性转移性或局部晚期不可切除唾液腺癌患者的前瞻性II期研究安。肿瘤防治杂志。29, 979-984(2018)。
Locati, L. D.等。雄激素剥夺治疗在转移性/复发性雄激素受体阳性唾液腺癌患者中的临床活动。头。脖子38, 724-731(2016)。
Uijen, m.j.m.等。复发性或转移性涎管癌的系统治疗:一项系统综述。癌症治疗。牧师。89, 102069(2020)。
Antonarakis, E. S.等。AR-V7和前列腺癌对enzalutamide和abiraterone的耐药性。心血管病。j .地中海。371, 1028-1038(2014)。
Cappelletti, V.等。液体活检治疗唾液腺管癌(SDC): ARv7在循环肿瘤细胞中的表达。安。肿瘤防治杂志。29, 1598-1600(2018)。
Dalin, m.g., Watson, p.a., Ho, A.L.和Morris, L.G.涎腺癌中的雄激素受体信号。癌症(巴塞尔)9,https://doi.org/10.3390/cancers9020017(2017)。
van Boxtel, W.等人。涎管癌患者雄激素剥夺治疗的临床疗效预测。癌症146, 3196-3206(2020)。
Lassche, G.等人。计划联合雄激素阻断的雄激素受体阳性涎管癌患者大队列的预测和预后生物标志物鉴定。癌症(巴塞尔)13,https://doi.org/10.3390/cancers13143527(2021)。
Saigusa, N.等人。EZH2和H3K27me3表达作为涎管癌患者临床结局预测因子的作用:一项大系列研究,强调与雄激素阻断联合her2靶向治疗的相关性。肿瘤防治杂志。11, 779882(2021)。
高桥,H.等。曲妥珠单抗和多西他赛在人表皮生长因子受体2阳性涎管癌患者中的II期试验。j .中国。肿瘤防治杂志。37, 125-134(2019)。
Limaye, S. A.等。曲妥珠单抗治疗涎管癌。肿瘤学家18, 294-300(2013)。
大林,m.g.等。涎管癌的综合分子特征揭示了其作用靶点和与大汗腺乳腺癌的相似性。中国。癌症Res。22, 4623-4633(2016)。
罗斯,J. S.等人。转移性和复发性唾液腺癌的全面基因组图谱与肿瘤类型相关,并揭示了靶向治疗的新途径。安。肿瘤防治杂志。28, 2539-2546(2017)。
米勒,检察官等。涎管癌的分子模式确定预后亚组。中华病理学国防部。.https://doi.org/10.1038/s41379-020-0576-2(2020)。
林克斯维勒,等人。侵袭性唾液腺癌的免疫微环境和新抗原景观因亚型而异。中国。癌症Res。26, 2859-2870(2020)。
Gargano, s.m.等人。综合分子分析发现涎管癌的新治疗靶点。癌症地中海。8, 7322-7329(2019)。
Alame, M.等人。涎管癌的分子图景和微环境揭示了新的治疗机会。开展10, 4383-4394(2020)。
王,K.等。149例涎管癌,癌前多形性腺瘤和腺癌,未明确显示可操作的基因组改变。中国。癌症Res。22, 6061-6068(2016)。
Griffith, C. C., Seethala, R. R., Luvison, A., Miller, M. & Chiosea, s.i. .涎管癌中PIK3CA突变和PTEN缺失。点。J.外科。37, 1201-1207(2013)。
格吕内瓦尔德等人。腮腺癌靶向下一代测序揭示遗传异质性。Oncotarget6, 18224-18237(2015)。
Chiosea, s.i.等。大汗腺涎管癌的分子特征。点。J.外科。39, 744-752(2015)。
志村,T.等。涎管癌中基因改变和关键潜在作用靶点表达的预后和组织发生作用。Oncotarget9, 1852-1867(2018)。
Kim, Y.等人。涎管癌的整合基因组分析。科学。代表。10, 14995(2020)。
Karpinets, T.V.等。常见唾液腺癌的全基因组测序:亚型限制和共享的遗传改变。中国。癌症Res.https://doi.org/10.1158/1078-0432.CCR-20-4071(2021)。
Kohsaka, S.等。从福尔马林固定的石蜡包埋标本中富集目标对癌症分子谱的综合分析。癌症科学。110, 1464-1479(2019)。
Urano, M.等。FOXA1的高表达与涎管癌的良好预后相关:一项142例的研究。组织病理学73, 943-952(2018)。
关于alfa, G. K.等人。Pemigatinib用于先前治疗的局部晚期或转移性胆管癌:一项多中心、开放标签的2期研究柳叶刀杂志。21, 671-684(2020)。
德隆,A.等人。Larotrectinib在成人和儿童TRK融合阳性癌症中的疗效。心血管病。j .地中海。378, 731-739(2018)。
莱奇,t.w.等人。Larotrectinib用于含NTRK基因融合的儿童实体肿瘤:1期来自一项多中心、开放标签、1/2期研究。柳叶刀杂志。19, 705-714(2018)。
Doebele, R. C.等。Entrectinib用于晚期或转移性NTRK融合阳性实体瘤患者:三项1-2期试验的综合分析柳叶刀杂志。21, 271-282(2020)。
Barber, E. K, Dasgupta, J. D, Schlossman, S. F, Trevillyan, J. M. & Rudd, C. E. CD4和CD8抗原与磷酸化CD3复合物的蛋白酪氨酸激酶(p56lck)偶联。国家科学院学报美国86, 3277-3281(1989)。
Galon, J.等。人结直肠肿瘤内免疫细胞的类型、密度和位置可预测临床结果。科学313, 1960-1964(2006)。
弗里德曼,W. H., Pages, F., Sautes-Fridman, C. & Galon, J.人类肿瘤中的免疫结构:对临床结果的影响。Nat. Rev. Cancer12, 298-306(2012)。
傅德曼,W. H., Zitvogel, L., Sautes-Fridman, C. & Kroemer, G.癌症预后和治疗中的免疫结构。Nat. Rev. clinin。肿瘤防治杂志。14, 717-734(2017)。
吕妮,安吉尔,H. K. & Galon, J.免疫结构和免疫评分在癌症预后和治疗效果中的作用。Nat. Rev. Cancer20., 662-680(2020)。
Pathak, P.等人。肌病相关的LDB3突变通过PKCalpha和TSC2-mTOR下调导致z盘解体和蛋白聚集。Commun。医学杂志。4, 355(2021)。
Cal, S. & Lopez-Otin, C. ADAMTS蛋白酶与癌症。矩阵杂志。44-46, 77-85(2015)。
谭ide, A., Ricciardelli, C. & Russell, D. L.金属蛋白酶ADAMTS1:其在肿瘤发生和转移途径中的作用的全面综述。癌症133, 2263-2276(2013)。
Masui, T.等。冰毒1和冰毒2在胰腺癌中的表达。中国。癌症Res。7, 3437-3443(2001)。
Casal, C.等。ADAMTS1有助于在可塑性肿瘤细胞中获得内皮样表型。癌症Res。70, 4676-4686(2010)。
Wen, Y.C.等。褪黑素触发的ADAMTS1转录后和翻译后修饰协调延缓肾细胞癌的肿瘤发生和转移。j .松果体。Res。69, e12668(2020)。
Faktor, J.等人。desmocolin -1和儿茶酚- o -甲基转移酶与乳腺癌细胞迁移和转移相关蛋白的蛋白质组学鉴定和验证。蛋白质组学19, e1900073(2019)。
对桥粒调节和天疱疮起泡作为桥粒重塑疾病的新见解。高雄市J.医学科学。29, 1-13(2013)。
冈田,T.等。卡铂和多西他赛治疗唾液腺癌:一项回顾性研究。体内33, 843-853(2019)。
沃尔夫,a.c.等。乳腺癌中人类表皮生长因子受体2检测的建议:美国临床肿瘤学会/美国病理学家学院临床实践指南更新。j .中国。肿瘤防治杂志。31, 3997-4013(2013)。
李,H. & Durbin R.快速和准确的短读对齐与Burrows-Wheeler变换。生物信息学25, 1754-1760(2009)。
Kohsaka, S.等。从日本从不吸烟者或轻度吸烟者肺腺癌的综合分析中鉴定新型cd74 - nrg2 α融合。j . Thorac。肿瘤防治杂志。15, 948-961(2020)。
Mermel, C. H.等。GISTIC2.0在人类癌症中促进了对焦点体细胞拷贝数改变靶点的敏感和自信的定位。基因组医学杂志。12, r41(2011)。
Kohsaka, S.等。纤维瘤的综合分子和临床病理分析。欧元。j .癌症145, 109-120(2021)。
确认
作者要感谢Y. Yamamoto和A. Maruyama-Shiino的技术援助。本研究由日本癌症研究与开发机构(AMED)资助的癌症研究与治疗进化项目(P-CREATE)资助,资助号为JP20cm0106502,以及日本医学研究与开发机构(AMED)资助的创新癌症控制实践研究(JP20ck0106536)。这项工作也得到了科学研究资助计划(C)的支持,资助号为18K09386和20K07417。
作者信息
作者及隶属关系
贡献
s.k., y.t.和H.M.构思了这个项目并设计了研究。s.k.、t.u.和H.M.开发了这个方法。S.K.和Y.S.进行了实验。S.K, T.U和S.K分析和解释了数据。Y.T硕士。梅西百货(M.N:行情,H.H。n ., S.K, K.T, T.T。H.O。T.K。好的,H.T, D.K,出口,t,其子as,入境官员,见反面,廖曜生,Y.I, Y.W,其子as, K.E,廖曜生,嘅曼联,,12,梁龙骥,Y.U, T.H。廖曜生,t,和T.N.提供行政、技术、或材料的支持。s.k., y.t., m.a., m.n., t.n.和H.M.根据所有作者的反馈编写和编辑了本文。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
Kohsaka, S, Tada, Y, Ando, M。et al。通过综合分子分析鉴定涎管癌的新型预后和预测生物标志物。npj大纲。Onc。6, 82(2022)。https://doi.org/10.1038/s41698-022-00324-1
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41698-022-00324-1