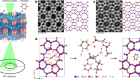
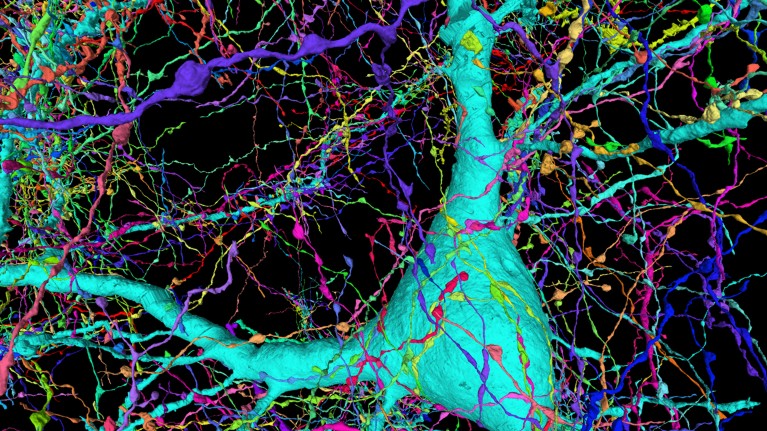
一立方毫米听起来并不多。但在人脑中,这一体积的组织包含约5万根神经“线”,由1.34亿个突触连接。杰夫·利希特曼想追踪所有人。
为了获得原始数据,他使用了一种被称为串行薄切片电子显微镜的方法,在11个月的时间里对数以千计的组织切片进行成像。但是数据集非常庞大,达到了1.4拍字节——相当于大约200万张cd - rom——对于研究人员来说,单靠他们自己的力量是远远不够的。“人类根本不可能手动追踪所有的电线,”马萨诸塞州剑桥市哈佛大学的分子和细胞生物学家Lichtman说。“地球上没有足够多的人真正有效地完成这项工作。”
这在连接组学(研究大脑结构和功能连接的学科)和其他生物科学中都是常见的重复,在这些科学中,显微镜技术的进步创造了大量的成像数据。但当人力资源失败时,计算机可以介入,尤其是深度学习算法,它经过优化,可以从大型数据集中梳理出模式。
麻省理工学院和哈佛大学布罗德研究所(Broad Institute of MIT and Harvard)的计算生物学家贝丝·西米尼(Beth Cimini)说:“在过去的几年里,我们真的经历了深度学习工具的寒武纪大爆发。”
深度学习是一种人工智能(AI)技术,它依赖于多层人工神经网络,灵感来自于大脑中神经元相互连接的方式。由于这些算法基于黑箱神经网络,因此也有其局限性。这包括依赖大量数据集来教会网络如何识别感兴趣的特征,以及有时难以理解的生成结果的方法。但大量快速增长的开源和基于网络的工具正使它比以往任何时候都更容易开始(参见“向深度学习迈出一步”)。
以下是深度学习对生物图像分析产生深远影响的五个领域。
大规模——神经
深度学习使研究人员能够从果蝇、老鼠甚至人类身上生成越来越复杂的连接体。这些数据可以帮助神经科学家了解大脑是如何工作的,以及大脑结构在发育和疾病中是如何变化的。但神经连接并不容易绘制。
2018年,利希特曼与加利福尼亚州山景城谷歌的连接组学主管维伦·贾恩(Viren Jain)合作,后者正在为他的团队的AI算法寻找合适的挑战。
“连接组学中的图像分析任务非常困难,”Jain说。“你必须能够跨越很远的距离追踪这些细丝,细胞的轴突和树突,而传统的图像处理方法犯了太多的错误,它们基本上无法完成这项任务。”这些导线可以比一微米还细,可以延伸到数百微米甚至毫米的组织中。深度学习算法提供了一种方法,使连接组学数据的分析自动化,同时仍能达到较高的准确性。
如何绘制基因活动的空间图——精确到细胞水平
在深度学习中,研究人员可以使用包含感兴趣特征的注释数据集来训练复杂的计算模型,以便快速识别其他数据中的相同特征。德国海德堡欧洲分子生物实验室(European Molecular Biology Laboratory)的计算机科学家安娜•克雷舒克(Anna Kreshuk)表示:“当你在进行深度学习时,你会说,‘好吧,我就举个例子,你把一切都弄明白’。”
但即使使用深度学习,利希特曼和贾恩在试图绘制人类皮层片段时也面临着艰巨的任务1.仅为5000个左右极薄的组织切片成像就花了326天。两名研究人员花了大约100个小时手工标注图像,并追踪神经元,创建“基本真相”数据集来训练算法,这种方法被称为监督机器学习。然后,经过训练的算法自动将图像拼接在一起,识别神经元和突触,生成最终的连接组。
Jain的团队为解决这一问题投入了大量的计算资源,包括数千个张量处理单元(tpu),这是谷歌内部的相当于专门为神经网络机器学习而构建的图形处理单元(gpu)。Jain说,处理数据需要在几个月的时间里达到100万TPU小时量级,之后人类志愿者在协作过程中对连接组进行校对和更正,“有点像谷歌文档”,Lichtman说。
他们说,最终的结果是,在任何物种中,在这种详细程度上重建的这种数据集是最大的。尽管如此,它只占人脑的0.0001%。但随着算法和硬件的改进,研究人员应该能够绘制更大的大脑区域,同时有分辨率发现更多的细胞特征,如细胞器甚至蛋白质。“在某些方面,”Jain说,“我们只是触及了从这些图像中可能提取到的东西的表面。”
虚拟组织学
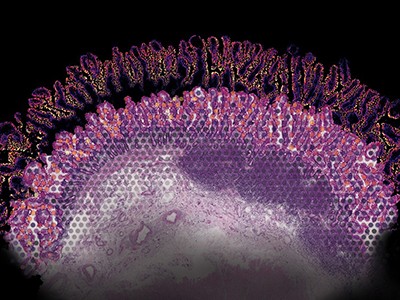
组织学是医学上的一个关键工具,并被用于在化学或分子染色的基础上诊断疾病。但这很费力,整个过程可能需要几天甚至几周的时间才能完成。活组织切片成薄片,染色以显示细胞和亚细胞特征。然后病理学家阅读切片并解释结果。Aydogan Ozcan认为他可以加速这个过程。

Python升级:新的图像工具可以可视化复杂的数据
作为加州大学洛杉矶分校(University of California, Los Angeles)的电子和计算机工程师,Ozcan训练了一个定制的深度学习模型,通过向它展示同一切片的数万个未染色和染色版本的例子,并让模型计算出它们之间的差异,从而对组织切片进行染色计算。
虚拟染色几乎是即时的,经过认证的病理学家发现几乎不可能将结果图像与常规染色图像区分开来2.Ozcan还表明,该算法可以在几秒钟内复制乳腺癌生物标志物HER2的分子染色,而在组织实验室中,这一过程通常需要至少24小时。一个由三名经委员会认证的乳腺病理学家组成的小组评价这些图像的质量和准确性与传统的免疫组化染色相当3..
Ozcan的目标是将虚拟染色商业化,他希望在药物开发中看到应用。但他说,通过消除对有毒染料和昂贵染色设备的需求,该技术还可以增加世界范围内组织学服务的可及性。
细胞的发现
如果你想从细胞图像中提取数据,你必须知道细胞在图像中的实际位置。
研究人员通常通过在显微镜下观察细胞或在软件中逐个图像勾勒出细胞的轮廓来执行这个过程,称为细胞分割。位于帕萨迪纳市的加州理工学院的计算生物学家摩根•施瓦茨(Morgan Schwartz)正在开发用于生物图像分析的深度学习工具,他说:“最能描述人们一直在做的事情的词是‘苦心经营’。”但随着成像数据集变得越来越大,这些艰苦的方法正在碰壁。“如果没有自动化过程,你就无法分析其中一些实验。”
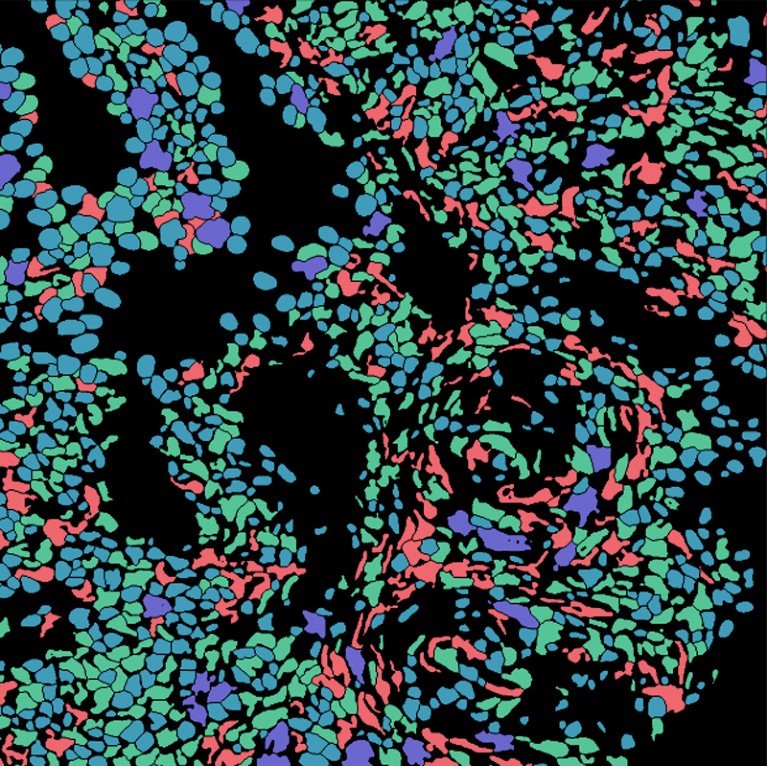
基于谱系的分割揭示了人类怀孕期间子宫内膜细胞的形状。图片来源:n.f. Greenwald等.生物科技自然》。40, 555-565(2022)。
施瓦茨的研究生导师、生物工程师大卫·范·瓦伦(David Van Valen)创建了一套人工智能模型,可以在deepcell.org上找到,用来从活细胞和保存的组织的图像中计算和分析细胞和其他特征。Van Valen与包括加利福尼亚斯坦福大学癌症生物学家Noah Greenwald在内的合作者合作,开发了一种名为Mesmer的深度学习模型,可以快速、准确地检测不同组织类型的细胞和细胞核4.Van Valen说:“如果你有需要处理的数据,现在你只需要上传它们,下载结果,并在门户网站或使用其他软件包中可视化。”
据格林沃尔德说,研究人员可以利用这些信息来区分癌组织和非癌组织,并寻找治疗前后的差异。他说:“你可以观察基于图像的变化,以更好地了解为什么有些病人有反应或没有反应,或者识别肿瘤的亚型。”
蛋白质定位定位
人类蛋白质图谱项目利用了深度学习的另一个应用:细胞内定位。“几十年来,我们一直在生成数百万张图像,勾勒出人体细胞和组织中的蛋白质表达,”斯坦福大学生物工程师、该项目的联合经理艾玛·伦德伯格(Emma Lundberg)说。起初,该项目手动注释这些图像。但由于这种方法无法长期持续,伦德伯格转向了人工智能。

NatureTech中心
伦德伯格首先将深度学习与公民科学结合起来,让志愿者在玩大型多人游戏《星战前夜》(EVE Online)时对数百万张图像进行注释5.在过去几年里,她转向了一种只面向人工智能的众包解决方案,发起了Kaggle挑战——科学家和人工智能爱好者在其中竞争完成各种计算任务——奖金为3.7万美元和2.5万美元,目的是设计有监督的机器学习模型,对蛋白质图谱图像进行标注。伦德伯格说:“后来的Kaggle挑战让玩家们大吃一惊。获胜的模型比Lundberg之前在蛋白质定位模式的多标签分类方面的努力高出约20%,并且可以在细胞系中推广6.她补充说,他们做到了以前发表的模型没有做到的事情,那就是准确地对存在于多个细胞位置的蛋白质进行分类。
伦德伯格说:“我们已经证明,一半的人类蛋白质定位于多个细胞隔层。”位置很重要,因为相同的蛋白质在不同的地方可能表现不同。她说:“知道一种蛋白质是在细胞核里还是在线粒体里,有助于了解它的许多功能。”

注释用于DeepLabCut训练的鱼。图片来源:J. Laueret al。自然方法19, 496-504(2022)。(Cc / 4.0)
追踪动物行为
位于日内瓦洛桑的瑞士联邦理工学院校园生物技术中心的神经科学家Mackenzie Mathis长期以来一直对大脑如何驱动行为感兴趣。她开发了一个名为DeepLabCut的程序,使神经科学家能够从视频中跟踪动物的姿势和精细动作,将“猫视频”和其他动物的记录转化为数据7.
DeepLabCut提供了一个图形用户界面,这样科学家就可以上传和标记他们的视频,并在点击一个按钮时训练一个深度学习模型。今年4月,马西斯的团队扩展了该软件,可以同时估计多种动物的姿势,这对人类和人工智能来说通常都是一个挑战8.
将多动物DeepLabCut应用到绒猴身上,研究人员发现,当这些动物靠近时,它们的身体是对齐的,它们倾向于看向相似的方向,而当它们分开时,它们倾向于面对彼此。马西斯说:“这是一个很好的例子,姿势确实很重要。”“如果你想了解两只动物是如何相互作用的,如何观察对方或观察世界。”

 如何绘制基因活动的空间图——精确到细胞水平
如何绘制基因活动的空间图——精确到细胞水平 Python升级:新的图像工具可以可视化复杂的数据
Python升级:新的图像工具可以可视化复杂的数据 深度学习挑战肿瘤
深度学习挑战肿瘤 深度学习推动了一场运动跟踪革命
深度学习推动了一场运动跟踪革命 生物深度学习
生物深度学习 NatureTech中心
NatureTech中心