摘要gydF4y2Ba
大量可用的肝细胞体外模型为研究人员提供了选择肝细胞样细胞(HLCs)用于特定研究目标的可能性。然而,直接比较肝细胞模型目前具有挑战性。我们系统地搜索了文献并比较了不同的HLCs,但报道的功能仅限于肝功能的一小部分。为了进行更全面的比较,我们开发了一种算法来比较来自肝细胞、胆道细胞、成纤维细胞和多能干细胞的HLCs与原代人肝细胞(PHHs)的研究的转录组数据。这表明没有HLC覆盖完整的肝脏转录组,突出了HLC选择的重要性。无论采用何种方案,来自肝细胞的HLCs与phh的转录相似性最高,而成纤维细胞和PSC衍生的HLCs的质量取决于所使用的方案。最后,我们开发并验证了一个web应用程序(HLCompR),可以比较特定的路径和添加新的HLCs。总之,我们对HLCs的全面转录组比较可以为特定的研究问题选择HLCs,并可以指导培养条件的改进。gydF4y2Ba
简介gydF4y2Ba
为了准确地研究人体肝脏的生理和病理,体外模型必须忠实地复制体内肝脏功能。这些包括毒素的消除,血浆蛋白和胆汁的产生和分泌,以及碳水化合物、氨基酸和脂类的代谢稳态。这些过程大部分是由肝细胞完成的,这些上皮细胞占肝脏细胞数量的60%和体积的80%gydF4y2Ba1gydF4y2Ba.因此,追求具有强大肝细胞功能的体外模型仍然是生物技术的主要目标。gydF4y2Ba
新分离的原代人肝细胞(PHHs)是研究肝功能的金标准。然而,标准的二维PHH培养难以扩张并迅速丧失肝功能gydF4y2Ba2 gydF4y2Ba.为了克服这些限制,许多研究小组试图改进PHH的长期培养方法,刺激增殖或减少去分化gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba.此外,从其他细胞来源建立了肝体外模型,包括胎儿肝细胞、肝内胆管细胞、多能干细胞、成纤维细胞、尿细胞和间充质干细胞gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba.我们在这里将这些模型统称为肝细胞样细胞(HLCs)。gydF4y2Ba
肝表型可能在不同的HLCs之间有所不同,这取决于细胞的起源和培养方案。为了选择合适的HLC模型来研究特定的生物学或临床问题,需要澄清这些差异并确定性能最佳的模型。在这项研究中,我们开始比较每个HLC方案的最后阶段之间的肝细胞功能。对文献的系统搜索和对报道的不同HLCs的功能测定和单个基因表达的分析,不允许对研究之间的肝脏表型进行彻底的比较。因此,我们开发了一种计算算法,用于比较不同研究中的整个转录组测序(RNA-seq)数据,并开发了一个web应用程序,以便在未来添加额外的HLC数据集(HLCompR,gydF4y2Bahttps://github.com/iardisasmita/HLCompRgydF4y2Ba).这一资源将指导选择适合特定研究目标的HLC,并帮助改进HLC培养方案。gydF4y2Ba
结果gydF4y2Ba
报道的肝细胞功能不足以进行HLC比较gydF4y2Ba
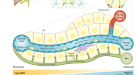
我们检索了文献中描述HLC培养方案和肝功能特征的文章。由于HLCs的交叉研究比较需要一个通用的标准,我们只考虑了同时测试HLCs和PHHs的研究。该策略产生了53项研究,描述来自多能干细胞(PSCs)、成纤维细胞、间充质干细胞、尿细胞、肝内胆管细胞和phh的HLCs(图2)。gydF4y2Ba1gydF4y2Ba和补充图。gydF4y2Ba1gydF4y2Ba).为了进行定量比较,我们考虑了在10多项研究中进行的肝功能测定、相关基因的表达以及常用的肝脏标记物基因(补充数据)gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba最常报道的肝功能和基因表达。在同一研究中,每个细胞中的值表示HLC的活性或表达水平占PHH对照的百分比。HLCs是根据产生它们的细胞类型进行分组的。多能干细胞(PSC)衍生的HLCs进一步分为:GF,使用生长因子的标准方案;SM,仅使用合成分子的协议;3D,利用3D矩阵的协议;联合崇拜,结合多种细胞类型的协议;LN,关注层粘连蛋白涂层效果的方案。间充质干细胞(MSC)来源的HLCs分为起源组织:AT脂肪组织,CB脐带血,BM骨髓。gydF4y2BabgydF4y2BaGao等人的研究显示,培养0、1、2和4天的PHHs和培养的真皮成纤维细胞中,与常见报道的肝功能相关基因的mRNA表达(log2)。gydF4y2Ba12gydF4y2Ba.注意某些基因的表达(如。gydF4y2BaCYP3A4gydF4y2Ba,gydF4y2BaCYP1A2gydF4y2Ba,gydF4y2Ba场外gydF4y2Ba)在培养后24小时内使>降低4倍,而其他基因(gydF4y2Ba铝青铜gydF4y2Ba,gydF4y2BaASS1gydF4y2Ba,gydF4y2BaSERPINA1gydF4y2Ba)保持相对稳定。源数据在补充数据中提供gydF4y2Ba3.gydF4y2Ba.gydF4y2Ba
大多数(>50%)研究评估了一些功能,包括白蛋白分泌和CYP3A4活性,而其他重要的肝功能,包括胆汁分泌、胆固醇代谢和糖异生通常没有得到解决(图2)。gydF4y2Ba1gydF4y2Ba及补充资料gydF4y2Ba1gydF4y2Ba).此外,许多研究没有将PHHs包括在所有功能测定中。同样,只有RNA的表达gydF4y2Ba铝青铜gydF4y2Ba,gydF4y2BaCYP3A4gydF4y2Ba,gydF4y2BaCYP1A2gydF4y2Ba,gydF4y2BaCYP2C9gydF4y2Ba在大多数研究中(>50%)报告,而其他重要的肝脏标记物缺乏。gydF4y2Ba
对于肝功能的交叉研究比较,我们考虑了HLCs的功能活性,计算为同一研究中PHH对照组的百分比(图2)。gydF4y2Ba1gydF4y2Ba).使用这种方法,不同的肝功能似乎相互独立地进化。例如,在Wang等人的psc来源的HLCs中,高白蛋白分泌与高CYP3A4活性相关。gydF4y2Ba46gydF4y2Ba,但在Boon等人的psc衍生的HLCs中不存在。gydF4y2Ba30.gydF4y2Ba.这表明,仅使用单一的肝功能是不可能预测整个肝细胞成熟的。gydF4y2Ba
重要的是,不同的研究没有指定PHH对照的培养时间。这可能导致对照PHH功能的显著变化,因为在PHH培养过程中,包括CYP3A4活性在内的特定肝功能迅速丧失gydF4y2Ba12gydF4y2Ba,gydF4y2Ba57gydF4y2Ba(无花果。gydF4y2Ba1 bgydF4y2Ba).因此,对照PHHs的培养时间和检测程序的微小变化可能对HLCs的肝功能相对活性产生深远影响。gydF4y2Ba
总之,PHH对照的频繁遗漏,功能测定的范围狭窄,PHH对照的可能变异性,使得不可能根据报道的测定比较HLCs之间的肝脏表型。gydF4y2Ba
转录组比较显示HLCs中明显的肝脏特异性分子特征gydF4y2Ba
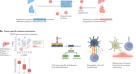
为了实现HLC转录组范围内的标准化比较,我们开发了一种计算算法来分析来自不同HLC研究的原始大量RNA-seq数据,其中包括phh或肝组织作为通用对照(图2)。gydF4y2Ba2gydF4y2Ba).这产生了11项描述来自肝细胞、肝内胆管细胞、成纤维细胞和PSCs的HLCs的研究。gydF4y2Ba2 bgydF4y2Ba及补充资料gydF4y2Ba2 gydF4y2Ba).此外,Huch等人开发的协议。gydF4y2Ba17gydF4y2BaHu等。gydF4y2Ba4 gydF4y2Ba通常在我们的实验室和其他地方分别用于生成肝内胆管细胞来源的类器官和(胎儿)肝细胞来源的类器官。由于这两项研究都没有提供大量RNA-seq数据,我们使用相应的方案生成了来自肝内胆管细胞(huch - hcl -HLCs)和胎儿肝细胞(Hu-FHep-HLCs)的HLCs RNA-seq数据gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba58gydF4y2Ba.我们没有包括成人肝细胞来源的类器官,因为我们不能长时间培养它们,据我们所知,其他组也不能gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba.用于生成不同hlc的协议在补充图中表示。gydF4y2Ba2 gydF4y2Ba.除了HLCs及其各自的PHH对照外,我们还包括胎儿肝细胞、PSCs、成纤维细胞、肝癌细胞系HepG2和常见胆管组织(CBD)gydF4y2Ba60gydF4y2Ba即肝外胆管细胞;补充数据gydF4y2Ba2 gydF4y2Ba).gydF4y2Ba
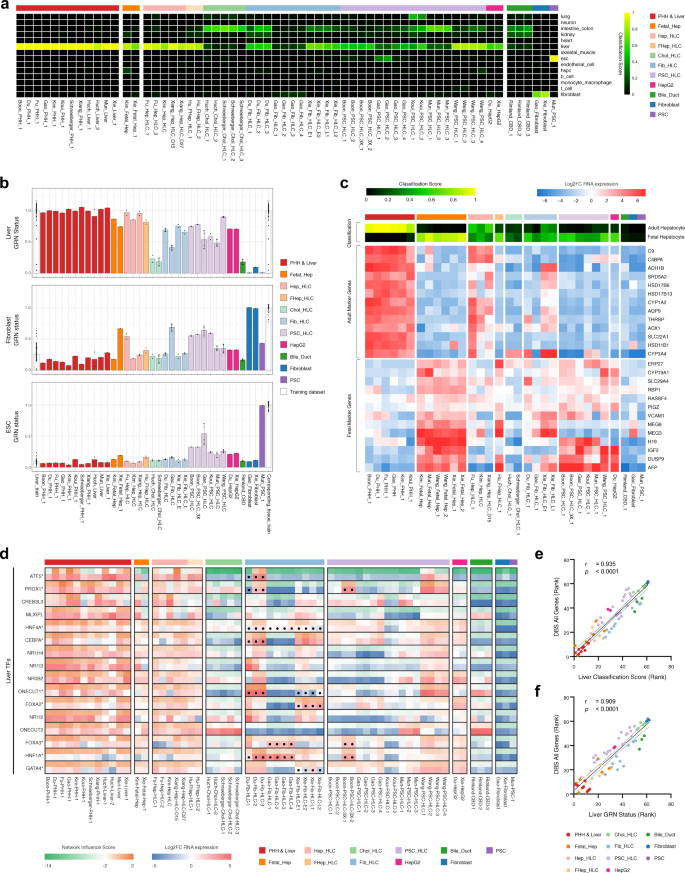
一个gydF4y2Ba用于进行转录组比较分析的算法示意图。所有灰色的步骤都是在银河网络平台上执行的。gydF4y2BabgydF4y2Ba转录组比较分析中用于生成HLCs的协议摘要。前体培养基是在最终成熟培养基之前直接使用的任何培养基,包括祖细胞/膨胀培养基(肝细胞、胆管细胞和成纤维细胞来源的HLCs)和肝细胞分化培养基(psc来源的HLCs)。gydF4y2BacgydF4y2Ba主成分分析使用5000个不同研究样本中方差最高的基因。gydF4y2BadgydF4y2Ba根据基因型-组织表达(GTEx)项目的最高表达肝脏基因热图。采用欧氏距离进行分层聚类。gydF4y2BaegydF4y2Ba基于距离的相似度评分(DBS)使用欧几里得距离计算,该距离描述了每个样本与基于最高表达的肝脏基因集的所有PHH/Liver样本之间的相似性。盒须图显示为中值(线)、四分位范围(框)和数据范围或1.5倍四分位范围(须)。gydF4y2BafgydF4y2Ba热图显示了使用各种肝功能相关基因集的所有样本的中位DBS。红色轮廓表示每个基因集的HLC具有最高的DBS。gydF4y2BaggydF4y2Ba基于中心周和门静脉周围模块,所有样本相对于PHH/Liver的中位DBS(图例见图2c)。源数据在补充数据中提供gydF4y2Ba4 gydF4y2Ba而且gydF4y2Ba5gydF4y2Ba.gydF4y2Ba
主成分分析(PCA)显示样本是根据细胞类型而不是根据研究聚类的,这证实了我们的计算方法允许交叉研究比较(图2)。gydF4y2Ba2摄氏度gydF4y2Ba).这也证实了来自不同研究的PHH/肝脏样本的转录同质性,它们可以共同作为肝脏的基准。大多数HLCs与各自的细胞来源紧密聚集,其中肝细胞来源的HLCs (Hep-HLCs)与phh和肝组织最紧密聚集(图2)。gydF4y2Ba2摄氏度gydF4y2Ba).来自Xie和Du的成纤维细胞来源的HLCs (Fib-HLCs)聚集在惊人地接近Hep-HLCs,而来自Gao的Fib-HLCs聚集在成纤维细胞附近。Du和Gao使用了相似的培养基组成,但不同的转录因子(tf)进行转分化(图2)。gydF4y2Ba2 bgydF4y2Ba),显示使用Du的TF组合更好的肝脏重编程。区分肝内胆管细胞与肝细胞(choll -HLCs)的方法导致HLCs与cbd聚集在一起。有趣的是,根据Huch等人的肝内胆管细胞培养方案,来自Mun的scs衍生的HLCs (pc -HLCs)经历了最后的成熟步骤。gydF4y2Ba17gydF4y2Ba,gydF4y2Ba24gydF4y2Ba(补充图。gydF4y2Ba3.gydF4y2Ba),也聚集在cbd附近,而不是PSCs或phh附近。这表明Huch等人的培养方案。gydF4y2Ba17gydF4y2Ba引导分化向胆管细胞而不是肝细胞。考虑到其他PSCs衍生的HLCs (pc - HLCs),我们观察到Wang的HLCs来自具有形成胚外组织能力的PSCsgydF4y2Ba61gydF4y2Ba, Hep-HLCs比来自标准PSCs的HLCs (Gao, Mun, Boon, Koui;无花果。gydF4y2Ba2摄氏度gydF4y2Ba).gydF4y2Ba
我们通过考虑基因型-组织表达(GTEx)数据库中肝脏中最高表达的基因,建立了一个一般的肝脏指纹。分层聚类显示,基于这些顶级表达的肝脏基因,只有Hep-HLCs与PHHs和肝组织聚在一起(图2)。gydF4y2Ba二维gydF4y2Ba).为了根据给定的基因集量化HLCs与PHHs的总体相似性,我们计算了基于距离的相似性评分(DBS)(图2)。gydF4y2Ba2 egydF4y2Ba).基于最高表达的肝脏基因,Hep-HLCs的DBS最高,其次是wang - pcs - hlcs, Fib-HLCs,其他pcs - hlcs,最后是choll - hlcs(图2)。gydF4y2Ba2 egydF4y2Ba).gydF4y2Ba
接下来,我们考虑了几个与特定肝功能相关的基因集。来自肝细胞的HLCs、来自Xie的成纤维细胞和来自Wang的扩展PSCs通常与PHHs最相似,但当考虑特定的基因集时,可以观察到明显的差异(图2)。gydF4y2Ba2 fgydF4y2Ba和补充图。gydF4y2Ba4 gydF4y2Ba而且gydF4y2Ba5gydF4y2Ba).例如,除糖异生基因组外,大多数基因组在Hep-HLCs中表达高于Fib-HLCs。在所有的Hep-HLC样本中,Xiang-Hep-HLC-D15通常与PHHs和肝组织最相似(图2)。gydF4y2Ba2 fgydF4y2Ba),这可能是由于与其他HLC(>3周)相比,该HLC样本的培养时间相对较短(15天)。gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.我们还观察到,在整体基因表达上,choll - hlcs与CBDs高度相似(图2)。gydF4y2Ba2摄氏度gydF4y2Ba),与胆固醇代谢和胆汁分泌相关的基因表达略有增加(图。gydF4y2Ba2 fgydF4y2Ba).在HepG2细胞中,包括胆固醇和糖原代谢和补体生成在内的特定基因组表达相对较好。gydF4y2Ba
然后,我们评估了与特定肝功能相关的基因集的表达是否反映了HLCs之间分区的差异。为此,我们从Aizarani及其同事对人肝细胞的单细胞分析中提取了门静脉周围和中心周围基因模块gydF4y2Ba62gydF4y2Ba.为了确保这些模块评估与肝细胞分区相关的肝特异性基因,我们只纳入PHH/肝脏样本中与其他HLC细胞来源(Fib, PSC, Chol)相比富集(>2倍)的分区基因。gydF4y2Ba6gydF4y2Ba).这种方法揭示了大多数HLCs以线性方式表达门静脉周围和中心周围模块(图。gydF4y2Ba2 ggydF4y2Ba).这可能表明,要么在大多数HLCs中没有强烈的分区模式(假设所有细胞中的基因表达谱都相同),要么由于批量测序分析的性质,存在无法识别的分区模式。单细胞RNA测序分析需要完全解决HLCs的分区。有趣的是,Chol-HLCs和HepG2细胞最不符合这种一般线性模式。Chol-HLCs显示主要的门静脉周围特征(图。gydF4y2Ba2 ggydF4y2Ba),对应于糖异生基因的相对高表达(图;gydF4y2Ba2 fgydF4y2Ba).相反,HepG2细胞表现出更多的中心周围特征(图2)。gydF4y2Ba2 ggydF4y2Ba),对应于胆固醇代谢相关基因的较高表达gydF4y2Ba63gydF4y2Ba(无花果。gydF4y2Ba2 fgydF4y2Ba).gydF4y2Ba
通过考虑所有可用研究中的HLCs,我们的方法还揭示了在个别研究中使用不同方案产生的HLCs之间肝脏差异的相对大小。例如,Gao研究中的HLCs要么直接从成纤维细胞转分化(Fib-HLCs),要么通过中间iPSC步骤(PSC-HLCs)产生。Gao等。gydF4y2Ba12gydF4y2Ba观察到他们的Fib-HLCs在I期和II期反应中表现更好,而他们的PSC-HLCs更好地模拟了肝脏脂肪酸代谢。我们的分析证实了这些发现(图。gydF4y2Ba2 fgydF4y2Ba和补充图。gydF4y2Ba4 egydF4y2Ba而且gydF4y2Ba5gydF4y2Ba),但也表明,与其他研究的HLCs相比,Gao研究中的Fib-HLCs和pc -HLCs都表现出相对较弱的肝脏表型。此外,Boon等人。gydF4y2Ba30.gydF4y2Ba试图通过TF转导来促进psc衍生的HLCs的成熟(gydF4y2BaHnf1a, foxa3, prox1gydF4y2Ba),并表明这种方法导致更高的白蛋白分泌和CYP3A4活性。然而,相对于其他HLCs, TF转导仅导致肝细胞分化的轻微改善(图。gydF4y2Ba2c f ggydF4y2Ba).此外,TF转导不仅增加了(例如,gydF4y2Ba铝青铜gydF4y2Ba而且gydF4y2BaHPX)gydF4y2Ba但也减少了(例如,gydF4y2BaAPOA2gydF4y2Ba而且gydF4y2Ba竞技场队伍gydF4y2Ba) Boon等人的PSC-HLCs中某些特定肝脏标记物的表达。gydF4y2Ba30.gydF4y2Ba(无花果。gydF4y2Ba二维gydF4y2Ba).gydF4y2Ba
HLCs显示不同的细胞/组织特性gydF4y2Ba
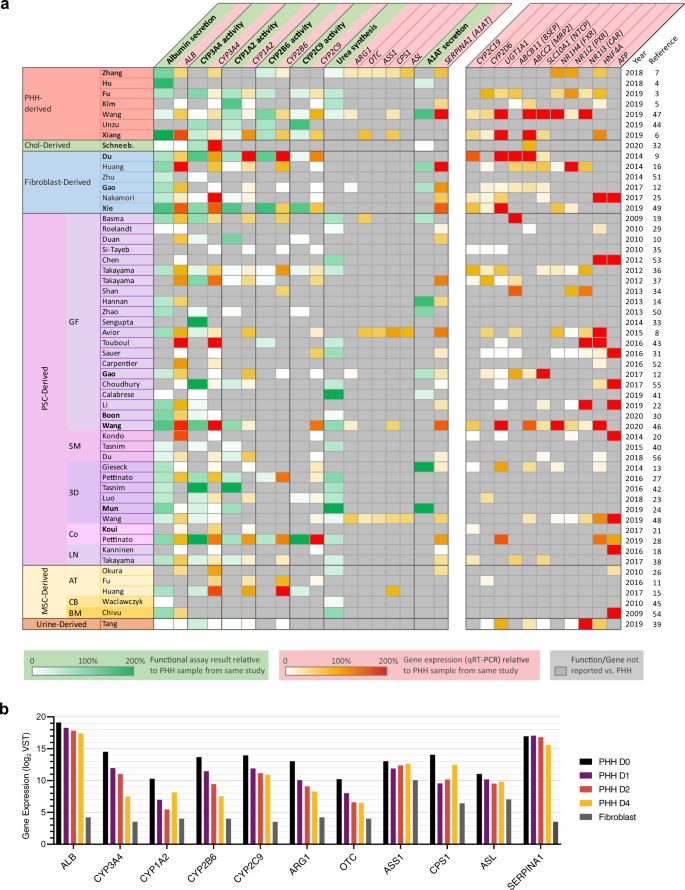
肝细胞体外模型的质量不应仅由肝细胞特性的存在来定义。gydF4y2Ba2 gydF4y2Ba),还因为不存在不想要的细胞/组织特征。因此,我们使用CellNet分析了HLCsgydF4y2Ba64gydF4y2Ba,gydF4y2Ba65gydF4y2Ba,该平台基于组织特异性基因调控网络(GRN状态)的建立,量化了与更多种人类细胞和组织类型(细胞/组织分类评分)的相似性。gydF4y2Ba
CellNet将大多数HLCs分类为肝脏,但只有(胎儿)肝细胞来源的HLCs获得了纯肝脏分类。来自PSCs、成纤维细胞和胆管细胞的HLCs要么被分类为多种细胞/组织类型,要么是非肝组织(图2)。gydF4y2Ba3gydF4y2Ba).这些HLCs不需要的细胞身份可归因于其原始细胞身份的不完全丧失或不需要的非肝脏身份的获得。gydF4y2Ba
一个gydF4y2Ba细胞/组织分类热图显示不同HLCs的不同细胞/组织特性。分类分数表示样本从训练数据集中表达不可区分基因调控网络(GRN)的概率。gydF4y2BabgydF4y2BaHLCs和CellNet训练数据集的肝脏、成纤维细胞和胚胎干细胞(ESC) GRN状态以平均值表示。每个点和误差条分别代表一个单独的样本和标准偏差。gydF4y2BacgydF4y2Ba热图显示成人/胎儿肝细胞分类评分和成人和胎儿肝细胞身份的代表性标记基因的表达。gydF4y2BadgydF4y2Ba热图显示网络影响评分(NIS)和肝脏相关转录因子(tf)的表达值。星号和圆点表示用于转导的tf。gydF4y2BaegydF4y2BaCellNet肝细胞/组织分类评分与Spearman分级之间的相关性gydF4y2BafgydF4y2BaCellNet肝脏GRN状态及所有基因的DBS排序。级别越低代表肝脏分级、GRN状态和DBS越高。斯皮尔曼相关系数(gydF4y2BargydF4y2Ba),gydF4y2BaPgydF4y2Ba值(gydF4y2BapgydF4y2Ba)显示在图表的左上方(gydF4y2BangydF4y2Ba= 62个生物独立样本)。虚线表示95%置信区间。源数据在补充数据中提供gydF4y2Ba6gydF4y2Ba.gydF4y2Ba
在所有的PSC-HLCs中都可以观察到原始细胞身份的不完全丧失,与PHHs相比,它表现出更高的胚胎/多能干细胞(ESC) GRN状态(图2)。gydF4y2Ba3 bgydF4y2Ba和补充图。gydF4y2Ba7gydF4y2Ba).有趣的是,尽管保持了可识别的ESC GRN状态,Wang的PSC HLCs显示出与Hep-HLCs相似的肝脏GRN状态(图2)。gydF4y2Ba3 bgydF4y2Ba).对于Fib-HLCs,只有来自Gao的样品未能完全消除其成纤维细胞GRN状态(图2)。gydF4y2Ba3 bgydF4y2Ba和补充图。gydF4y2Ba7gydF4y2Ba).这种纤维母细胞GRN的不完全丢失和肝脏GRN的部分获得导致所有特定细胞/组织类型的低分类评分(图2)。gydF4y2Ba3gydF4y2Ba).来自同一细胞源的HLCs (wang - pc -HLCs vs.其他pc -HLCs和high -Fib-HLCs vs.其他Fib-HLCs)之间细胞身份的异质性反映了每项研究中应用的不同培养方案的影响。gydF4y2Ba
所有非肝细胞来源的HLCs都表现出不希望的肠/结肠身份的增加(图。gydF4y2Ba3gydF4y2Ba).此外,来自Koui的pc - hlcs也获得了肺身份,这可能是由于它们的方案涉及向多谱系分化(图2)。gydF4y2Ba3gydF4y2Ba和补充图。gydF4y2Ba2 gydF4y2Ba).这种非肝脏身份的获得可能发生在所有细胞中,也可能只发生在一个细胞亚群中,这是由异质分化引起的。无论如何,这表明所有的HLCs生成方案仍然不完善,微调这些HLCs的细胞命运规范可能改善肝(反)分化gydF4y2Ba66gydF4y2Ba.令人惊讶的是,来自uch和Schneeberger的Chol-HLCs和来自Mun的PSC-HLCs与肠/结肠的相似性高于与肝脏的相似性(图2)。gydF4y2Ba3gydF4y2Ba).此外,我们的CBD对照样品的肠/结肠分类低于Huch方案培养的HLCs(图2)。gydF4y2Ba3gydF4y2Ba).然而,pca分析显示,Huch方案培养的HLCs与CBD更相似,而不是肠/结肠样本(补充图。gydF4y2Ba3 bgydF4y2Ba),表明uch方案不仅促进了CBD的表达,还促进了肠道基因的表达。这与Aizarani等人发现肝内胆管细胞在Huch膨胀培养基中作为类器官培养时上调肠道标记基因一致gydF4y2Ba62gydF4y2Ba.gydF4y2Ba
除了不需要的成人细胞/组织特性外,HLCs还可能显示不成熟的肝脏特性,类似于胎儿肝细胞gydF4y2Ba67gydF4y2Ba.由于CellNet被训练为只区分成人组织身份,我们创建了一个基于成人和胎儿肝细胞转录组的分类算法。与CellNet的结果相对应,Fu和Xiang的Hep-HLCs与成人肝细胞的转录组最相似(图2)。gydF4y2Ba3 a, cgydF4y2Ba).有趣的是,由于缺乏成人标记物或胎儿标记物,没有一个HLCs显示完全成熟的肝细胞指纹。gydF4y2Ba3 cgydF4y2Ba).Chol-HLCs未被分类为成人或胎儿肝细胞(图2)。gydF4y2Ba3 cgydF4y2Ba),表明它们的肝脏分化较弱。在所有成纤维细胞和psc来源的HLCs中,只有Xie的Fib-HLCs获得了强烈的成人特征,表达成熟的肝细胞标记,包括gydF4y2BaADH1BgydF4y2Ba而且gydF4y2BaSRD5A2gydF4y2Ba.尽管在CellNet分析中有说服力的肝脏GRN状态,来自Wang的psc来源的HLCs仍然表现出胎儿身份(图2)。gydF4y2Ba3 b, cgydF4y2Ba).gydF4y2Ba
对于使用TF转导的HLC方案,肝(转)分化的程度部分取决于所使用的TF组合的有效性。这种有效性可以使用CellNet分析中的网络影响评分进行评估。网络影响评分表明TF转导在高(gydF4y2BaHnf4a, foxa3, hnf1agydF4y2Ba)及布恩的pc - hlcs (gydF4y2BaProx1, foxa3, hnf1agydF4y2Ba)增加了TF的表达和相关TF网络的激活,而没有导致其他肝脏TF网络的激活(图。gydF4y2Ba3 dgydF4y2Ba).相比之下,Du (gydF4y2BaAtf5, prox1, cebpa, onecut1gydF4y2Ba(亦称gydF4y2BaHNF6gydF4y2Ba)和谢(gydF4y2BaOnecut1, foxa2, gata4gydF4y2Ba)产生了其他肝脏TF网络的广泛激活。值得注意的是,低RNA表达和低网络影响评分表明无效转导gydF4y2BaATF5gydF4y2Ba而且gydF4y2BaPROX1gydF4y2Ba其中一个Du样品(Du- fib - hlc -1),但有效转导gydF4y2BaONECUT1gydF4y2Ba,这仍然导致广泛的肝TF网络激活。综上所述,这表明gydF4y2BaONECUT1gydF4y2Ba是肝脏转分化鸡尾酒的重要因素。gydF4y2Ba
此外,我们使用CellNet来验证我们的计算算法,并发现我们分析中包括的PHH/肝脏、成纤维细胞和PSC样本与其相应的CellNet训练数据集具有很高的相似性(图2)。gydF4y2Ba3 a, bgydF4y2Ba).此外,CellNet肝脏分级评分和肝脏GRN状态与通过我们自己的方法获得的DBS评分(使用所有基因)具有良好的相关性(图。gydF4y2Ba3 e, fgydF4y2Ba).这证实了我们的转录组分析准确地量化了肝脏相似性。gydF4y2Ba
转录组比较可以预测HLC功能gydF4y2Ba
我们的转录组比较表明,在纳入的HLCs之间,肝脏相关基因的表达有很大的变异性。gydF4y2Ba2 fgydF4y2Ba).为了确定我们的比较是否可以预测特定的HLCs模拟肝功能的能力,我们评估了转录组差异是否转化为蛋白质组差异并最终转化为功能差异。gydF4y2Ba
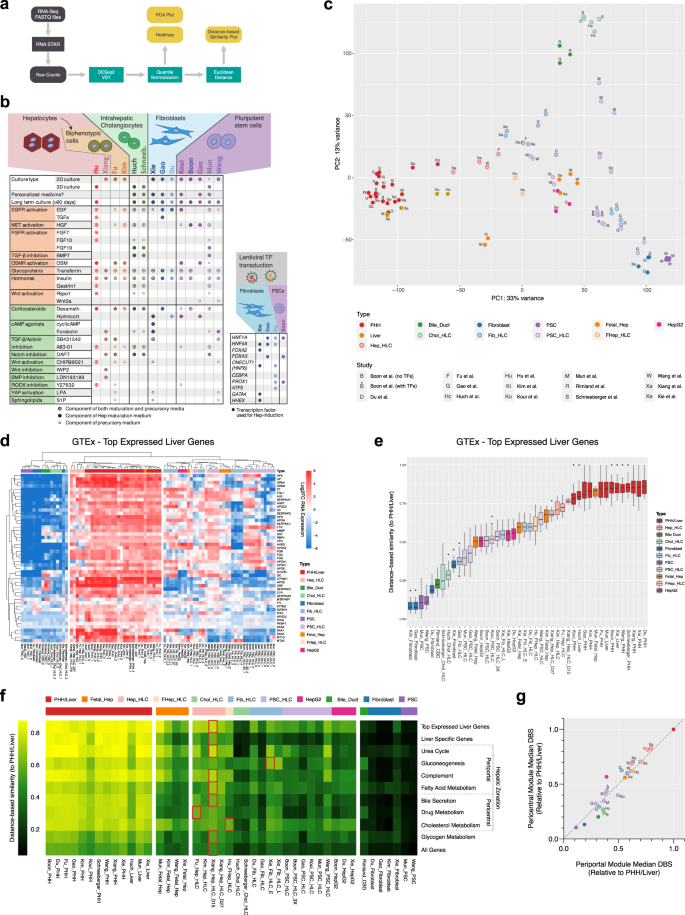
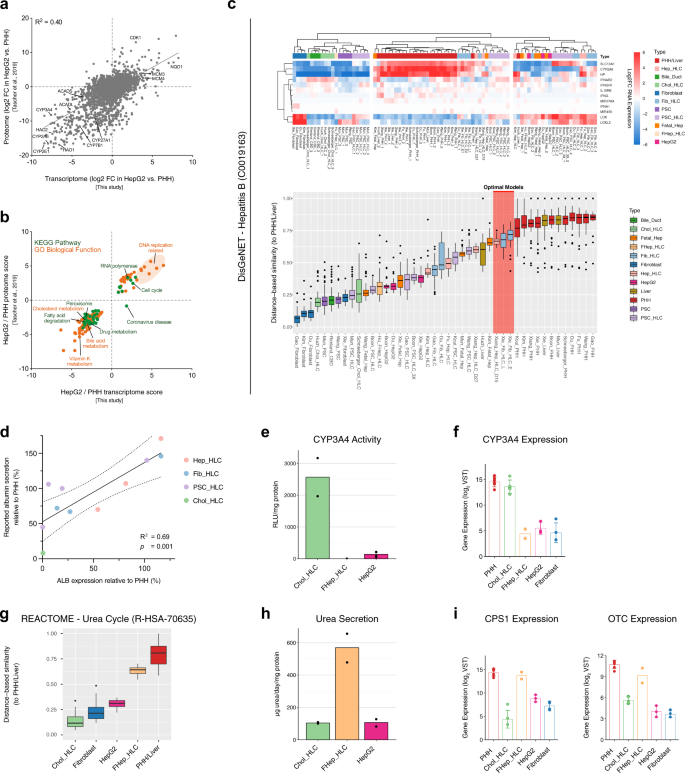
我们首先考虑了公开的HepG2细胞和phh的蛋白质组gydF4y2Ba68gydF4y2Ba发现HepG2细胞的转录组和我们数据集中的phh相关性非常好(gydF4y2BaRgydF4y2Ba2 gydF4y2Ba= 0.40,gydF4y2BapgydF4y2Ba< 0.0001;无花果。gydF4y2Ba4gydF4y2Ba)到这项独立研究的蛋白质组gydF4y2Ba68gydF4y2Ba.事实上,这种相关性与研究中的转录组-蛋白质组相关性相当gydF4y2Ba69gydF4y2Ba(gydF4y2BaRgydF4y2Ba2 gydF4y2Ba= 0.41)。转录组学和蛋白质组学富集分析同时表明,与PHHs相比,HepG2细胞表现出更高的细胞周期途径表达和较低的肝脏相关途径表达(图2)。gydF4y2Ba4 bgydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba对于在转录组(来自我们的转录组比较)和蛋白质组(来自Tascher等人)中检测到的蛋白质/基因,在HepG2细胞与PHHs中蛋白质丰度比率相对于相应mRNA比率的散点图。gydF4y2Ba68gydF4y2Ba).皮尔逊决定系数(gydF4y2BaRgydF4y2Ba2 gydF4y2Ba)显示在图的左上方。gydF4y2BabgydF4y2Ba基于转录组和蛋白质组的富集通路散点图。只有在转录组和蛋白质组水平上都富集的通路被显示出来。gydF4y2BacgydF4y2Ba根据DisGeNET数据库进行乙型肝炎相关基因的热图和DBS。红色高亮表示预测的最佳肝细胞体外模型。gydF4y2BadgydF4y2Ba转录组比较数据中ALB基因表达水平的相关图(gydF4y2BaxgydF4y2Ba-轴)和报告的相关白蛋白分泌(y轴)。皮尔逊决定系数(gydF4y2BaRgydF4y2Ba2 gydF4y2Ba),gydF4y2BaPgydF4y2Ba值(gydF4y2BapgydF4y2Ba)显示在图表的右下方(gydF4y2BangydF4y2Ba= 11个生物独立样本)。虚线表示95%置信区间。gydF4y2BaegydF4y2BaCYP3A4活性表现为均值。gydF4y2BafgydF4y2Ba基因表达水平gydF4y2BaCYP3A4gydF4y2Ba转录组比较数据以均数±标准差表示。gydF4y2BaggydF4y2Ba根据Reactome数据库对尿素循环基因进行DBS。介绍了来自不同研究的Chol-HLC、成纤维细胞、HepG2细胞、FHep-HLC和PHH/肝脏样本的组合值。gydF4y2BahgydF4y2Ba尿素分泌水平为平均值。gydF4y2Ba我gydF4y2Ba基因表达水平gydF4y2BaCPS1gydF4y2Ba而且gydF4y2Ba场外gydF4y2Ba转录组比较数据以均数±标准差表示。盒须图显示为中值(线)、四分位范围(框)和数据范围或1.5倍四分位范围(须)。源数据在补充数据中提供gydF4y2Ba7gydF4y2Ba.gydF4y2Ba
接下来,我们通过考虑乙型肝炎病毒(HBV)感染来评估转录差异是否转化为功能差异。这种疾病已经成功地使用来自Xiang的Hep-HLCs和来自Xie的Fib-HLC进行了建模gydF4y2Ba6gydF4y2Ba,gydF4y2Ba49gydF4y2Ba.因此,HBV感染和传播相关基因的表达被这两种HLCs最好地概括了(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).这证实了对于HBV,我们的转录组比较可以预测体外建模的适用性。gydF4y2Ba
通过考虑报道的白蛋白分泌,我们进一步验证了我们的转录组比较的预测能力。gydF4y2Ba1gydF4y2Ba的基因表达gydF4y2Ba铝青铜gydF4y2Ba在培养的PHHs中保持稳定数天(图;gydF4y2Ba1 bgydF4y2Ba).因此,不同hcs的相对白蛋白分泌不受PHH对照可变性的影响,因此可以在各个研究中进行比较。的表达式gydF4y2Ba铝青铜gydF4y2Ba在我们的转录组比较中是一个很好的预测因子(gydF4y2BaRgydF4y2Ba2 gydF4y2Ba= 0.69;gydF4y2BapgydF4y2Ba= 0.001)的报告白蛋白分泌的11个纳入的HLCs(图。gydF4y2Ba4 dgydF4y2Ba).gydF4y2Ba
最后,我们验证了我们的转录组比较也可以预测受内部PHH控制的新鲜度高度影响的功能的活性,如CYP3A4和尿素循环活性(图3)。gydF4y2Ba1 bgydF4y2Ba).为了确保可比性,我们在统一的设置中使用我们组中已建立的HLCs (Chol-HLCs, FHep-HLCs和HepG2细胞)对这些功能进行了分析。我们发现CYP3A4的酶活性在Chol-HLCs中最高(图2)。gydF4y2Ba4 egydF4y2Ba),通过转录组比较可以正确预测(图。gydF4y2Ba4 fgydF4y2Ba).从尿素循环基因表达来看,FHep-HLCs与PHH/Liver最相似,其次为HepG2细胞和choll - hlcs(图2)。gydF4y2Ba4 ggydF4y2Ba).与此一致,FHep-HLCs在这些HLCs中表现出最高的尿素分泌(图2)。gydF4y2Ba4 hgydF4y2Ba).hl - hlcs与HepG2细胞之间的表达差异较小(图2)。gydF4y2Ba4 ggydF4y2Ba)并没有导致尿素分泌水平的差异(图。gydF4y2Ba4 hgydF4y2Ba),可能是因为尿素循环的两种必需酶(gydF4y2BaCPS1gydF4y2Ba而且gydF4y2Ba场外gydF4y2Ba)在Chol-HLCs和HepG2细胞中均低表达(图2)。gydF4y2Ba4我gydF4y2Ba).事实上,两种HLCs的表达水平都与成纤维细胞相似。gydF4y2Ba4 g,我gydF4y2Ba),它们也很难将氨转化为尿素gydF4y2Ba70gydF4y2Ba.总之,这些发现支持了我们的交叉研究转录组比较预测了HLC蛋白的丰度和功能。gydF4y2Ba
HLCompR web应用程序的HLC选择gydF4y2Ba
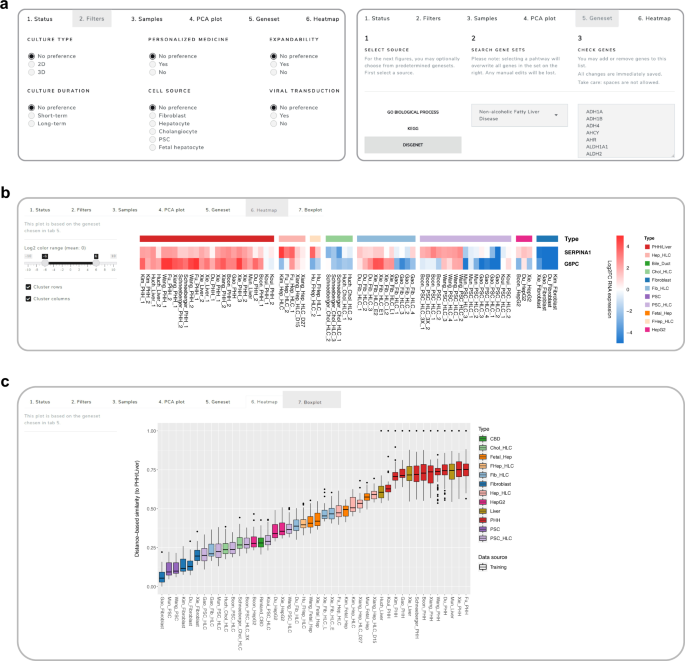
为了让其他研究人员检查一个重要基因或整个基因集的表达,以选择合适的hplc模型,我们创建了HLCompR,这是一个web应用程序,可以轻松探索任何感兴趣的基因(集)的相对表达(gydF4y2Bahttps://github.com/iardisasmita/HLCompRgydF4y2Ba).此外,HLCompR允许研究人员根据特定的特征(包括培养时间、可扩展性和细胞来源)筛选HLCs,并轻松选择功能或疾病相关的基因集(图2)。gydF4y2Ba5gydF4y2Ba).gydF4y2Ba
为了说明HLCompR如何帮助为特定的研究目的选择最佳的HLCs,我们考虑了与单基因缺陷相关的肝脏疾病(图2)。gydF4y2Ba5 bgydF4y2Ba).这表明多种HLCs表达gydF4y2BaSERPINA1gydF4y2Ba与PHHs水平相似的基因,因此可能适用于研究α -1抗胰蛋白酶缺乏症的蛋白质分泌和活性。相比之下,只有少数HLCs可以最佳地模拟糖原储存病1a型中受影响的酶的功能(gydF4y2BaG6PCgydF4y2Ba;无花果。gydF4y2Ba5 bgydF4y2Ba).对这种疾病建模的最佳选择是Xie的Fib-HLCs。gydF4y2Ba
对于与多个基因相关的更复杂的肝脏疾病,疾病相关基因集的基于距离的相似性评分可以更好地概述各种HLCs的建模能力。例如,与通常用于模拟NAFLD的HepG2细胞相比,Hep-HLCs更类似于非酒精性脂肪性肝病(NAFLD)相关基因的肝脏表达gydF4y2Ba71gydF4y2Ba(无花果。gydF4y2Ba5度gydF4y2Ba).除了与PHH的转录相似性外,NAFLD建模还需要长期培养完全成熟的HLCs。因此,来自Xiang的Hep-HLCs能够维持分化状态1个月,可能是本病的最佳体外模型。gydF4y2Ba
在HLCompR web应用程序中添加新的HLC转录组gydF4y2Ba
考虑到HLC协议固有的技术挑战和可变性,这种协议的复制可能不会产生表型相同的HLC。此外,新的HLC协议也在不断开发。因此,我们支持将新的RNA-seq数据添加到HLCompR分析中,使用根据我们的管道处理的读取计数作为输入(gydF4y2Bahttps://github.com/iardisasmita/HLCompRgydF4y2Ba).gydF4y2Ba
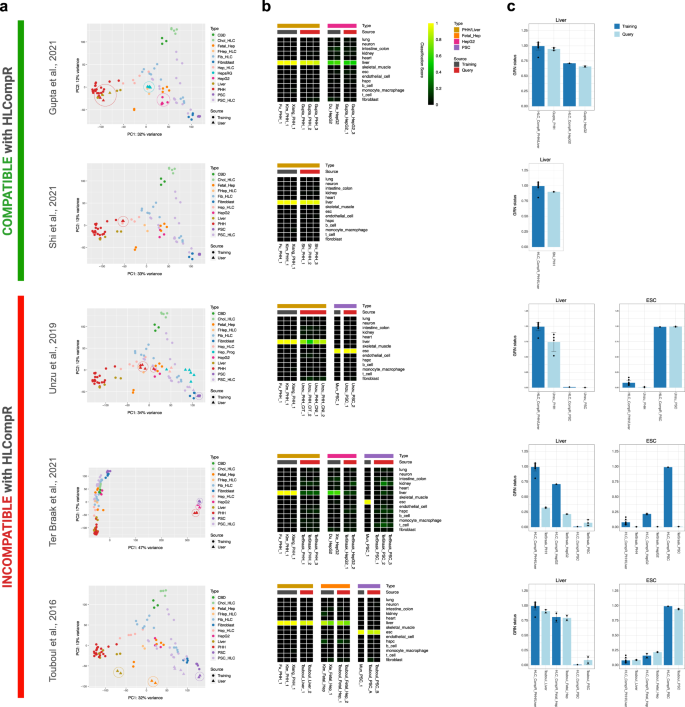
为了确定是否可以使用HLCompR比较新数据集,我们在应用程序中测试了公开可用的PHH或肝组织样本RNA-seq数据集gydF4y2Ba48gydF4y2Ba,gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba,gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba.我们还纳入了几项不符合RNA-seq纳入标准的研究gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba(查询数据集)gydF4y2Ba1gydF4y2Ba).图中所示的样品。gydF4y2Ba2 gydF4y2Ba作为HLCompR的训练数据集。gydF4y2Ba
来自Gupta等人的查询数据集。gydF4y2Ba74gydF4y2Ba与HLCompR兼容,因为他们的PHH和肝脏样本与训练样本聚集在一起(图2)。gydF4y2Ba6gydF4y2Ba).因此,这些样本在CellNet中获得了较高的肝脏分类和肝脏GRN状态评分(图2)。gydF4y2Ba6 b, cgydF4y2Ba).查询Shi等人的数据集。gydF4y2Ba75gydF4y2Ba和Vieyres等人。gydF4y2Ba76gydF4y2Ba也与HLCompR兼容,即使它们的PHH聚集在训练的PHH/肝脏样本和肝细胞来源的HLCs之间(图2)。gydF4y2Ba6gydF4y2Ba和补充图。gydF4y2Ba8gydF4y2Ba).CellNet将他们的PHH样本归类为肝脏,但他们的肝脏GRN状态略低于训练的PHH/肝脏样本(图2)。gydF4y2Ba6 b, cgydF4y2Ba和补充图。gydF4y2Ba8 b, cgydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba查询数据集的主成分分析。圆圈表示查询数据集中也出现在训练数据集中的细胞或组织类型(例如,PHH,肝脏,HepG2,胎儿肝细胞和PSC)或Gupta中的HepaRG样本gydF4y2Ba74gydF4y2Ba数据集。HLCompR兼容性是根据查询数据集和训练数据集之间phh和肝组织的可比性进行分类的。gydF4y2BabgydF4y2Ba代表性训练样本和查询样本的细胞/组织分类热图。gydF4y2BacgydF4y2Ba训练数据集和查询数据集中所有PHH、肝脏、HepG2和PSC样本的肝脏和胚胎干细胞(ESCs)基因调控网络状态以均数±标准差表示。源数据在补充数据中提供gydF4y2Ba8gydF4y2Ba.gydF4y2Ba
Unzu等人的查询数据集。gydF4y2Ba44gydF4y2Ba与HLCompR不兼容,因为来自他们数据集的phh没有与训练数据聚类,但PSCs可以(图2)。gydF4y2Ba6gydF4y2Ba).同时,CellNet分析显示phh分类不正确,PSCs分类正确(图。gydF4y2Ba6 b, cgydF4y2Ba).类似地,在ter Braak等人的查询数据集中包含phh。gydF4y2Ba72gydF4y2Ba, Wang等。gydF4y2Ba48gydF4y2Ba, Guan等。gydF4y2Ba73gydF4y2Ba没有与训练数据集聚类(图;gydF4y2Ba6gydF4y2Ba和补充图。gydF4y2Ba8gydF4y2Ba),这也反映在低CellNet肝脏评分(图。gydF4y2Ba6 b, cgydF4y2Ba和补充图。gydF4y2Ba8 b, cgydF4y2Ba).样本来自Touboul等人。gydF4y2Ba43gydF4y2Ba在主成分1 (PC1)上聚集在各自的训练样本附近,但被主成分2 (PC2)分开(图2)。gydF4y2Ba6gydF4y2Ba).尽管如此,CellNet分析显示分类分数与训练样本相似(图2)。gydF4y2Ba6 b, cgydF4y2Ba).有趣的是,当改变考虑用于PCA的基因数量或减少包含的样本数量时(补充图。gydF4y2Ba8 dgydF4y2Ba), Touboul等人的样本。gydF4y2Ba43gydF4y2Ba与训练样本聚在一起。然而,我们考虑了Touboul等人的样本。gydF4y2Ba43gydF4y2Ba与HLCompR不兼容。我们建议仅当对照PHH/肝脏样本与训练PHH/肝脏样本相当时才使用HLCompR。我们添加了一个随机森林分类器,它根据这个参数自动报告新数据集的兼容性。gydF4y2Ba
由于标准化的映射和RNA-seq处理仍然可能导致与HLCompR不兼容,我们假设RNA-seq文库制备或测序仪的类型可能影响兼容性(补充数据)gydF4y2Ba2 gydF4y2Ba).基于这种可能性,我们建议使用标准的Illumina TruSeq RNA样品制备试剂盒制备文库,并使用Illumina测序仪进行测序。由于我们不能保证兼容性,新的数据集应始终包括PHH/肝脏对照样本。gydF4y2Ba
当数据集与HLCompR兼容时,新的HLC样本可以与训练数据集中的其他HLC进行比较。例如,在Gupta等人的查询数据集中。gydF4y2Ba74gydF4y2Ba,与HepG2细胞相比,肝分化肝癌细胞系HepaRG与PHHs的相似性更好(图2)。gydF4y2Ba6gydF4y2Ba).相应地,也有报道HepaRG细胞在肝功能检测中比HepG2细胞表现更好gydF4y2Ba77gydF4y2Ba.gydF4y2Ba
讨论gydF4y2Ba
大量肝细胞模型系统的可用性使研究人员可以选择最适合他们的研究,但由于缺乏标准化评估,这种明智的选择受到了阻碍。我们通过对来自不同细胞来源的HLCs进行全面的交叉研究比较来解决这一问题。我们的文献检索显示,只有一小部分肝功能常规检测与PHH对照直接比较,排除了HLCs的交叉研究比较。此外,随着培养的PHHs迅速失去特定的肝功能,这些功能在HLCs中相对活性的一个主要决定因素是对照PHHs的培养时间。由于各种HLC研究使用培养不同时间的phh对照,大多数报道的肝功能的相对活动无法在研究之间进行可靠的比较。因此,我们不仅建议描述新hcs的方案报告一组最小的肝脏特征gydF4y2Ba78gydF4y2Ba,但我们也强调了使用标准化检测方法和纳入在标准化时间内培养的对照PHHs的重要性。gydF4y2Ba
利用目前可用的RNA-seq数据,我们开发了一种计算算法,用于HLCs的深入交叉研究比较,具有最小的研究特异性批效应。这使得在个别研究中很少调查的基因得以评估,但对特定的肝功能或肝病模型可能很重要。我们的分析显示,HLCs的转录组谱由细胞来源和使用的方案决定,肝细胞来源的HLCs最接近PHHs。此外,我们还发现了在大多数HLCs中低表达的肝脏标记基因,包括gydF4y2BaCYP2E1gydF4y2Ba,gydF4y2BaADH1AgydF4y2Ba,gydF4y2BaF9gydF4y2Ba,gydF4y2BaSERPINC1gydF4y2Ba.这些基因可能是肝分化成熟的重要指标,除了常见的标记,如gydF4y2Ba铝青铜gydF4y2Ba,gydF4y2BaCYP3A4gydF4y2Ba,gydF4y2BaSERPINA1gydF4y2Ba.gydF4y2Ba
我们发现肝脏基因在不同的HLCs之间有不同的表达。这强调了为特定的研究问题选择最合适的高效液相色谱的重要性。我们验证了我们的转录组HLC比较可以在我们实验室建立的模型中预测功能性能。转录和功能谱之间的强相关性支持我们将转录组比较作为一种资源来选择用于特定研究目标的HLCs。选择最优的HLC是由我们的web应用程序(HLCompR)提供的,它允许通过HLC属性进行过滤并选择自定义基因集。这一功能将HLCompR与其他比较平台(如CellNet)区别开来,后者专注于评估HLCs的一般细胞身份。HLCompR web应用程序还允许研究人员测试在他们自己的实验室中生成的HLCs,包括本研究中包括的HLC协议的复制或新的HLC协议的开发,如果PHH控制与训练数据集相当。因此,我们建议描述新hcs的方案提供公开的转录组数据以及PHH对照,作为交叉研究可比性的基准。gydF4y2Ba
除了与PHHs的转录和功能相似性外,在选择最佳肝脏体外模型时,其他特征可能也很重要,包括从小型活检中获得HLCs用于个性化医疗的能力。这可以通过使用直接转分化(Xie, Gao, Du)或重新编程为诱导pscs (iPSCs),随后分化为HLCs (Koui, Gao, Mun)来实现。使用tf的特定组合,成纤维细胞的直接转分化导致HLCs在转录上比大多数psc衍生的HLCs更接近PHHs。我们的方法指定了这些tf的效果。例如,Xie使用的tf组合不仅诱导了被转导的tf调节的基因网络的激活,而且还诱导了其他网络的激活(图2)。gydF4y2Ba3 dgydF4y2Ba).添加gydF4y2BaATF5gydF4y2Ba而且gydF4y2BaPROX1gydF4y2Ba,在Xie的Fib-HLCs中建立相对较差,可能进一步改善肝脏分化。gydF4y2Ba
肝细胞成熟度不能仅由转录组决定,还应由蛋白质组决定,并最终由功能决定。然而,HLCs之间肝功能的可靠比较需要完全标准化的方法和PHH对照。在实践中,这需要使用相同的实验装置同时在所有HLCs中进行分析。考虑到重现所有hcs的困难,这将是非常具有挑战性的。因此,转录组比较可能是确保来自不同研究的多个样本标准化的最引人注目的解决方案。事实上,这允许使用多个研究中PHH对照的共同基准来评估HLCs,防止在个别研究中由于次优PHH对照而导致的肝脏特征的高估或低估。此外,转录组比较能够基于少数报道的标记基因进行全面的描述。这包括评估多个细胞/组织的特性和肝细胞成熟的程度(图。gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
总之,这项研究提供了一种方法和一个web应用程序(HLCompR)来比较和评估来自多项研究的HLCs。我们发现肝细胞来源的HLCs的转录组目前与原代肝细胞最相似。然而,出于个性化医疗的目的,皮肤成纤维细胞更容易获得,成纤维细胞直接转分化的改进导致了优秀的HLCs。重要的是,我们的策略允许识别个体tf或可能改善肝脏分化的培养条件。此外,与相关对照组织一起评估HLCs可深入了解其组织特性。这些见解将指导HLC培养方案的改进,从而推进肝脏体外建模和支持再生策略。最后,虽然我们关注的是肝细胞的体外模型,但同样的方法也可以应用于评估其他细胞类型或器官的体外模型。gydF4y2Ba
方法gydF4y2Ba
数据收集gydF4y2Ba
系统检索PubMed数据库中提及开发或评估hplc培养方案的研究。选择的检索词包括进行人原代肝细胞长期培养或将人体细胞或干细胞(反式)分化为HLCs的研究,包括功能或转录组评估(补充图)。gydF4y2Ba1gydF4y2Ba).筛选了所有热门文献的标题和/或摘要,包括描述和评价新HLC方案的研究,并排除了使用先前描述的HLC方案的研究。在随后的全文筛选中,仅选择以PHH作为共同标准对新的HLC方案进行功能评估的研究进行功能和表达比较。gydF4y2Ba1gydF4y2Ba.最后,从选定的研究中,只有那些提供公开可用的大量RNA-seq数据集并包含PHH/肝脏对照的研究被纳入我们的计算算法进行分析(补充图。gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
使用已报道的数据比较HLCs的功能和表达gydF4y2Ba
根据原始研究中提供的图形和图表,估计qPCR定量功能分析和mRNA表达。将HLCs的功能和表达数据归一化为同一研究中相应的PHH数据,并以百分比表示。使用GraphPad Prism 8计算和可视化分析分析和/或基因表达之间的Pearson相关性。gydF4y2Ba
研究批准和人类受试者gydF4y2Ba
该研究得到了当地负责的伦理委员会的批准(乌得勒支大学医学中心机构审查委员会(STEM: 10-402/K;TcBio 14 - 008;代谢生物库:19-489),伊拉斯谟MC医学伦理委员会(MEC-2014-060)和荷兰伦理医学委员会(莱顿大学MC))。在鹿特丹Erasmus MC的手术中,从健康捐赠者的肝脏中获得组织活检。人类胎儿肝脏来自莱顿大学医学中心(MC)。所有患者材料均经书面知情同意后使用。gydF4y2Ba
类器官的建立和培养gydF4y2Ba
如前所述,建立和培养胆管细胞来源的类器官gydF4y2Ba17gydF4y2Ba.为了获得细胞,将肝活检切片切成小块,用10 mg/ml胶原酶D (Sigma, 11088866001)在37℃下消化20分钟。然后用Advanced DMEM/F12 (Gibco, 12634028)加2 mM GlutaMAX (Gibco, 35050061)、10 mM HEPES (Gibco, 15630080)、100 U/ml PenStrep (Gibco, 15140122)的冷水洗涤样品,以1500 rpm转速旋转5分钟。细胞球镀于基质(Corning, 356231)中,加入培养基。培养基为Advanced DMEM/F12,添加2 mM GlutaMAX, 10 mM HEPES, 100 U/ml PenStrep, 2% B27不含维生素A (Gibco, 12587010), 10 mM烟酰胺(Sigma, N0636), 1.25 mMgydF4y2BaNgydF4y2Ba-乙酰半胱氨酸(Sigma, A9165), 10% RSPO1条件培养基(自制),10 nM Gastrin (Tocris, 3006/1), 50 ng/ml EGF (Peprotech, AF-100-15), 100 ng/ml FGF10 (Peprotech, 100-26), 25 ng/ml HGF (Peprotech, 100-39), 50 μg/ml Primocin (Invivogen, ant-pm-2), 5 μM A83-01 (Tocris, 2939/10), 10 μM Forskolin (Tocris, 1099/10)。活检分离后的前3天,培养基中添加30% Wnt条件培养基(自制)、25 ng/ml Noggin (Peprotech, 120-10C)和hES细胞克隆恢复液(Stemgent, 010014500)。每3-4天更换一次培养基,每周以1:4-1:8的比例传递类器官。在含有25 ng/ml BMP7 (Peprotech, 120-03)的培养基中培养5-7天,开始向肝细胞分化。培养基改为添加2 mM GlutaMAX、10 mM HEPES、100 U/ml PenStrep、2% B27不含维生素A、1.25 mM的Advanced DMEM/F12gydF4y2BaNgydF4y2Ba-乙酰半胱氨酸、10 nM Gastrin、50 ng/ml EGF、25 ng/ml HGF、100 ng/ml FGF19 (Peprotech, 100-32)、50 μg/ml Primocin、500 nM A83-01、25 ng/ml BMP7、10 μM DAPT (Sigma, D5942)、30 μM地塞米松(Sigma, D4902)连续8天。gydF4y2Ba
如前所述,建立和培养胎儿肝细胞来源的类器官gydF4y2Ba58gydF4y2Ba.人胎肝组织切片,用100 μg/ml胶原酶IV型(Sigma, C5138)消化5 min。用Advanced DMEM/F12 + 2 mM GlutaMAX、10 mM HEPES、100 U/ml PenStrep清洗细胞,100 μm过滤器过滤后,镀膜。待基质凝固后,加入HEP介质。HEP培养基由Advanced DMEM/F12添加2 mM GlutaMAX, 10 mM HEPES, 100 U/ml PenStrep, 2%不含维生素A的B27, 15% RSPO1条件培养基,2.5 mM烟酰胺,1.25 mM组成gydF4y2BaNgydF4y2Ba-乙酰半胱氨酸,3 μM ir -99021 (Sigma, SML1046), 50 μg/ml Primocin, 50 ng/ml FGF7 (Peprotech, AF-100-19), 50 ng/ml FGF10, 50 ng/ml HGF, 50 μM Y-27632 (Abmole Bioscience, M1817), 1 μM A83-01, 20 ng/ml TGFα (Peprotech, 100-16 A), 50 ng/ml EGF, 10 nM Gastrin。培养基每2-3天更新一次,类器官每周1:2-1:5传递。gydF4y2Ba
肝内胆管细胞和胎儿肝细胞来源类器官的RNA测序gydF4y2Ba
对于RNA测序分析,我们包括肝脏样本、肝内胆管细胞来源的类器官和如上所述培养的胎儿肝细胞类器官。使用Trizol LS试剂(Invitrogen)分离RNA,并在-80°C保存,直到进一步处理。mRNA用Poly(A) Beads (NEXTflex)分离。RNA完整性使用Agilent RNA 6000 Nano试剂盒进行评估,浓度使用Qubit RNA HS检测试剂盒进行测定。仅使用RIN > 8.0的RNA样本进行测序。测序文库使用快速定向RNA-Seq Kit (NEXTflex)制备,并在NextSeq500 (Illumina)上测序,产生75个碱基长序列(Utrecht Sequencing Facility)。gydF4y2Ba
原始读取处理和规范化管道gydF4y2Ba
RNA-seq数据的原始读数来自欧洲核苷酸档案(ENA,gydF4y2Bahttps://www.ebi.ac.uk/enagydF4y2Ba).使用Galaxy (gydF4y2Bahttps://usegalaxy.eu/gydF4y2Ba)网上平台gydF4y2Ba79gydF4y2Ba.使用FastQC工具(Galaxy Version 0.72)评估样品质量。低质量的读取和适配器序列使用Cutadapt (Galaxy Version 1.66.6)修剪。使用RNA STAR工具(Galaxy Version 2.7.2b)对原始reads进行比对并定量基因表达。使用默认参数将Reads映射到Gencode人类参考基因组序列release 33 (GRCh38.p13)和Gencode综合基因注释v33。读取计数使用RNA STAR工具中的“-quantMode GeneCounts”选项获得。通过使用' DESeq2 ' R包对读取计数应用DESeq2方差稳定转换(VST)来获得标准化计数gydF4y2Ba80gydF4y2Ba然后使用' preprocessCore ' R包进行分位数规范化gydF4y2Ba81gydF4y2Ba.关于我们的正常化管道的其他信息在补充说明中提供gydF4y2Ba1gydF4y2Ba和补充图。gydF4y2Ba9gydF4y2Ba.gydF4y2Ba
主成分分析gydF4y2Ba
主成分分析(PCA)使用归一化计数进行,并采用' DESeq2 ' R包中的R函数' plotPCA '(包括前5000个最高方差基因),使用' ggplot2 ' R包绘制。gydF4y2Ba
欧几里得距离和基于距离的相似性gydF4y2Ba
欧几里得距离是通过将R函数dist (method= Euclidean)应用于归一化计数来计算的。定义了基于距离的相似度评分(DBS), DBS为“1”表示与PHH对照完全相似,“0”表示样本与PHH对照最不相似。每个样品的DBS由以下公式得到:gydF4y2Ba
马克斯gydF4y2Ba收购PHHgydF4y2Ba:某一基因集样本矩阵中到PHH的最大距离值。gydF4y2Ba
经销gydF4y2Ba收购PHHgydF4y2Ba:样本到PHH的距离值。gydF4y2Ba
基因表达热图gydF4y2Ba
为了可视化基因表达,标准化计数以每一行或基因(Log2FC RNA表达)的均值为中心,并使用' pheatmap ' R包在热图中绘制。当热图中没有簇树时,按细胞源类型排列列。gydF4y2Ba
CellNet分析gydF4y2Ba
大量RNA-Seq CellNet管道被用于量化基因表达估计,如前所述gydF4y2Ba65gydF4y2Ba,使用' cn_salmon '函数比对参考基因组GRCh38。基于来自97项研究的14种细胞和组织训练的人类cnProc_HS_RS_Jun_20_2017对象,使用' cn_apply '函数进行分类和基因调控网络(GRN)状态分析。使用“pheatmap”R包导出组织分类评分并绘制在热图中。使用' ggplot2 ' R包导出GRN状态分数并绘制成柱状图。使用“肝脏”的“cn_nis_all”函数计算组织特异性转录因子的网络影响评分。使用“pheatmap”R包导出网络影响分数,并在热图中与相应基因的标准化表达一起绘制。gydF4y2Ba
成人与胎儿肝细胞鉴别的分类gydF4y2Ba
采用不同细胞类型(原代人肝细胞、胎儿肝细胞、总胆管、成纤维细胞和多能干细胞)中方差最大的前5000个基因,使用“randomForest”R包构建随机森林分类器。用于训练分类器的样本在补充数据中列出gydF4y2Ba2 gydF4y2Ba.用出袋误差率来评价分类器的性能。gydF4y2Ba
转录组分析与蛋白质组的比较gydF4y2Ba
将不同HepG2细胞相对于对照PHH/肝脏的基因表达与Tascher等人公开的HepG2细胞和PHH的蛋白质组学数据进行比较。gydF4y2Ba68gydF4y2Ba.在3995个鉴定出的蛋白质中,3703个(93%)可以与转录组分析中包含的唯一基因匹配。在ernichR中进行富集分析gydF4y2Ba82gydF4y2Ba利用差异丰度蛋白(TukeygydF4y2BapgydF4y2Ba值< 0.05,lfc > 1;数据来自Tascher等人。gydF4y2Ba68gydF4y2Ba)和差异表达基因(padj <0.05, LFC >1)作为单独输入。蛋白质组和转录组富集评分分别定义为HepG2和PHH样本中上调和下调基因的比值比的log2和反向比值比的log2。gydF4y2Ba
功能分析gydF4y2Ba
根据制造商的说明,使用尿素测定试剂盒(Abcam, ab83362)测定尿素分泌。简单地说,在培养基中加入1mm氯化铵。24 h后,测定培养基中尿素浓度。根据制造商的建议,使用P450-Glo CYP3A4检测试剂盒(Promega, V9001)测量CYP3A4活性。所有数据归一化,使用Pierce BCA蛋白测定试剂盒(赛默飞世尔科学公司,23225)测量总蛋白含量。gydF4y2Ba
PHH/肝脏质量控制HLCompR纳入gydF4y2Ba
使用随机森林分类器评估新PHH/肝脏数据的质量。图中使用的RNA-seq数据。gydF4y2Ba2 gydF4y2Ba作为构建分类器的训练数据集。当新的PHH/肝脏样本PHH/肝脏分类概率低于45%时,不建议使用数据集。gydF4y2Ba
统计和再现性gydF4y2Ba
使用GraphPad Prism 8进行统计分析(Pearson和Spearman相关)。当gydF4y2BapgydF4y2Ba< 0.05。样本大小一般在图中表示。对于转录组学研究,我们在每种肝细胞样细胞(HLC)模型中至少纳入两个样本,如果有的话。在旨在比较HLCs功能能力的实验中,我们为每个HLCs模型至少包括两个生物重复。除非另有说明,柱状图以均值±标准差表示,盒状-须状图以中位数(线)、四分位范围(盒)和数据范围或1.5倍四分位范围(须)表示。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
本研究中包括的先前发表的分析报告和基因表达数据的摘要可在补充数据中获得gydF4y2Ba1gydF4y2Ba.本研究中使用的rna测序数据列表可在补充数据中获得gydF4y2Ba2 gydF4y2Ba.本研究中使用的rna测序数据的原始计数、归一化计数和元数据可在补充数据中获得gydF4y2Ba4 gydF4y2Ba.数据背后的源数据可在补充数据中找到gydF4y2Ba3.gydF4y2Ba- - - - - -gydF4y2Ba8gydF4y2Ba.本研究中处理过的rna测序数据已存入NCBI基因表达综合数据库,登录号为GSE214097。由于潜在的信息可能会影响捐赠者的同意,原始rna测序数据无法公开。支持本研究结果的所有材料均可根据合理要求从通讯作者处获得。gydF4y2Ba
代码的可用性gydF4y2Ba
用于运行HLCompR web应用程序的R代码可在GitHub (gydF4y2Bahttps://github.com/iardisasmita/HLCompRgydF4y2Ba)和泽诺多(gydF4y2Bahttps://doi.org/10.5281/zenodo.7071219gydF4y2Ba).进一步的要求可以直接联系相应的作者。gydF4y2Ba
参考文献gydF4y2Ba
Kmiec Z。gydF4y2Ba肝细胞在健康与疾病中的合作gydF4y2Ba.gydF4y2Ba放置阿娜特。Embryol。细胞生物。gydF4y2Ba161gydF4y2Ba, 1-151(2001)。gydF4y2Ba
刘建军,刘建军,刘建军,刘建军。关于成人肝细胞来源和肝细胞体外增殖的争议。gydF4y2Ba细胞。摩尔。杂志。乙醇。gydF4y2Ba11gydF4y2Ba, 273-290(2021)。gydF4y2Ba
傅,G. B.等。人肝细胞来源的肝祖样细胞的扩增和分化及其在嗜肝病原体研究中的应用。gydF4y2Ba细胞Res。gydF4y2Ba29gydF4y2Ba, 8-22(2019)。gydF4y2Ba
Hu, H.等。功能性小鼠和人肝细胞作为三维类器官的长期扩展。gydF4y2Ba细胞gydF4y2Ba175gydF4y2Ba, 1591 - 1606。e19(2018)。gydF4y2Ba
Kim, Y.等人。小分子介导的人肝细胞重编程成双能祖细胞。gydF4y2Baj .乙醇。gydF4y2Ba70gydF4y2Ba, 97-107(2019)。gydF4y2Ba
Xiang, C.等。原代人肝细胞体外长期功能维持。gydF4y2Ba科学gydF4y2Ba364gydF4y2Ba, 399-402(2019)。gydF4y2Ba
张,K.等。具有高效肝再生能力的原代人肝细胞体外扩增。gydF4y2Ba细胞干细胞gydF4y2Ba23gydF4y2Ba, 806 - 819。e4(2018)。gydF4y2Ba
Avior, Y.等。微生物来源的石胆酸和维生素K2驱动多能干细胞来源和胎儿肝细胞的代谢成熟。gydF4y2Ba肝脏病学gydF4y2Ba62gydF4y2Ba, 265-278(2015)。gydF4y2Ba
Du, Y.等。由成纤维细胞经谱系重编程诱导具有药物代谢功能的人肝细胞。gydF4y2Ba细胞干细胞gydF4y2Ba14gydF4y2Ba, 394-403(2014)。gydF4y2Ba
段勇,等。人胚胎干细胞代谢功能肝细胞的分化与鉴定。gydF4y2Ba干细胞gydF4y2Ba28gydF4y2Ba, 674-686(2010)。gydF4y2Ba
傅,Y.等。从人脂肪来源的干细胞快速生成功能性肝细胞样细胞。gydF4y2Ba干细胞Res. Ther。gydF4y2Ba7gydF4y2Ba, 1-12(2016)。gydF4y2Ba
高,Y.等。同一供体不同策略产生的肝细胞样细胞的不同基因表达和表观遗传特征。gydF4y2Ba干细胞代表gydF4y2Ba9gydF4y2Ba, 1813-1824(2017)。gydF4y2Ba
Gieseck, R. L.等。三维培养诱导多能干细胞肝细胞的成熟。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Ba, e86372(2014)。gydF4y2Ba
汉南,N. R. F., Segeritz, C. P., Touboul, T. & Vallier, L.从人多能干细胞中生产肝细胞样细胞。gydF4y2BaProtoc Nat。gydF4y2Ba8gydF4y2Ba, 430-437(2013)。gydF4y2Ba
黄娟,郭晓霞,李伟,张慧。GSK3抑制剂激活Wnt/β-catenin信号通路直接诱导人脂肪干细胞向功能性肝细胞分化。gydF4y2Ba科学。代表。gydF4y2Ba7gydF4y2Ba, 1-12(2017)。gydF4y2Ba
Huang, P.等。直接重编程人类成纤维细胞到功能性和可扩张肝细胞。gydF4y2Ba细胞干细胞gydF4y2Ba14gydF4y2Ba, 370-384(2014)。gydF4y2Ba
等等。成人肝脏基因组稳定双能干细胞的长期培养。gydF4y2Ba细胞gydF4y2Ba160gydF4y2Ba, 299-312(2015)。gydF4y2Ba
Kanninen, L. K.等。基于Laminin-511和laminin-521的矩阵用于人类多能干细胞的高效肝脏规范。gydF4y2Ba生物材料gydF4y2Ba103gydF4y2Ba, 86-100(2016)。gydF4y2Ba
巴斯马,H.等。人胚胎干细胞源性肝细胞的分化与移植。gydF4y2Ba胃肠病学gydF4y2Ba136gydF4y2Ba, 990 - 999。e4(2009)。gydF4y2Ba
近藤,Y.等。组蛋白去乙酰酶抑制剂丙戊酸促进人诱导多能干细胞向肝细胞样细胞分化。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Ba, 1-11(2014)。gydF4y2Ba
Koui, Y.等。体外人肝模型由ipsc来源的实质和非实质细胞。gydF4y2Ba干细胞代表gydF4y2Ba9gydF4y2Ba, 490-498(2017)。gydF4y2Ba
李,Z.等。在化学条件下从人胚胎干细胞中产生合格的临床级功能性肝细胞。gydF4y2Ba细胞死亡gydF4y2Ba.gydF4y2Ba10gydF4y2Ba, 763(2019)。gydF4y2Ba
罗,Y.等。三维水凝胶培养条件促进人诱导多能干细胞向肝细胞分化。gydF4y2BaCytotherapygydF4y2Ba20.gydF4y2Ba, 95-107(2018)。gydF4y2Ba
Mun, S. J.等。可扩展的人多能干细胞来源的肝细胞样肝类器官的生成。gydF4y2Baj .乙醇。gydF4y2Ba71gydF4y2Ba, 970-985(2019)。gydF4y2Ba
Nakamori, D., Akamine, H., Takayama, K., Sakurai, F. & Mizuguchi, H.通过ATF5, PROX1, FOXA2, FOXA3和HNF4A转导将人成纤维细胞直接转化为肝细胞样细胞。gydF4y2Ba科学。代表gydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 16675(2017)。gydF4y2Ba
大仓,H.等人。人脂肪组织来源间充质干细胞的肝细胞样细胞簇的性质。gydF4y2Ba组织中。部分。C方法gydF4y2Ba16gydF4y2Ba, 761-770(2010)。gydF4y2Ba
Pettinato, G.等人。通过直接抑制Wnt/β-catenin通路,人诱导多能干细胞在多细胞球形三维培养中可扩展分化为肝细胞样细胞。gydF4y2Ba科学。代表。gydF4y2Ba6gydF4y2Ba, 1-17(2016)。gydF4y2Ba
Pettinato, G.等人。从人诱导多能干细胞与内皮细胞混合生成全功能肝细胞样器官。gydF4y2Ba科学。代表。gydF4y2Ba9gydF4y2Ba, 1-21(2019)。gydF4y2Ba
Roelandt, P.等人。人胚胎干细胞和大鼠成体干细胞具有原始的内胚层样表型,最终可发育为定型的内胚层样细胞,最终可发育为肝细胞样细胞。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba5gydF4y2Ba, e12101(2010)。gydF4y2Ba
布恩,R.等。氨基酸水平决定肝细胞和肝癌细胞系的代谢和CYP450功能。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba11gydF4y2Ba, 1393(2020)。gydF4y2Ba
绍尔,V.等。利用干细胞技术,人尿上皮细胞作为可移植肝细胞样细胞的来源。gydF4y2Ba细胞Transpl。gydF4y2Ba25gydF4y2Ba, 2221-2243(2016)。gydF4y2Ba
Schneeberger, K.等人。lgr5阳性双潜能人肝干细胞的大规模生产。gydF4y2Ba肝脏病学gydF4y2Ba72gydF4y2Ba, 257-270(2020)。gydF4y2Ba
Sengupta, S.等人。人胚胎干细胞肝细胞悬浮聚合培养是一种改进的体外药物代谢和毒性试验模型。gydF4y2BaToxicol。科学。gydF4y2Ba140gydF4y2Ba, 236-245(2014)。gydF4y2Ba
Shan, J.等。鉴定用于人肝细胞扩张和iPS分化的小分子。gydF4y2BaNat,化学。医学杂志。gydF4y2Ba9gydF4y2Ba, 514-520(2013)。gydF4y2Ba
Si-Tayeb, K.等。从诱导多能干细胞高效生成人肝细胞样细胞。gydF4y2Ba肝脏病学gydF4y2Ba51gydF4y2Ba, 297-305(2010)。gydF4y2Ba
高山,K.等。通过FOXA2和HNF1α转导,从人多能干细胞中产生代谢功能肝细胞。gydF4y2Baj .乙醇。gydF4y2Ba57gydF4y2Ba, 628-636(2012)。gydF4y2Ba
高山,K.等。通过HNF4α转导从人胚胎干细胞和诱导多能干细胞高效生成功能性肝细胞。gydF4y2Ba摩尔。其他。gydF4y2Ba20.gydF4y2Ba, 127-137(2012)。gydF4y2Ba
高山,K.等。用于再生医学的安全和治疗有效的人诱导多能干细胞来源的肝细胞样细胞的生成。gydF4y2Ba乙醇。Commun。gydF4y2Ba1gydF4y2Ba, 1058-1069(2017)。gydF4y2Ba
Tang, W.等。化学鸡尾酒使人类尿源性细胞的肝脏重编程与单一的转录因子。gydF4y2Ba学报杂志。罪。gydF4y2Ba40gydF4y2Ba, 620-629(2019)。gydF4y2Ba
杜永春,刘志刚,刘志刚,刘志刚。人多能干细胞诱导肝细胞样细胞分化的研究进展。gydF4y2Ba生物材料gydF4y2Ba70gydF4y2Ba, 115-125(2015)。gydF4y2Ba
卡拉布雷斯,D.等。肝活检衍生的诱导多能干细胞为肝样细胞的生成提供了无限的供应。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba14gydF4y2Ba, 1-27(2019)。gydF4y2Ba
Tasnim, F.等人。功能性增强的人干细胞来源的肝细胞在半乳糖化纤维海绵用于肝毒性测试。gydF4y2Ba摩尔。制药。gydF4y2Ba13gydF4y2Ba, 1947-1957(2016)。gydF4y2Ba
Touboul, T.等人。WNT/β-catenin通路的分期特异性调控增强了hESCs向肝细胞的分化。gydF4y2Baj .乙醇。gydF4y2Ba64gydF4y2Ba, 1315-1326(2016)。gydF4y2Ba
Unzu, C.等。原代人肝细胞有效扩增的祖细胞状态的药理学诱导。gydF4y2Ba肝脏病学gydF4y2Ba69gydF4y2Ba, 2214-2231(2019)。gydF4y2Ba
Waclawczyk, S., Buchheiser, A., Flögel, U., Radke, T. F. & Kögler, G.无限制体细胞干细胞体外分化为功能性肝样细胞,显示肝细胞样葡萄糖代谢。gydF4y2Baj .细胞。杂志。gydF4y2Ba225gydF4y2Ba, 545-554(2010)。gydF4y2Ba
王,Q.等。从扩展多能干细胞生成人肝细胞。gydF4y2Ba细胞Res。gydF4y2Ba30.gydF4y2Ba, 810-813(2020)。gydF4y2Ba
王哲,等。使用人肝细胞来源的肝祖样细胞生成肝球状体用于肝毒性筛选。gydF4y2Ba开展gydF4y2Ba9gydF4y2Ba, 6690-6705(2019)。gydF4y2Ba
王,S.等。人esc衍生的可扩张肝类器官使治疗性肝再生和酒精性肝损伤的病理生理建模成为可能。gydF4y2Ba细胞Res。gydF4y2Ba29gydF4y2Ba, 1009-1026(2019)。gydF4y2Ba
谢B.等。一种两步谱系重编程策略,从成纤维细胞产生功能有能力的人肝细胞。gydF4y2Ba细胞Res。gydF4y2Ba29gydF4y2Ba, 696-710(2019)。gydF4y2Ba
赵,D.等。通过纠正规格缺陷促进人类多能干细胞来源的肝细胞的高效代谢成熟。gydF4y2Ba细胞Res。gydF4y2Ba23gydF4y2Ba, 157-161(2013)。gydF4y2Ba
朱,S.等。用人类成纤维细胞产生的肝细胞再生小鼠肝脏。gydF4y2Ba自然gydF4y2Ba508gydF4y2Ba, 93-97(2014)。gydF4y2Ba
Carpentier, A.等人。适合于高通量筛选的小型化形式的人类多能干细胞的肝脏分化。gydF4y2Ba干细胞研究。gydF4y2Ba16gydF4y2Ba, 640-650(2016)。gydF4y2Ba
陈玉峰等。快速生成成熟的肝细胞样细胞从人诱导多能干细胞通过一个有效的三步协议。gydF4y2Ba肝脏病学gydF4y2Ba55gydF4y2Ba, 1193-1203(2012)。gydF4y2Ba
Chivu等人。人骨髓间充质干细胞在肝特异性因子差异暴露下的体外肝分化。gydF4y2BaTransl。Res。gydF4y2Ba154gydF4y2Ba, 122-132(2009)。gydF4y2Ba
乔杜里等人。诱导多能干细胞衍生的患者特异性肝细胞样细胞模型pazopanib介导的肝毒性。gydF4y2Ba科学。代表。gydF4y2Ba7gydF4y2Ba, 41238(2017)。gydF4y2Ba
Du, C.等。通过纯小分子鸡尾酒从人类多能干细胞高效和加速肝脏分化。gydF4y2Ba干细胞Res. Ther。gydF4y2Ba9gydF4y2Ba, 58(2018)。gydF4y2Ba
Ulvestad, M.等人。人胚胎和诱导多能干细胞肝细胞的药物代谢酶和转运蛋白谱。gydF4y2Ba物化学。杂志。gydF4y2Ba86gydF4y2Ba, 691-702(2013)。gydF4y2Ba
Hendriks, D., Artegiani, B., Hu, H., Chuva de Sousa Lopes, S. & Clevers, H.在人肝脏类器官培养物中建立人胎儿肝细胞类器官和基于crispr - cas9的基因敲入和敲除。gydF4y2BaProtoc Nat。gydF4y2Ba16gydF4y2Ba, 182-217(2021)。gydF4y2Ba
彭,W. C., Kraaier, L. J. & Kluiver, T. A.肝细胞类器官和细胞移植:未来会怎样。gydF4y2BaExp、Mol、Med。gydF4y2Ba53gydF4y2Ba, 1512-1528(2021)。gydF4y2Ba
Rimland, c.a.等。人类胆道组织的区域差异及相应的体外衍生类器官。gydF4y2Ba肝脏病学gydF4y2Ba73gydF4y2Ba, 247-267(2021)。gydF4y2Ba
杨,Y.等。获得具有体内胚胎和胚胎外潜能的多能干细胞。gydF4y2Ba细胞gydF4y2Ba169gydF4y2Ba, 243 - 257。e25(2017).
Aizarani, N.等。人肝细胞图谱揭示了异质性和上皮祖细胞。gydF4y2Ba自然gydF4y2Ba572gydF4y2Ba, 199-204(2019)。gydF4y2Ba
Cunningham, r.p. & Porat-Shliom, N.肝脏分区-用新技术重新审视旧问题。gydF4y2Ba前面。杂志。gydF4y2Ba12gydF4y2Ba, 1-17(2021)。gydF4y2Ba
Cahan, P.等人。CellNet:应用于干细胞工程的网络生物学。gydF4y2Ba细胞gydF4y2Ba158gydF4y2Ba, 903-915(2014)。gydF4y2Ba
拉德利,A. H.等。利用CellNet和RNA-seq评估工程细胞。gydF4y2BaProtoc Nat。gydF4y2Ba12gydF4y2Ba, 1089-1102(2017)。gydF4y2Ba
莫里斯,s.a.等。通过细胞网解剖工程细胞类型和增强细胞命运转换。gydF4y2Ba细胞gydF4y2Ba158gydF4y2Ba, 889-902(2014)。gydF4y2Ba
巴克斯特,等人。表型和功能分析表明,干细胞来源的肝细胞样细胞更好地模拟胎儿而不是成人肝细胞。gydF4y2Baj .乙醇。gydF4y2Ba62gydF4y2Ba, 581-589(2015)。gydF4y2Ba
塔舍,G.等人。深入的蛋白质组分析强调了肝细胞作为原代人肝细胞的多功能细胞系统替代品。gydF4y2Ba细胞gydF4y2Ba8gydF4y2Ba, 1-25(2019)。gydF4y2Ba
Schwanhüusser, B.等。哺乳动物基因表达控制的全球定量研究。gydF4y2Ba自然gydF4y2Ba473gydF4y2Ba, 337-342(2011)。gydF4y2Ba
Häberle, J.等。尿素循环紊乱的诊断和管理建议指南。gydF4y2Ba欧菲尼特。gydF4y2Ba7gydF4y2Ba, 0-1(2012)。gydF4y2Ba
Müller, F. A. & Sturla, S. J.非酒精性脂肪肝疾病的人类体外模型。gydF4y2Ba咕咕叫。当今。Toxicol。gydF4y2Ba16gydF4y2Ba, 9-16(2019)。gydF4y2Ba
ter Braak, B.等人。基于转录组的肝干细胞衍生子代和原代人肝细胞细胞适应性应激反应激活网络的系统比较。gydF4y2BaToxicol。在体外gydF4y2Ba73gydF4y2Ba, 105107(2021)。gydF4y2Ba
关,Y.等。用于分析人类遗传疾病的人类肝脏类器官。gydF4y2Ba江森自控的洞察力gydF4y2Ba2 gydF4y2Ba, e94954(2017)。gydF4y2Ba
古普塔等人。利用RNA-Seq对体外人肝模型与体内人肝进行比较。gydF4y2Ba拱门。Toxicol。gydF4y2Ba95gydF4y2Ba, 573-589(2021)。gydF4y2Ba
Shi Y.等。在营养基因组学对膳食补充omega-3脂肪酸反应的个体差异中,鉴定可获得的肝脏基因签名。gydF4y2Ba细胞gydF4y2Ba10gydF4y2Ba, 467(2021)。gydF4y2Ba
Vieyres, G., Reichert, I., Carpentier, A., Vondran, F. W. R. & Pietschmann, T. ATGL脂肪酶与ABHD5合作动员脂质用于丙型肝炎病毒组装。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba16gydF4y2Ba, 1-41(2020)。gydF4y2Ba
斯坦利,洛杉矶&沃尔夫,c.r透过玻璃,黑暗?HepaRG和HepG2细胞作为人类I期药物代谢模型。gydF4y2Ba药物。金属底座。牧师。gydF4y2Ba54gydF4y2Ba, 46-62(2022)。gydF4y2Ba
Rombaut等人。体细胞直接重编程成诱导肝细胞:破解恩尼格玛密码。gydF4y2Baj .乙醇gydF4y2Ba.gydF4y2Ba75gydF4y2Ba, 690-705(2021)。gydF4y2Ba
Afgan, E.等。用于可访问、可复制和协作生物医学分析的银河平台:2018年更新。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, w537-w544(2018)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 1-21(2014)。gydF4y2Ba
Bolstad, B. M., Irizarry, R. A., Åstrand, M. & Speed, T. P.基于方差和偏差的高密度寡核苷酸阵列数据归一化方法的比较。gydF4y2Ba生物信息学gydF4y2Ba19gydF4y2Ba, 185-193(2003)。gydF4y2Ba
陈,e.y .等。enrichment:交互式协作HTML5基因列表丰富分析工具。gydF4y2BaBMC Bioinform。gydF4y2Ba14gydF4y2Ba, 128(2013)。gydF4y2Ba
确认gydF4y2Ba
这项研究得到了ZonMW TAS(“用干细胞再生肠道组织”项目)和Metakids(“Minilevertjes”项目)的支持。这项研究是由国家多学科合作代谢性疾病联合(UMD)促进的,以改善对代谢性疾病患者的护理。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
概念化:A.I.A I.F.S。规则,和S.A.F.;形式分析:A.I.A.和I.F.S.;调查:a.i.a.、i.f.s.、I.P.J、G.K.;资源:d.h., b.a., S.A.F.;软件:A.I.A、i.f.s.、G.K.;写作-初稿:A.I.A.和I.F.S.;写作——审查和编辑:A.I.A, I.F.S, I.P.J,答,E.E.S.N, S.A.F.;可视化:A.I.A.和I.F.S.;监督:E.E.S.N.和S.A.F;以及资金收购:E.E.S.N.和S.A.F.gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
通信生物学gydF4y2Ba感谢孙天良、Nadim Aizarani和其他匿名审稿人对这项工作的同行评审所做的贡献。主要处理编辑:Simona Chera和George Inglis。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Ardisasmita, a.i., Schene, i.f., Joore, I.P.gydF4y2Baet al。gydF4y2Ba肝细胞模型系统的全面转录组比较提高了实验使用模型的选择。gydF4y2BaCommun杂志gydF4y2Ba5gydF4y2Ba, 1094(2022)。https://doi.org/10.1038/s42003-022-04046-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s42003-022-04046-9gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba