摘要gydF4y2Ba
了解所有人冠状病毒(hcov) Spike (S)蛋白的抗原特征对于泛hcov表位鉴定和广泛有效的疫苗开发至关重要。为了描述目前难以捉摸的α-HCoVs蛋白的抗原特征,我们分离了一组抗HCoV-229E S蛋白的抗体,并表征了它们的表位和中和潜能。我们发现HCoV-229E S蛋白的n端结构域具有抗原性优势,其中存在一个抗原超位点,在HCoV-NL63中表现保守,具有作为泛α- hcov表位的潜力。在受体结合域中,中和表位在受体结合位点远端的末端被捕获,这让人想起SARS-CoV-2 RBD隐性表位的位置。我们还发现了一种识别连接器结构域的中和抗体,因此代表了首个针对α- hcov的s2定向中和抗体。HCoVs蛋白在属间抗原相似性和差异的揭示突出了泛hcov疫苗设计面临的挑战,同时支持针对hcov子集开发广泛有效疫苗的可行性。gydF4y2Ba
简介gydF4y2Ba
作为RNA病毒,冠状病毒(CoVs)不断进化,经常从蝙蝠等自然宿主跳到人类体内gydF4y2Ba1gydF4y2Ba.目前可感染人类的新型冠状病毒有7种,包括α属HCoV-229E (229E)和HCoV-NL63 (NL63), β属HCoV-OC43 (OC43)、HCoV-HKU1 (HKU1)、MERS-CoV、SARS-CoV和SARS-CoV-2,均为人畜共患病源gydF4y2Ba2gydF4y2Ba.在这些人类新型冠状病毒(hcov)中,SARS-CoV、MERS-CoV和SARS-CoV-2最近在人群中外溢,具有高传染性和高致病性。与此同时,其他hcov,如229E,很久以前就跨越了物种壁垒,已经适应了与人类共存,通常会引起自限性呼吸道感染,但对儿童、老年人和免疫功能低下的人来说可能是致命的gydF4y2Ba3.gydF4y2Ba.鉴于未来10至50年内再次发生冠状病毒外溢的可能性很大,制定广泛有效的应对冠状病毒的对策是全球优先事项gydF4y2Ba4 gydF4y2Ba.然而,尽管最近针对SARS-CoV-2的疫苗和治疗方法取得了进展,但目前还没有具有泛hcov活性的疫苗。gydF4y2Ba
新型冠状病毒的刺突(S)蛋白介导其进入宿主,是开发新型冠状病毒疫苗或治疗方法的主要靶点gydF4y2Ba5gydF4y2Ba.S蛋白由S1和S2两个亚基组成。S1亚基包含n端结构域(NTD)和c端结构域(CTD),两者都可参与宿主受体识别和病毒附着gydF4y2Ba6克ydF4y2Ba.同时,S2子单元是一个弹簧加载的聚变装置gydF4y2Ba7gydF4y2Ba.在宿主受体附着之前,S蛋白通常采用亚稳态预融合构象,其中S1三聚体盖住了S2三聚体的柄。当宿主受体参与和S1和S2蛋白水解分离时,S2s中原本埋藏的融合肽(FPs)暴露出来并插入到宿主膜中,进而触发S2s内七角重复序列(HRs)的重排,形成3HR1-3HR2六螺旋束(6-HB),从而使病毒和宿主膜靠近,促进膜融合gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba.值得注意的是,α-和β-HCoVs S蛋白在融合前构象上存在巨大差异,例如它们的NTDs和CTDs之间的包装模式不同gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba.此外,来自SARS-CoV、MERS-CoV和SARS-CoV-2的CTDs在融合前状态下具有相似频率的“up”和“down”构象,而来自α- hcov的CTDs仅以受体无法接近的“down”构象捕获gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba.总之,这些结构差异可能导致α-和β-HCoVs S蛋白的免疫原性不同。gydF4y2Ba
本世纪SARS-CoV和MERS-CoV的相继出现,以及前所未有的SARS-CoV-2大流行,促使人们从患者血清或抗体库和S蛋白免疫小鼠中发现了针对它们的中和抗体(nab)gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba.这些报道的nab大多靶向它们的CTDs,这也是它们的受体结合结构域(RBDs),而剩下的少数则识别NTDs或S2亚基,这表明rbd在这三种β- hcov中具有免疫显性作用gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba.事实上,据报道,COVID-19恢复期血清中90%的中和活性都是针对SARS-CoV-2 RBD的gydF4y2Ba20.gydF4y2Ba.rbd定向的NAbs一般通过直接或间接阻断S蛋白与宿主受体之间的相互作用起作用,而定向到S蛋白其他区域的NAbs的工作机制可能涉及空间位阻宿主受体结合或抑制S蛋白构象前后的变化gydF4y2Ba21gydF4y2Ba.值得注意的是,针对这三种高致病性β- cov的ntd导向的NAbs经常在其ntd上识别一个相似的向外的无甘聚糖区(补充图)。gydF4y2Ba4gydF4y2Ba)gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,甚至发现几乎所有SARS-CoV-2 ntd特异性nab都能识别单个“抗原超位点”gydF4y2Ba25gydF4y2Ba.巧合的是,最近对普通感冒β-HCoV OC43的两项研究也揭示了它S蛋白NTD中存在两个相邻的“抗原超位点”gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.与S1帽相比,融合装置S2在不同的hcov中似乎更加保守。然而,尽管S2碱基可能作为干预靶点,具有更好的跨物种潜力gydF4y2Ba30.gydF4y2Ba, S2亚基的抗原描述仍然有限。到目前为止,只有少数s2导向的nab被描述用于上述三种高致病性β- hcovgydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Baα- hcov尚未见此类nab报道。gydF4y2Ba
与广泛的针对β-HCoVs S蛋白的抗体研究相比,针对α-HCoVs S蛋白的抗体的发现和鉴定非常少。为了探索泛hcov疫苗开发的可行性,并在S蛋白上鉴定潜在的泛hcov表位,了解不同hcov之间S蛋白的免疫原性差异非常重要,其中α属S蛋白的抗原格局目前尚不清楚。本文以229E为代表的α-HCoV,旨在揭示其S蛋白的抗原特征。我们从噬菌体展示库中鉴定了一组针对229E S蛋白的NTD, CTD(也包括其RBD)和S2亚基的抗体,并描述了所选抗体的中和能力。接下来,我们使用了结合氢-氘交换耦合质谱(HDX-MS)的综合结构生物学方法。gydF4y2Ba39gydF4y2Ba、冷冻电镜和x射线晶体学,以了解每种抗体的结合模式。总之,这些工作揭示了229E S蛋白的抗原特征,使我们能够更好地理解α-和β-HCoVs蛋白之间的区别和相似之处,这不仅突出了泛hcov疫苗设计面临的挑战,而且揭示了开发针对hcov亚群的广泛有效疫苗的机会。gydF4y2Ba
结果gydF4y2Ba
抗229E S三聚体抗体的筛选与验证gydF4y2Ba
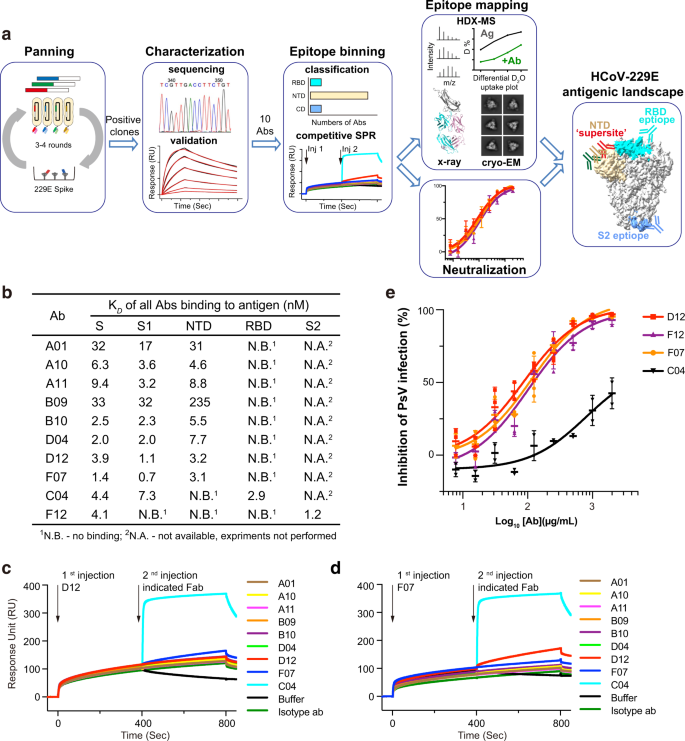
我们使用噬菌体展示的合成人抗原结合片段(Fabs)文库(F文库)来选择能够识别229E S三聚体的抗体(图2)。gydF4y2Ba1gydF4y2Ba,补充图。gydF4y2Ba1得了gydF4y2Ba).F库的理论多样性为3 × 10gydF4y2Ba10gydF4y2Ba并成功地选择了多种蛋白质,包括多种重要的治疗靶点,如IL-18、整合素和卷曲受体gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba.对229E S三聚体进行连续4步富集筛选,对ELISA中与229E S三聚体结合而不与阴性对照蛋白结合的单个阳性克隆进行DNA测序。总共鉴定出13个具有独特互补决定区(CDR)序列的fab。gydF4y2Ba1 dgydF4y2Ba).然后纯化重组fab,并通过表面等离子体共振(SPR)检测其与229E S三聚体的结合。具有平衡解离常数的13个Fabs结合229E S三聚体中的10个(gydF4y2BaKgydF4y2BaDgydF4y2Ba)范围为1-50 nM(图;gydF4y2Ba1 bgydF4y2Ba,补充图。gydF4y2Ba1 egydF4y2Ba).三种fab B03、B06和E12与229E S蛋白的结合很少或没有,因此被排除在后续的研究之外。gydF4y2Ba1 egydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba概述了229E S三聚体抗原景观定位过程的示意图。gydF4y2BabgydF4y2Ba用SPR表征了各Fab与229E S三聚体、S1或S2亚基、NTD和RBD之间的相互作用动力学。结果表明,C04直接作用于229E的RBD, F12直接作用于229E的S2亚基,其他晶圆均与229E的NTD相互作用。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba竞争SPR结果表明,8个NTD定向晶圆厂都在竞争229E NTD上类似的绑定接口。229es1固定在传感器上,然后用饱和Fab D12 (gydF4y2BacgydF4y2Ba)或F07 (gydF4y2BadgydF4y2Ba),然后只注入第二次缓冲液或指定的晶圆。Fab C04作为阳性对照。gydF4y2BaegydF4y2BaIgG D12(红色)、F12(紫色)、F07(橙色)和C04(黑色)对229E PsV的中和谱。中和活性表示为单个数据点和平均值±SD (gydF4y2BangydF4y2Ba= 3个生物独立实验)。Mean±SD表示为gydF4y2Ba .单个数据点分别显示为填充的红色方块(D12)、紫色三角形(F12)、橙色圆圈(F07)和黑色三角形(C04)。gydF4y2Ba
.单个数据点分别显示为填充的红色方块(D12)、紫色三角形(F12)、橙色圆圈(F07)和黑色三角形(C04)。gydF4y2Ba
抗体分类和下选gydF4y2Ba
接下来,我们表达并纯化229E的NTD、RBD、S1和S2亚基(补充图)。gydF4y2Ba2 a, bgydF4y2Ba),并分别评估它们与10个验证过的fab的结合(图。gydF4y2Ba1 bgydF4y2Ba,补充图。gydF4y2Ba2氟gydF4y2Ba).10个fab中有9个有效地结合S1亚基gydF4y2BaKgydF4y2BaDgydF4y2Ba与完整的229E S三聚体的亲和度相当的值(图2)。gydF4y2Ba1 bgydF4y2Ba,补充图。gydF4y2Ba2摄氏度gydF4y2Ba).与此同时,Fab F12与完整的S三聚体紧密结合,但与S1、NTD或RBD均无结合(图2)。gydF4y2Ba1 bgydF4y2Ba,补充图。gydF4y2Ba2 c, dgydF4y2Ba),提示其可能与S2亚基或仅存在于三聚体S中的结构表位结合gydF4y2BaDgydF4y2Ba值(1.2和4.1 nM)。gydF4y2Ba1 bgydF4y2Ba,补充图。gydF4y2Ba1 egydF4y2Ba,补充图。gydF4y2Ba二维gydF4y2Ba),证实F12的表位局限于S2亚基。在结合S1亚基的9个Fab中,只有Fab C04能识别RBD,其他8个抗体都能结合NTD(图2)。gydF4y2Ba1 bgydF4y2Ba,补充图。gydF4y2Ba2 e, fgydF4y2Ba).这一现象说明229E的NTD比其RBD更具有抗原性,这与之前对三种高致病性β- hcov的RBD而非NTDs具有免疫显性形成了鲜明的对比gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba.gydF4y2Ba
到目前为止,10个229E S反应晶片可以分为3个主要组,每个组都识别229E S的NTD, RBD或S2亚基(图2)。gydF4y2Ba1 bgydF4y2Ba).与分别包括C04和F12的组2和组3相比,ntd识别组1是一个由8个不同的fab组成的大组。为了了解这些fab是否与NTD上相似或不同的表位结合,我们进行了竞争性SPR,以检查它们与229E S1亚基的相互结合。考虑到针对相似表位的抗体对在第二种抗体的亲和力远高于第一种抗体时,会出现虚假的相互结合,因此我们首先使用最强的NTD结合剂D12(或F07)来饱和固定的S1蛋白,然后再与其他NTD fab进行挑战(图7)。gydF4y2Ba1 c, dgydF4y2Ba).第二次注射rbd结合C04作为阳性对照,确实诱导了干扰模式的显著改变,而第二次注射D12(或F07)作为有效阻断的对照(图7)。gydF4y2Ba1 c, dgydF4y2Ba).第二次注入任何其他NTD晶片几乎没有引起波长漂移,这表明它们的结合在D12(或F07)之前被阻断了(图12)。gydF4y2Ba1 c, dgydF4y2Ba).此外,F07和D12之间也未观察到并发结合(图12)。gydF4y2Ba1 c, dgydF4y2Ba).总之,这些结果表明,A01、A10、A11、B09、B10、D04、D12和F07这8个NTD导向晶圆厂都在NTD上竞争类似的结合位点。gydF4y2Ba
显性ntd定向抗体的表位和中和作用gydF4y2Ba
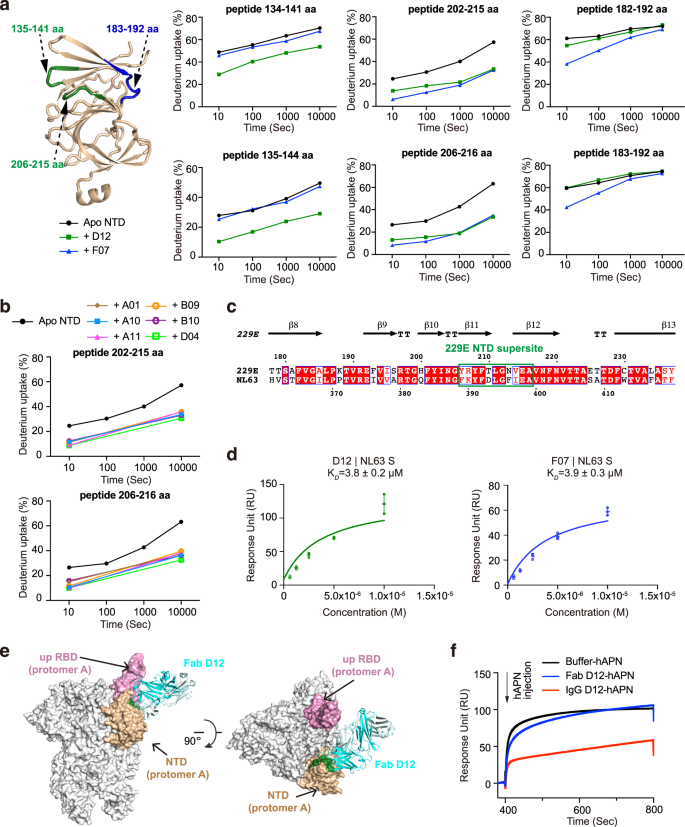
此前对β-HCoVs S蛋白的研究表明,SARS-CoV-2和OC43的ntd中存在“抗原超位点”gydF4y2Ba25gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.为了了解α- cov的ntd是否具有相似的抗原特征,我们开始绘制8个ntd定向fab的表位,并评估它们的中和潜力。我们首先使用HDX-MSgydF4y2Ba39gydF4y2Ba在229E的NTD上精确定位D12的表位(图。gydF4y2Ba2gydF4y2Ba,补充图。gydF4y2Ba3gydF4y2Ba,gydF4y2Ba7一个gydF4y2Ba).与NTD单独相比,在Fab D12存在的情况下,134-141 aa、135-144 aa、202-215 aa和206-216 aa在所有测量时间点的HDX均显著降低,这表明包含135-141 aa和206-215 aa的NTD区域可能参与了D12相互作用(图12)。gydF4y2Ba2gydF4y2Ba,补充图。gydF4y2Ba3gydF4y2Ba).接下来,使用HDX-MS对所有其他NTD抗体进行表位定位(补充图。gydF4y2Ba3.gydF4y2Ba).值得注意的是,一些抗体表现出与D12相似的HDX保护特征(补充图)。gydF4y2Ba3gydF4y2Ba),而以F07为例的其他基因则在183-192 aa和206-215 aa区域表现出了深刻的HDX保护(图7)。gydF4y2Ba2gydF4y2Ba,补充图。gydF4y2Ba3 bgydF4y2Ba).135-141 aa、183-192 aa和206-215 aa在空间上相互接近(图2)。gydF4y2Ba2gydF4y2Ba),所有抗体都表现出最强的HDX保护在206-215 aa(图。gydF4y2Ba2 a, bgydF4y2Ba;补充图。gydF4y2Ba3.gydF4y2Ba),因此HDX-MS结果表明包含206-215 aa的NTD环路可能在参与所有NTD抗体中起决定性作用。这些抗体分为两个亚群,分别以F07和D12为代表,它们可能从不同的角度接近206-215 aa,从而在135-141 aa和183-192 aa等外周区域产生不同的保护谱。与HDX-MS结果一致,在206-215 aa内的单残基ala -突变导致抗体结合降低96%,而四突变体(NTDgydF4y2BaT210A / L211A N213A / V214AgydF4y2Ba)几乎不再绑定D12或F07(补充表gydF4y2Ba1gydF4y2Ba).因此,与竞争性SPR的结果一致,HDX-MS数据也表明,8个NTD定向fab的表位高度相似,并代表229E NTD中的一个“抗原超位点”(补充图)。gydF4y2Ba4 bgydF4y2Ba,特写)。gydF4y2Ba
一个gydF4y2Ba抗体依赖性的酰胺氢保护显示了D12和F07在229E NTD上潜在的相互作用区域。229E NTD段在所有测量的时间点上,在Fab D12或F07存在的情况下,HDX显著下降,在NTD的卡通表示上突出显示并标记。在不存在(黑色)和存在Fab D12(深绿色)或F07(蓝色)的情况下,这些片段的氘吸收图以对数刻度上的氘吸收百分比与时间的关系绘制,以说明它们的HDX动力学。gydF4y2BabgydF4y2Ba氘摄取图显示,在所有其他229E NTD定向抗体存在的情况下,NTD肽202-215 aa和206-216 aa在10秒和10,000秒的HDX显著降低,这表明这些抗体也针对229E NTD的这一区域。gydF4y2BacgydF4y2Ba229E和NL63 NTDs的序列比对。229E NTD的抗原超位点用绿色矩形标记。gydF4y2BadgydF4y2Ba平衡分析图描述了固定化NL63 S蛋白与Fabs D12(左)和F07(右)之间的相互作用。KgydF4y2BaDgydF4y2Ba数值以单个数据点和平均值±SD (gydF4y2BangydF4y2Ba= 3个生物独立实验)。Mean±SD表示为gydF4y2Ba 各个数据点用开放的绿色(D12)或蓝色(F07)圈表示。gydF4y2BaegydF4y2Bafabd12结合229E S三聚体的单rbd -up配置模型。Fab D12被描绘成丝带(用不同深浅的蓝色表示轻链和重链)。229E S三聚体以灰色表面表示,其“向上”构型的RBD为粉红色,亚基内NTD为小麦色。NTD区域206-215 aa为深绿色,以突出D12、F07和所有其他NTD靶向抗体的一致表位。gydF4y2BafgydF4y2Ba在IgG中预结合D12而不是Fab格式干扰hAPN与229E S三聚体的结合。将固定化的229E S三聚体以Fab或IgG形式用D12饱和后注射重组hAPN蛋白(注射点为箭头)。所有的SPR实验都独立进行了至少两次,并显示了一个实验的代表性剖面。gydF4y2Ba
各个数据点用开放的绿色(D12)或蓝色(F07)圈表示。gydF4y2BaegydF4y2Bafabd12结合229E S三聚体的单rbd -up配置模型。Fab D12被描绘成丝带(用不同深浅的蓝色表示轻链和重链)。229E S三聚体以灰色表面表示,其“向上”构型的RBD为粉红色,亚基内NTD为小麦色。NTD区域206-215 aa为深绿色,以突出D12、F07和所有其他NTD靶向抗体的一致表位。gydF4y2BafgydF4y2Ba在IgG中预结合D12而不是Fab格式干扰hAPN与229E S三聚体的结合。将固定化的229E S三聚体以Fab或IgG形式用D12饱和后注射重组hAPN蛋白(注射点为箭头)。所有的SPR实验都独立进行了至少两次,并显示了一个实验的代表性剖面。gydF4y2Ba
接下来,我们询问这个抗原超位点206-215 aa是否保存在另一种α-HCoV NL63中。值得注意的是,对NL63 NTD上B细胞表位的策划预测表明在同一位置存在一个强B细胞表位(补充图)。gydF4y2Ba4 dgydF4y2Ba,最右面板,393-397 aa)。此外,该NTD表位在各种α- hcov分离株之间显示出约80%的序列相似性(图2)。gydF4y2Ba2摄氏度gydF4y2Ba、框区和补充图。gydF4y2Ba4 egydF4y2Ba).事实上,Fabs D12和F07可以与NL63 S蛋白发生交叉反应(图6)。gydF4y2Ba二维gydF4y2Ba和补充图。gydF4y2Ba4 fgydF4y2Ba)(尽管微弱),很可能是由于它们在NL63 NTD中识别了相似的区域(图。gydF4y2Ba2摄氏度gydF4y2Ba,方框区域)。上述结果共同表明,与之前在β- hcov中的观察结果类似,α- hcov的NTDs中也存在“抗原超位点”,尽管这种“抗原超位点”在α- hcov和β- hcov之间的位置是不同的。gydF4y2Ba4 dgydF4y2Ba).gydF4y2Ba
与β- hcov中向外的抗原性supersite不同,NTDs的抗原性是向外的。gydF4y2Ba4gydF4y2Ba)gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,在229E的NTD中发现的抗原超位点似乎部分埋藏在229E S三聚体的全rbd -down构象中。gydF4y2Ba4 bgydF4y2Ba),尽管当亚基内RBD处于“向上”构象时,它将完全可访问(补充图。gydF4y2Ba4摄氏度gydF4y2Ba).然后,我们使用NTD的206-215 aa作为接口约束,将D12停靠在229E S三聚体的one-RBD-up模型上(图2)。gydF4y2Ba2 egydF4y2Ba).基于对接模型,以IgG形式结合D12会阻碍229E RBD与其宿主受体人氨基肽酶N (hAPN)的相互作用。gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba.事实上,我们发现229E S与IgG D12的预孵育减少了hAPN的结合(图2)。gydF4y2Ba2 fgydF4y2Ba), IgG D12可抑制IC对229E伪病毒(PsV)的进入gydF4y2Ba50gydF4y2Ba浓度为86 μg/mL(图;gydF4y2Ba1 egydF4y2Ba).同样,IgG F07也抑制了IC对229E PsV的进入gydF4y2Ba50gydF4y2Ba浓度为115 μg/mL(图;gydF4y2Ba1 egydF4y2Ba).gydF4y2Ba
总的来说,这些结果表明所有229E NTD定向抗体识别229E NTD相似的中和表位,范围为206-215 aa。鉴于该表位在各种α- hcov分离株中的高度序列保守性(补充图)。gydF4y2Ba4 egydF4y2Ba),它甚至可能是所有α- hcov ntd的抗原易感性热点。gydF4y2Ba
首个α-HCoV s2定向抗体F12的表位和中和效力gydF4y2Ba
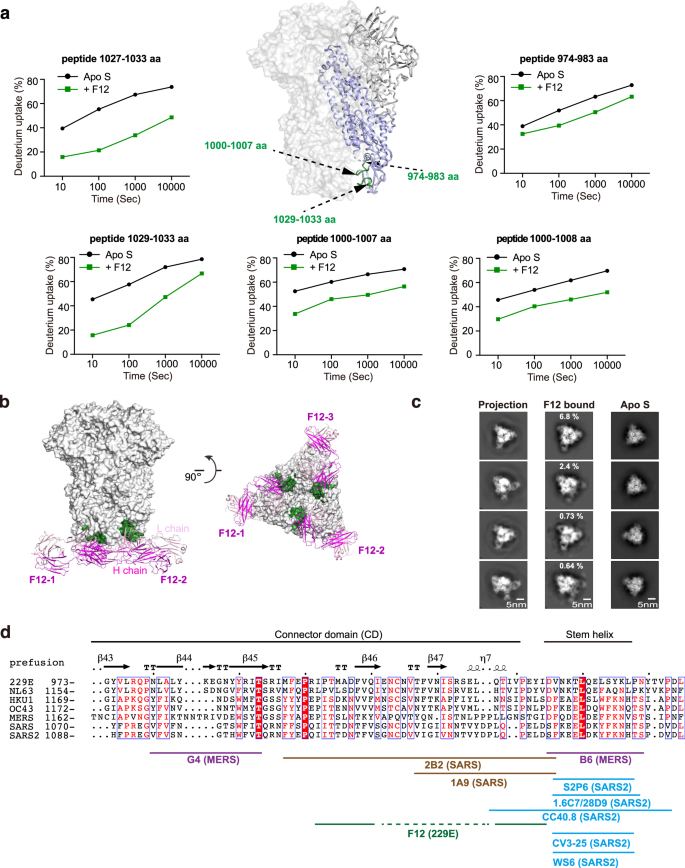
S2亚基比S1亚基更有可能作为泛hcov干预靶点,这激发了人们寻找S2定向抗体的热情gydF4y2Ba46gydF4y2Ba,gydF4y2Ba47gydF4y2Ba.虽然有少量针对SARS-CoV、SARS-CoV-2和MERS-CoV的s2定向抗体报道,但在本研究鉴定F12之前,尚未报道针对α- hcov的此类抗体gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba(无花果。gydF4y2Ba1 bgydF4y2Ba).因此,我们继续在完整的229E S三聚体上精细定位F12表位,并表征其在属内的宽度。在F12存在的情况下,229E S2中974-983 aa、1000-1007 aa、1000-1008 aa、1027-1033 aa和1029-1033 aa肽的HDX下降。gydF4y2Ba3gydF4y2Ba,补充图。gydF4y2Ba5gydF4y2Ba,gydF4y2Ba7 bgydF4y2Ba),表明这些区域的残基有很大概率直接或间接与F12相互作用。HDX的保护作用在1027-1033 aa范围内最为显著,其次是1000-1007 aa,在974-983 aa范围内中等(图2)。gydF4y2Ba3gydF4y2Ba;补充图。gydF4y2Ba5gydF4y2Ba).这样的观察结果,加上1000-1007 aa和1029-1033 aa在构象上是相邻的。gydF4y2Ba3gydF4y2Ba),提示这两个区域可能构成了抗体F12的真实表位,而空间距离较远的974-983 aa的HDX下降可能源于间接的稳定作用。与此观点一致的是,1000-1007 aa或1029-1033 aa的单一残基突变完全阻止了F12的结合(补充表gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba在229E S2中,f12依赖性的酰胺氢保护作用显示出其潜在的相互作用区域。在229E S三聚体(浅灰色表面)的背景下,在Fab F12存在的所有测量时间点上,一个229E S原聚体(S2用紫色标出)的S2段在229E S三聚体的动画表示中以深绿色突出显示。在没有Fab F12时(黑色)和有Fab F12时(深绿色),这些片段的氘吸收图以对数刻度上的氘吸收百分比与时间的关系绘制,以说明它们的HDX动力学。gydF4y2BabgydF4y2BaFab F12在全rbd -down状态下与229E S三聚体结合的模型。Fab F12被描绘成缎带(品红深浅不同的轻链和重链)。229E S三聚体的灰色表面以深绿色突出显示为HDX-MS结果和诱变验证双重定义的真实F12表位。gydF4y2BacgydF4y2Ba为了检验低温em 2D类平均值与f12结合的229E S三聚体模型之间的对应关系,将模型的代表性2D投影(左侧面板)与f12结合的229E S三聚体(中间面板)或单独229E S三聚体(右侧面板)的无参考平均值进行比较。gydF4y2BadgydF4y2Baα-和β- hcov S2亚基内CD和茎螺旋区的序列比对。F12的结构表位和其他先前报道的s2定向抗体的表位在对齐下方用实线表示。gydF4y2Ba
利用这些发现作为约束,我们将F12对接到229E S三聚体的预融合构象上(图2)。gydF4y2Ba3 bgydF4y2Ba)gydF4y2Ba10gydF4y2Ba.为了进一步确认对接结果,我们还收集了f12结合229E S三聚体的冷冻- em数据。尽管由于强烈的方向偏好,我们没有获得该复合体的高分辨率模型,但我们能够获得一些视图的高质量无参考类平均(图。gydF4y2Ba3 cgydF4y2Ba).对比F12结合和F12结合的229E S三聚体的无参考类平均,可以清楚地看到F12的化学计量结合(图12)。gydF4y2Ba3 cgydF4y2Ba,比较右边两个面板)。此外,f12结合的229E S三聚体的无参考类平均值与对接模型的2D投影匹配得非常好(图2)。gydF4y2Ba3 cgydF4y2Ba,对比左侧两面板),进一步验证了对接模型。接下来,我们测试了F12的中和效力。IgG F12可抑制IC进入229E型PsVgydF4y2Ba50gydF4y2Ba101 μg/mL(图;gydF4y2Ba1 egydF4y2Ba),可能是通过启动S蛋白的过早构象变化或通过阻断6-HB的形成,就像其他s2结合nab一样gydF4y2Ba35gydF4y2Ba,gydF4y2Ba38gydF4y2Ba.综上所述,这些结果表明F12识别229E S2亚基连接域(CD)中的一个结构表位,并确实中和(图2)。gydF4y2Ba3 dgydF4y2Ba).gydF4y2Ba
在此之前,已经报道了一些s2导向的nabgydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba.在针对MERS-CoV S2的两个nab中,G4与CD内的一个可变糖基化环结合,缺乏交叉反应性,而B6识别相对保守的茎螺旋区域,紧随着HR2,并已发现交叉中和MERS-CoV和OC43(图2)。gydF4y2Ba3 dgydF4y2Ba)gydF4y2Ba35gydF4y2Ba,gydF4y2Ba38gydF4y2Ba.同时,2B2和1A9在SARS-CoV的CD中识别相似的区域,后者对SARS-CoV-2的交叉反应有限(图2)。gydF4y2Ba3 dgydF4y2Ba)gydF4y2Ba37gydF4y2Ba,gydF4y2Ba48gydF4y2Ba.对于SARS-CoV-2,所有s2导向的NAbs的表位都聚集在茎螺旋区,其中一个NAbs S2P6被发现对几乎所有β- hcov具有广泛的中和活性gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba(无花果。gydF4y2Ba3 dgydF4y2Ba).综上所述,这些发现表明S2内的茎螺旋区似乎是一个比CD区更具有泛hcov潜力的表位。为了进一步支持这一观点,我们发现CD-targeting F12即使与同一属的NL63也缺乏交叉反应性(补充图。gydF4y2Ba5 bgydF4y2Ba).gydF4y2Ba
229E RBD远端中和表位gydF4y2Ba
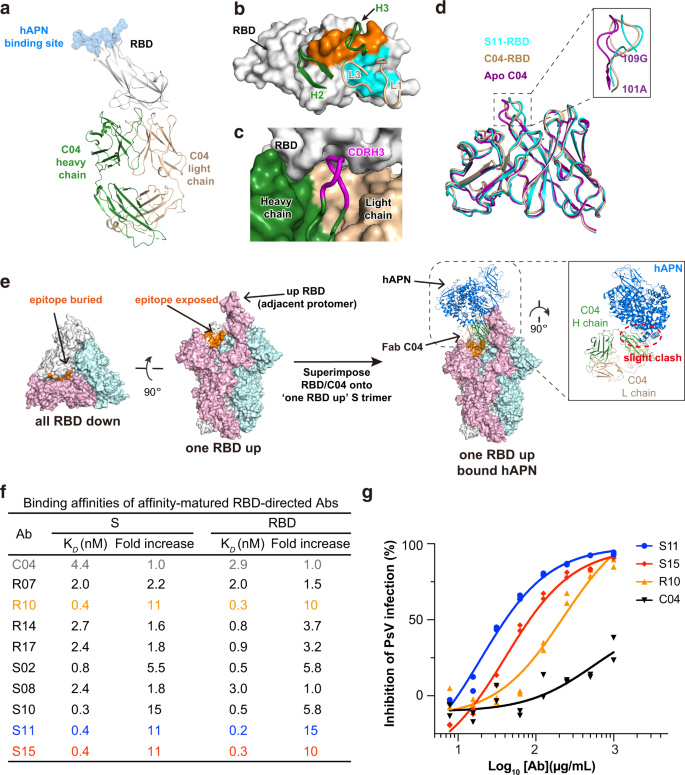
在SARS-CoV-2病例中,值得注意的是,最有效的nab通常识别RBD中的表位,其次是针对其NTD和S2亚基的抗体gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba.与这一观察结果相反,在所有针对229E S的抗体中,rbd导向的C04似乎是最不中和的gydF4y2Ba50gydF4y2Ba> 500 μg/mLgydF4y2Ba1 egydF4y2Ba).为了了解其低中和活性是否与其作用方式相关,我们随后测定了Fab C04单独和C04与229E RBD配合物的晶体结构(图2)。gydF4y2Ba4gydF4y2Ba,补充图。gydF4y2Ba6gydF4y2Ba和表gydF4y2Ba1gydF4y2Ba).C04埋525 ÅgydF4y2Ba2gydF4y2BaRBD上的表面积及其CDRL1和CDRL3,以及462 ÅgydF4y2Ba2gydF4y2Ba其CDRH2和CDRH3(图;gydF4y2Ba4 bgydF4y2Ba).在这些CDR环中,CDRH3是特别有趣的,因为它在CDRH3环中包含多达5个Gly残基。gydF4y2Ba1 dgydF4y2Ba),这可能赋予了它很大的结构可塑性,可以整齐地放入RBD表面的一个口袋中(图。gydF4y2Ba4摄氏度gydF4y2Ba),主要通过主干驱动的交互作用(补充图。gydF4y2Ba6克gydF4y2Ba).CDRH3结合RBD时的巨大构象变化也很好地证明了它的结构可塑性(图。gydF4y2Ba4 dgydF4y2Ba).RBD结合后CDRH3的RMSD达到1.51 Å,而CDRL1、CDRL3和CDRH2的平均RMSD仅为0.66 Å。gydF4y2Ba
一个gydF4y2BaFab C04的晶体结构与229E RBD的卡通表示。C04的重链和轻链分别为深绿色和小麦色,而229E RBD为灰色。宿主受体hAPN的结合位点用蓝色球体描绘。gydF4y2BabgydF4y2Ba接触残基在229E RBD的表面表示为橙色(重链)和蓝色(轻链)。CDR环路参与229E RBD显示和标记。gydF4y2BacgydF4y2BaC04的重链(深绿色)和轻链(小麦)和RBD(灰色)作为表面表示。CDRH3(洋红色)在漫画中描绘,以说明其整齐地安装到229E RBD的表面口袋。gydF4y2BadgydF4y2Ba游离C04(紫色)、RBD结合C04(小麦)和RBD结合S11(蓝色)的结构叠加说明了CDRH3结合RBD后的构象变化。所有结构均以卡通形式显示,插图为CDRH3区域的特写视图。gydF4y2BaegydF4y2Ba左图:C04表位以全rbd -down构象隐藏,以one-RBD-up构象完全暴露。右图:Fab C04的结合会轻微干扰hAPN与相邻的“向上”原聚体的结合(粉红色,逆时针方向)。通过将RBD-C04的结构叠加到一个rbd -up的229E S三聚体上,模拟了Fab c04结合229E S三聚体的结构。接下来,该结构与基于229E RBD的“向上”位置的hpn -229E RBD复合物对齐。gydF4y2BafgydF4y2Ba亲本C04与抗229E S三聚体或RBD的亲和成熟抗体的结合亲和力通过SPR来表征。在亲和成熟抗体中,抗体S11(蓝色)和S15(红色)对229E S三聚体和RBD的亲和增加了11倍。gydF4y2BaggydF4y2Ba将IgG S11(蓝色)、S15(红色)和R10(橙色)的中和谱与亲本IgG C04(黑色)对229E PsV的中和谱进行比较。单个数据点分别显示为填充的蓝色圆圈(S11)、红色菱形(S15)、橙色三角形(R10)和黑色三角形(C04)。gydF4y2Ba
值得注意的是,C04的表位位于229E RBD上hAPN受体结合位点的远端(图2)。gydF4y2Ba4gydF4y2Ba),与包括CR3022、EY6A、S304和553-49在内的几种SARS-CoV-2 nab的结合模式有些相似,它们也靶向SARS-CoV-2 RBD远端的“隐性”表位gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba.与这些SARS-CoV-2 nab相似,C04的表位也隐藏在229E S三聚体的全RBD-down构象中,但当其逆时针相邻的RBD处于“向上”位置时,C04的表位就可以完全接近(图2)。gydF4y2Ba4 egydF4y2Ba,左面板)。C04- 229e RBD配合物与one-RBD-up S三聚体的结构对齐进一步表明,Fab C04的结合会轻微干扰hAPN与邻近的“up”原聚体的结合(图4)。gydF4y2Ba4 egydF4y2Ba,右侧面板)。同样,229E S与C04以Fab或IgG形式预孵育确实减少了hAPN的结合(补充图。gydF4y2Ba6 bgydF4y2Ba),尽管在一定程度上低于IgG D12预孵育诱导的(图。gydF4y2Ba2 fgydF4y2Ba).值得注意的是,CR3022或553-49的结合已被证明可诱导SARS-CoV-2 S蛋白的破坏gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba.由于C04也靶向229E S蛋白的“隐层”表面,我们想知道C04的结合是否会诱导229E S的分解,从而在不存在C04或存在C04的情况下,在EM阴性染色下观察229E S(补充图)。gydF4y2Ba6摄氏度gydF4y2Ba).在C04存在的情况下,229E S三聚体的很大一部分分解为C04结合的229E S原聚体(补充图)。gydF4y2Ba6摄氏度gydF4y2Ba(右)孵育1小时后,大多数229E S三聚体在没有C04的情况下保持完整(补充图。gydF4y2Ba6摄氏度gydF4y2Ba(左)。因此,C04除了通过空间位阻作用阻断APN的结合外,还可能通过诱导229E S蛋白的分解而起到中和作用。gydF4y2Ba
C04表位的位置解释了其相对较低的中和活性。在CR3022等远端结合抗体的情况下,中和活性与其对SARS-CoV-2 RBD的亲和力高度相关gydF4y2Ba56gydF4y2Ba.考虑到C04是从naïve合成库中选择的,显然缺乏通过体细胞超突变来提高亲和力的机会。为了增强C04的中和效力,我们使用基于亲本克隆的软随机库进行了体外亲和成熟,并获得了一个阳性克隆子集,这些阳性克隆对229E RBD和S三聚体都表现出更高的亲和性(图2)。gydF4y2Ba4 fgydF4y2Ba,补充图。gydF4y2Ba6 d, egydF4y2Ba).在这些亲和成熟抗体中,抗体S11和S15对229E RBD和S三聚体的亲和度增加了11倍(图2)。gydF4y2Ba4 fgydF4y2Ba),很大程度上是因为它们与目标的分离要慢得多(补充图。gydF4y2Ba6 d, egydF4y2Ba).因此,与IC相比,IgG C04和IgG S11和S15对229E PsV感染的中和活性明显增强gydF4y2Ba50gydF4y2Ba分别为19 μg/mL和40 μg/mL(图;gydF4y2Ba4 ggydF4y2Ba).我们还进一步对S11- rbd复合物的结构进行了表征,发现S11的结合模式与C04相同(Supplementary Fig。gydF4y2Ba6 fgydF4y2Ba),整体RMSD为0.69 Å。S11和C04的主要区别在于它们独特的富g CDRH3环,其中CDRH3残基为AgydF4y2Ba109gydF4y2Ba年代gydF4y2Ba110gydF4y2BaVgydF4y2Ba111gydF4y2Ba成熟成IgydF4y2Ba109gydF4y2BaTgydF4y2Ba110gydF4y2BaFgydF4y2Ba111gydF4y2Ba在S11中,从而与RBD的P393和W419形成额外的侧链到侧链疏水填料(补充图)。gydF4y2Ba6克gydF4y2Ba).因此,类似于SARS-CoV-2 RBD远端的“隐性”表位,一个中和表位也位于229E RBD远端。gydF4y2Ba
讨论gydF4y2Ba
迄今为止,对α-HCoVs S蛋白的抗原性认识尚不清楚。本研究以229E为代表的α-HCoV,对其S蛋白进行了抗原性定位。由于我们无法获得α-HCoVs感染个体的恢复期血清样本,我们选择利用高度验证的合成Fab库来选择识别三聚体229E S的抗体(图2)。gydF4y2Ba1gydF4y2Ba).在鉴定的10个nab中,有8个识别NTD,这表明229E的NTD具有抗原显性(图2)。gydF4y2Ba1 bgydF4y2Ba).这一观察结果与以往对SARS-CoV、MERS-CoV和SARS-CoV- 2s蛋白的研究形成了鲜明对比,在这些研究中,它们的rbd蛋白而不是ntd蛋白最常被nab靶向gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba.Shi最近的发现也是如此gydF4y2Ba等gydF4y2Ba.还发现229E RBD在小鼠血清中诱导中和效力的能力明显低于完整的229E S三聚体或229E S1gydF4y2Ba57gydF4y2Ba.gydF4y2Ba
我们还结合HDX-MS、cryo-EM和晶体学对所选NAbs的结构表位进行了描述。在本研究之前,仅发现了少数针对三种高致病性β- hcov的s2定向抗体gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,其目标是位于HR2基序上游的CD或茎螺旋区域(图2)。gydF4y2Ba3 dgydF4y2Ba).本研究中的抗体F12是第一个针对α-HCoV S2亚基识别的NAb,它可以识别CD中的结构表位(图2)。gydF4y2Ba3.gydF4y2Ba).值得注意的是,F12甚至与NL63缺乏交叉反应性,NL63是同一属中229E的近邻(补充图)。gydF4y2Ba5 bgydF4y2Ba).同样,针对MERS-CoV CD的G4抗体也缺乏交叉反应性,而SARS-CoV CD靶向抗体2B2和1A9仅表现出对SARS-CoV-2的有限交叉反应性gydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba48gydF4y2Ba.相比之下,分别针对MERS-CoV和SARS-CoV-2茎螺旋区域的抗体B6和S2P6似乎具有更广泛的交叉反应性。B6可以交叉中和MERS-CoV和OC43这两种来自不同亚属的β- hcov,而S2P6对几乎所有的β- hcov都具有广泛的中和活性gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba.总的来说,这些发现表明,如果要诱导广泛有效的抗体,S2内的茎螺旋区域似乎是比CD更好的靶点。在这项工作提交期间,三个独立的研究已经确定FP是S2亚基内另一个潜在的泛hcov干预靶点gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba.然而,值得注意的是,茎螺旋区或FP表位部分埋藏在S三聚体的预融合状态,因此可能不易被宿主免疫识别所接近gydF4y2Ba36gydF4y2Ba,gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba.茎螺旋区较低的抗原性还体现在我们在本研究中没有获得任何针对229E S这一区域的抗体,而cd结合F12被鉴定出来(图2)。gydF4y2Ba3.gydF4y2Ba).因此,为了充分探索茎螺旋或FP表位在广泛有效疫苗设计中的潜力,免疫聚焦方法有必要将宿主反应集中在这两个表位上,例如掩盖附近不太广泛但更具有抗原性的表位(例如CD表位),并通过结构引导的免疫原呈现增强它们的可及性gydF4y2Ba61gydF4y2Ba.gydF4y2Ba
除了S2内的中和表位外,中和表位也在229E RBD上hAPN结合位点远端被捕获(图2)。gydF4y2Ba4 gydF4y2Ba),这让人联想到SARS-CoV-2 RBD远端的nab CR3022、EY6A、S304和553-49所识别的隐性表位gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba51gydF4y2Ba,gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba.值得注意的是,结合CR3022或553-49可诱导SARS-CoV-2 S蛋白的分解gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba.类似地,C04与229E RBD中的“隐表位”结合也诱导229E S三聚体的破坏,这表明诱导S三聚体的分解可能是nab靶向cov RBD远端“隐表位”的常见机制。在229E的NTD中,由206-215 aa组成的区域被所有8种229E NTD定向抗体识别(图2)。gydF4y2Ba1gydF4y2Bac, d,gydF4y2Ba2 a, bgydF4y2Ba和补充图。gydF4y2Ba3.gydF4y2Ba),表明该区域具有较高的抗原性,并提醒我们之前报道的SARS-CoV-2和OC43 NTDs中的“抗原超位点”(补充图。gydF4y2Ba4gydF4y2Ba)gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.值得注意的是,这个229E NTD“抗原超位点”似乎在α-HCoV中保守。gydF4y2Ba2摄氏度gydF4y2Ba和补充图。gydF4y2Ba4 egydF4y2Ba),两种针对该超位点的229E抗体与NL63发生交叉反应(图3)。gydF4y2Ba二维gydF4y2Ba和补充图。gydF4y2Ba4 fgydF4y2Ba),另一种α-HCoV。综上所述,这些事实表明该NTD超位点有潜力作为α- hcov中广泛有效的表位。gydF4y2Ba
综上所述,我们以229E为代表的α-HCoV,并确定了其S蛋白的抗原格局。虽然这些发现突出了S蛋白在属间的抗原差异以及泛hcov疫苗设计面临的挑战,但它们也揭示了一些共同的特征,这可能为开发针对至少一部分hcov的广泛有效疫苗提供了信息。我们预计,我们的工作将作为一个起点,全面了解所有流行的hcov之间的异同,gydF4y2BaespgydF4y2Ba.α- hcov和β- hcov之间的差异,这将为开发有效的预防措施提供有用的信息,以便更好地为未来的' CoV-X '暴发做好准备。gydF4y2Ba
方法gydF4y2Ba
基因克隆、蛋白表达及纯化gydF4y2Ba
229E基因S外结构域(17-1115 aa)、S1 (17-560 aa)、S2 (575-1115 aa)、NTD (17-265 aa)和RBD (294-435 aa) (Uniprot ID: P15423)和NL63基因S外结构域(16-1291 aa) (Uniprot ID: Q6Q1S2)分别克隆到修饰的pFastBac1载体(Invitrogen)上,n端gp67信号肽和c端T4三聚体基序,TEV裂解位点和6 × His标记。229E S蛋白和NTD的突变体通过定点诱变构建,并通过DNA测序验证。野生型蛋白和突变体的表达和纯化使用如下所述的方法。gydF4y2Ba
229E S外结构域的表达使用Bac-to-Bac系统(Invitrogen),按照制造商的协议进行。简单地说,杆状病毒是通过FuGENE HD (Promega)将新鲜制备的bacmid转染到Sf9细胞中制备的,并用于感染MOI为5至10的悬浮High Five细胞。72小时后收集感染的High Five细胞的培养基上清液,补充1 mM PMSF,然后加载到Excel Ni-NTA柱上(GE Healthcare)。然后将含有229E S三聚体的洗脱组分混合,并在Superpose 6 10/300 GL柱(GE Healthcare)上用20 mM Tris 8.0, 150 mM NaCl平衡,通过两次连续凝胶过滤层析进一步纯化。然后将含有229E S三聚体的凝胶过滤峰馏分浓缩并储存在−80°C下,直到进一步使用。用同样的方法制备NL63的S外畴。229E的S1, S2, NTD和RBD以类似的方式表达和纯化,除了其凝胶过滤步骤在Superdex 200 10/300色谱柱上进行(GE Healthcare)。gydF4y2Ba
将hAPN (Uniprot ID: P15144)的可溶性外膜(66-967 aa)克隆到pACEBac1载体中进行昆虫细胞表达。添加n端蜜蜂蜂毒素(HBM)信号肽和c端6 × His标签,促进蛋白表达和纯化。hAPN蛋白的纯化方法与上述方法相似。纯化的hAPN浓缩并储存在含有20 mM Tris 8.0, 150 mM NaCl, 5%甘油的缓冲液中,在−80°C下,直到进一步使用。gydF4y2Ba
为了表达Fabs,以噬菌体DNA为模板,通过PCR扩增出编码重链和轻链序列的DNA,亚克隆到修饰后的pRH2.2表达载体上,该载体n端含有一个23残基信号肽(MKKNIAFLLASMFVFSIATNAYA), c端含有6 × His标签。Fab结构然后被转换成gydF4y2Ba大肠杆菌gydF4y2BaBL21 (DE3)蛋白表达。简单地说,细胞在37℃的Luria-Bertani (LB)培养基中培养,直到OD600达到0.8。培养物在0.5 mM异丙基硫代- β - d -半乳糖苷(IPTG)存在下,在20°C下孵育16小时,然后在5000℃离心收集gydF4y2BaggydF4y2Ba5分钟。然后将细胞球重新悬浮在pH 7.4的PBS缓冲液中,并使用NANO均质机(ATS Engineering Limited)在800 bar, 4°C下裂解。离心后,用Talon金属亲和树脂(Clontech)混合上清液,用5-500 mM咪唑线性梯度洗脱。然后在HiTrap Protein A HP柱(GE Healthcare)上进一步纯化洗脱馏分,然后装入Superdex 200 10/300 GL凝胶过滤柱(GE Healthcare)。将含有目标抗体的凝胶过滤峰组分合并、浓缩,并在−80°C保存以供进一步使用。gydF4y2Ba
对于全长IgG1格式的抗体,重链和轻链序列分别克隆到pFuse-hIgG1-Fc2和pFuse2ss-CLIg-hK表达载体(InvivoGen)中。重链质粒和轻链质粒以1:1摩尔比共转染HEK293F细胞表达IgGs。转染后的细胞再培养6天,然后采集。用HiTrap Protein A HP柱(GE Healthcare)捕获培养基中的抗体,并通过尺寸排除层析进一步纯化。纯化的抗体浓缩并储存在−80°C的PBS缓冲液(pH 7.4)中。gydF4y2Ba
抗229E S三聚体抗体的选择和亲和成熟gydF4y2Ba
使用经过验证的噬菌体展示,合成的抗原结合片段(Fab)库进行噬菌体选择,其多样性为3 × 10gydF4y2Ba10gydF4y2Ba唯一克隆,如以下修改所述gydF4y2Ba40gydF4y2Ba.简而言之,用5 μg/mL的neutravidin (Manufacturer) PBS溶液在4℃下在96孔捕获板(ThermoFisher Scientific)的孔上涂一夜,然后用0.2% BSA溶液堵塞1小时,然后用0.05% Tween (PT)缓冲液PBS洗涤4次。同时,将噬菌体文库与生物素化的50 nM 229E S三聚体孵育2小时,然后转移到neutravidin涂层的捕获板上。15分钟孵育后,用PT缓冲液冲洗8次,噬菌体克隆如先前报道的那样被洗脱和扩增gydF4y2Ba40gydF4y2Ba.ELISA检测与229E S蛋白结合而不与BSA对照蛋白结合的克隆的可变区gydF4y2Ba62gydF4y2Ba,进行PCR扩增并进行DNA测序以解码抗体可变区。gydF4y2Ba
对于C04的亲和成熟,通过在cdr中引入停止位点,并通过Kunkel方法使用诱变寡核苷酸进行随机化,从而生成库模板gydF4y2Ba63gydF4y2Ba.模板DNA的cdr通过寡核苷酸定向诱变软随机化,使用70:10:10:10的核苷酸混合物,使用相同的方法偏向于C04亲本序列。然后将突变的共价闭合环状DNA电穿孔成电活性DNAgydF4y2Ba大肠杆菌gydF4y2BaSS320细胞和噬菌体扩增如先前报道gydF4y2Ba64gydF4y2Ba生成成熟库。按照上述方法进行筛选,在随后的每一轮筛选中,生物素化RBD的浓度都在降低。gydF4y2Ba
229E假病毒(PsV)gydF4y2Ba
冠状病毒PsV如先前报道的那样产生gydF4y2Ba65gydF4y2Ba.简单地说,HEK293T细胞共转染了质粒pcDNA3.1-229E-spike和HIV骨干质粒pNL4.3-HIV-luc。48小时后,收集含有229E PsV的细胞上清液,并保存在−80°C中。gydF4y2Ba
抗体抗229e PsV活性评价gydF4y2Ba
抗体抗229e PsV活性的测定与先前报道的相同gydF4y2Ba66gydF4y2Ba.简单地说,抗体首先被连续稀释,并与229E PsV共孵育30分钟。接下来,将含有229E PsV和稀释抗体的混合物添加到96孔板中的Caco2细胞中。12 h后加入含2% FBS的新鲜DMEM。另48 h后测定荧光素酶活性和ICgydF4y2Ba50gydF4y2Ba计算数值。gydF4y2Ba
表面等离子体共振(SPR)gydF4y2Ba
SPR实验使用Biacore 8 K (GE Healthcare)进行。所有实验均在25°C下使用含有10 mM HEPES pH 7.4、150 mM NaCl、3 mM EDTA和0.01% v/v Tween-20的运行缓冲液进行。对于大多数抗体的亲和力测定,重组229E S蛋白、S1、RBD、NTD和NL63 S蛋白分别被固定在CM5传感器芯片上的单个流细胞上(GE Healthcare)。将3个只含有运行缓冲液的样品注射到样品和参比流式细胞上,然后将纯化抗体连续稀释2倍(30 μL/min,结合240 s,解离540 s)。为了测量Fab F12对229E S2或nl63s蛋白的结合亲和力,将重组229E S2或nl63s的连续稀释液流过固定化的Fab F12。所有绑定数据采用空白循环和参考流单元减法双引用。使用Biacore Insight评估软件(GE医疗保健)将所得的传感器图拟合为1:1的Langmuir绑定模型。gydF4y2Ba
对于抗体对的竞争性结合分析,S1蛋白如上所述固定在CM5传感器芯片上。然后,使用双重注射功能对不同的抗体进行配对测试。简单地说,将一抗以20 μL/min的速度流过固定化S1蛋白表面400 s以达到饱和,然后第二次注射含有一抗和二抗对的混合物。响应单元采用与上述相同的软件进行分析。gydF4y2Ba
氢-氘交换质谱(HDX-MS)gydF4y2Ba
如先前报道的那样,229E S或229E S抗体复合物的酰胺氢交换是手动进行的gydF4y2Ba67gydF4y2Ba.简单地说,229E S三聚体的酰胺氢交换是通过将2 μL蛋白质样品在30 μM下稀释成18 μL D开始的gydF4y2Ba2gydF4y2BaO缓冲液(20 mM Tris, pD 8.0, 150 mM NaCl)在20°C。在不同的时间点(0 s、10 s、100 s、1000 s和10000 s),将冷却过的淬火缓冲液加入最终浓度为1% FA、pH 3.0、1.6 M尿素、100 mM TCEP中淬火,并立即在液氮中冷冻。对于有Fab F12存在的S蛋白的HDX-MS, 6倍摩尔过量的Fab F12与229E S三聚体在冰上孵育60分钟。2 μL混合后标记,淬火,闪冻。所有冷冻样品保存在−80°C,直到分析。对于229E NTD的HDX-MS,在没有和存在Fab D12的情况下,单独标记2 μL NTD或2 μL D12结合的NTD,按上述方法进行标记、淬火和速冻。对于其余的ntd定向抗体,除了仅测量0 s、10 s和10,000 s时间点以增加实验通量外,采用类似的方法进行标记反应。所有实验均独立重复两次。gydF4y2Ba
解冻后的样品立即注射到配备在线消化和脱盐的HPLC-MS (Agilent 1100)系统中。然后用Hypersil Gold分离脱盐的消化液gydF4y2BaTMgydF4y2Ba分析柱(ThermoFisher)超过18分钟的梯度,并直接用Orbitrap聚变质谱仪(ThermoFisher)进行分析。HPLC系统在样品之间用空白注射进行广泛清洁,以尽量减少任何残留。多肽鉴定采用串联质谱(MS/MS)法进行。所有的MS/MS谱均使用Proteome Discoverer Software (ThermoFisher)进行分析。我们使用HD-Examiner v2.3 (Sierra Analytics)对肽质心进行了初步分析,然后手动验证每个肽以检查保留时间、电荷状态、m/z范围和重叠肽的存在。229E S和NTD的肽覆盖率分别为90%和89%(补充图)。gydF4y2Ba7gydF4y2Ba).每个肽的相对氘化水平(%D)由HD-Examiner自动计算,假设完全氘化的样品在当前LC设置下保留90% D。gydF4y2Ba
b细胞表位预测gydF4y2Ba
采用BepiPred 2.0算法预测229E S蛋白和NL63 S蛋白的b细胞表位。gydF4y2Bahttps://www.iedb.org/gydF4y2Ba),截断值为0.52(对应于特异性大于0.6而灵敏度小于0.4)。根据以下标准进一步筛选预测的表位:(1)是否至少5 aa长;(2)它们是否以一种rbd -up 229E或NL63 S三聚体的形式暴露于溶剂中;(3)它们是否接近229E或nl63s三聚体上已知的屏蔽聚糖(5 Å以内)。策划的预测结果使用程序Pymol (The Pymol Molecular Graphics System, Version 2.1 Schrödinger, LLC)在相应的结构中进行标记。gydF4y2Ba
分子对接gydF4y2Ba
利用瑞士模型网站建立了F12和D12晶圆厂的同调模型gydF4y2Ba68gydF4y2Ba.在Chimera中手动调整带有一个RBD“up”的229E S三聚体模型gydF4y2Ba69gydF4y2Ba,以SARS-CoV-2 S三聚体与一个RBD“up”(PDB:6VYB)为参考。F12和D12模型分别使用PatchDock以全RBD-down构象(PDB:6U7H)连接到229E S三聚体上,或以“one RBD up”构象连接到229E S三聚体上gydF4y2Ba70gydF4y2Ba.在PatchDock模拟中,抗体和229E S三聚体分别在抗体-抗原复合模式下作为受体和配体,并将229E S上的D12或F12表位作为配体结合位点提供给PatchDock算法,HDX-MS和诱变实验双重定义229E S。对接模型用FireDock进一步细化gydF4y2Ba71gydF4y2Ba.gydF4y2Ba
结晶和x射线结构测定gydF4y2Ba
纯化后的RBD蛋白与Fab C04或Fab S11按摩尔比1:1.2混合,在4℃下孵育过夜,形成RBD/C04或RBD/S11复合物。采用悬滴蒸汽扩散法在20℃下对Fab C04、RBD/C04配合物和RBD/S11配合物进行结晶筛选。在含有15% w/v聚乙二醇3350、0.01 M六水氯化镁、0.005 M六水氯化镍和0.1 M HEPES pH 7.0的条件下,得到了衍射质量的C04晶体。在含有1.6 M硫酸铵,0.1 M临界酸pH 5.0的条件下,得到了与C04配合物的RBD晶体,而在含有3.6 M甲酸钠,10%甘油的条件下,得到了RBD/S11配合物的晶体。用20% (v/v)甘油作为冷冻保护剂收集晶体,并在液氮中快速冷却以收集数据。gydF4y2Ba
x射线衍射数据采集于上海同步辐射设备(SSRF) BL19U1束线0.978 Å波长处,使用HKL2000或Xia2-DIAL程序处理gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba.每个结构都是通过分子置换(MR)相移器求解的gydF4y2Ba74gydF4y2Ba,使用来自PDB沉积物6ATK和6DF2的MR模板。分别在COOT和PHENIX中进行迭代建模和优化gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba.利用MolProbity软件对最终模型的质量进行分析gydF4y2Ba77gydF4y2Ba.表中列出了数据收集和细化统计信息gydF4y2Ba1gydF4y2Ba.对Fab C04、RBD-C04复合体和RBD-S11复合体结构的Ramachandran统计结果表明,三种结构不存在异常值,“允许”得分分别为2.3%、4.9%和3.9%,“赞成”得分分别为97.7%、95.1%和96.1%。gydF4y2Ba
使用Pymol程序(The Pymol分子图形系统,Version 2.1 Schrödinger, LLC)来制备图形。用PDBePISA方法鉴定了表位和副位残基,以及埋没的溶剂可及表面积gydF4y2Ba78gydF4y2Ba.gydF4y2Ba
负染电子显微镜(nsEM)gydF4y2Ba
要检查的蛋白质样品被稀释到适当的浓度,然后放在一个刚刚发光放电的碳涂层网格上。用缓冲液(20 mM Tris, 150 mM NaCl, pH 8.0)冲洗两次后,用2%甲酸铀酰(pH 7.0)染色,然后加载到120 kV TEM上进行检查。为了观察C04诱导的229E S的分解,229E S蛋白在室温下孵育1小时,加入或不添加多余的C04。然后按照上述方法制备孵卵样品,并使用FEI Talos C-Twin电子显微镜在120 kV下进行观察。每个样本在73,000 ×nominal倍率(1.92 Å/pixel)下人工采集23-31张具有代表性的图像,离焦范围为−2至−3 μm。图像处理、粒子选取、无参考的二维分类均在RELION v3.1中进行gydF4y2Ba79gydF4y2Ba.使用EMAN2命令e2classsvspropr .py将229E S protomer-C04模型的投影与实验2D类平均值进行匹配gydF4y2Ba80gydF4y2Ba
低温电子显微镜数据收集和图像处理gydF4y2Ba
229E S蛋白(2.4 mg/mL)与6倍摩尔过量的Fab F12孵育60分钟,然后应用于发光放电的多孔碳栅格(Quantifoil, r2 / 1,300)。使用Vitrobot (ThermoFisher, USA)在100%湿度和8°C条件下,在力水平为−1的情况下对网格进行3秒的吸湿。被吸干的网格立即被注入液氮冷却的液态乙烷中。Cryo-EM数据使用300 kV Titan Krios G2电子显微镜(ThermoFisher, USA)和K3 Summit直接电子探测器(Gatan, USA)在超分辨率模式下收集,像素大小为0.53 Å。每部电影的曝光时间为2.8秒,剂量分为40帧,总剂量约为50 egydF4y2Ba−gydF4y2Ba/一个gydF4y2Ba2gydF4y2Ba在样本上。数据采集时使用的离焦值范围为0.8 ~ 2.5 μm。所有图像使用SerialEM自动数据收集软件包收集。电影使用MotionCor2进行对齐gydF4y2Ba81gydF4y2Ba将超分辨率电影分2个(1.06 Å /pixel),通过CTFIND4的对比度传递函数(CTF)估计离焦值gydF4y2Ba82gydF4y2Ba.使用Relion 3.1自动挑选颗粒gydF4y2Ba79gydF4y2Ba从手动选择的显微照片。使用cryoSPARC v2.5提取颗粒并进行多轮无参考2D分类gydF4y2Ba83gydF4y2Ba.定义良好的粒子图像被保留以作进一步分析。颗粒的取向偏差阻碍了三维重建。使用EMAN2命令e2classsvsproject .py将229E S trimer-F12对接模型的投影与那些定义良好的粒子的实验2D类平均值进行匹配gydF4y2Ba80gydF4y2Ba.gydF4y2Ba
序列守恒计算gydF4y2Ba
计算66个229E和70个nl63s蛋白序列在α- hcov 229E NTD抗原超位点上的序列保守性(请参见补充表)gydF4y2Ba3.gydF4y2Ba)已从Uniprot网站(gydF4y2Bahttp://www.uniprot.orggydF4y2Ba),并与Clustal Omega结盟gydF4y2Ba84gydF4y2Ba.然后利用熵网络服务器计算超位点上的氨基酸位置频率(gydF4y2Bahttps://www.hiv.lanl.gov/content/sequence/ENTROPYgydF4y2Ba),并在WebLogo图中显示gydF4y2Ba85gydF4y2Ba.利用ConSeq网络服务器计算超级遗址的位置保护得分gydF4y2Ba86gydF4y2Ba.gydF4y2Ba
统计和再现性gydF4y2Ba
在Prism 9软件(GraphPad)中使用四参数逻辑回归模型分析来自至少两个生物重复序列的假病毒中和数据。数据以单个数据点和平均值±标准差表示。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
C04, 229E RBD与C04或S11的复合体坐标已分别存入PDB,编号为7VMZ, 7VN9和7VNG。补充资料中提供了本研究的其他数据,图形和图表的源数据可在补充资料中找到gydF4y2Ba1gydF4y2Ba与本文相关。未裁剪和未编辑的凝胶图像包括在补充图中。gydF4y2Ba8gydF4y2Ba.与本文相关的所有其他数据可向通讯作者索取。gydF4y2Ba
参考文献gydF4y2Ba
崔健,李峰,史志林。病原性冠状病毒的起源与演化。gydF4y2BaNat. Rev.微生物。gydF4y2Ba17gydF4y2Ba, 181-192(2019)。gydF4y2Ba
冯涛生,刘东霞。人冠状病毒:宿主-病原体相互作用。gydF4y2Ba微生物。gydF4y2Ba73gydF4y2Ba, 529-557(2019)。gydF4y2Ba
Rucinski, s.l, Binnicker, m.j, Thomas, a.s.和Patel, R. 2014 - 2020年冠状病毒229E、HKU1、NL63和OC43的季节性。gydF4y2Ba梅奥中国。Proc。gydF4y2Ba95gydF4y2Ba, 1701-1703(2020)。gydF4y2Ba
科恩,j。梦想疫苗。gydF4y2Ba科学gydF4y2Ba372gydF4y2Ba, 227-231(2021)。gydF4y2Ba
阿蒂斯,A.等人。SARS - CoV-2和其他人类致病性冠状病毒的抗病毒药物和可药物靶点的现状。gydF4y2Ba药物抵抗。Updat。gydF4y2Ba53gydF4y2Ba, 100721(2020)。gydF4y2Ba
贾丁,J. G.等。最小变异的HIV-1广泛中和抗体指导还原论疫苗设计。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba12gydF4y2Ba, e1005815(2016)。gydF4y2Ba
哈里森,S. C.病毒膜融合。gydF4y2Ba病毒学gydF4y2Ba479 - 480gydF4y2Ba, 498-507(2015)。gydF4y2Ba
Baquero, E.等人。病毒融合糖蛋白结构转变过程中的中间构象。gydF4y2Ba咕咕叫。当今。性研究。gydF4y2Ba3.gydF4y2Ba, 143-150(2013)。gydF4y2Ba
沃尔斯,a.c.等。低温电子显微镜观察到冠状病毒刺突蛋白的聚糖屏蔽和表位掩蔽。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba23gydF4y2Ba, 899-905(2016)。gydF4y2Ba
李,Z.等。人冠状病毒HCoV-229E s蛋白结构和受体结合。gydF4y2BaElifegydF4y2Ba8gydF4y2Ba,gydF4y2Bahttps://doi.org/10.7554/eLife.51230gydF4y2Ba(2019)。gydF4y2Ba
宋,x等。HCoV-229E刺突糖蛋白的冷冻电镜分析显示了动态的融合前构象变化。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 141(2021)。gydF4y2Ba
Gui, M.等。SARS-CoV刺突糖蛋白的冷冻电子显微镜结构揭示了受体结合的先决构象状态。gydF4y2Ba细胞Res。gydF4y2Ba27gydF4y2Ba, 119-129(2017)。gydF4y2Ba
Yuan, Y.等。MERS-CoV和SARS-CoV刺突糖蛋白的Cryo-EM结构揭示了动态受体结合域。gydF4y2BaCommun Nat。gydF4y2Ba8gydF4y2Ba, 15092(2017)。gydF4y2Ba
沃尔斯,a.c.等。SARS-CoV-2刺突糖蛋白的结构、功能和抗原性。gydF4y2Ba细胞gydF4y2Ba181gydF4y2Ba, 281 - 292。e286(2020)。gydF4y2Ba
Wrapp, D.等。预融合构象中2019-nCoV峰值的冷冻电镜结构。gydF4y2Ba科学gydF4y2Ba367gydF4y2Ba, 1260-1263(2020)。gydF4y2Ba
朱姆拉,陈俊峰,艾泽尔,许德生,袁桂云。冠状病毒药物的发现和治疗方案。gydF4y2BaNat. Rev. Drug Disco。gydF4y2Ba15gydF4y2Ba, 327-347(2016)。gydF4y2Ba
泰勒,p.c.等。中和单克隆抗体治疗COVID-19。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba21gydF4y2Ba, 382-393(2021)。gydF4y2Ba
Coughlin, m.m. & Prabhakar, B. S.中和严重急性呼吸综合征冠状病毒的人单克隆抗体:靶点,作用机制和治疗潜力。gydF4y2BaRev. Med Virol。gydF4y2Ba22gydF4y2Ba, 2-17(2012)。gydF4y2Ba
哈维,W. T.等。SARS-CoV-2变体、spike突变和免疫逃逸。gydF4y2BaNat. Rev.微生物gydF4y2Ba19gydF4y2Ba, 409-424(2021)。gydF4y2Ba
Piccoli, L.等。通过结构引导的高分辨率血清学绘制SARS-CoV-2刺突受体结合域上的中和和免疫显性位点。gydF4y2Ba细胞gydF4y2Ba183gydF4y2Ba, 1024 - 1042。e1021(2020)。gydF4y2Ba
郑晓东,钟永凯,陈晓东,陈晓东,陈晓东,陈晓东。新型冠状病毒与新型冠状病毒抗体相互作用的结构基础。gydF4y2BaImmunol趋势。gydF4y2Ba41gydF4y2Ba, 1006-1022(2020)。gydF4y2Ba
王,N.等。MERS-CoV S1-NTD中和敏感表位的结构定义。gydF4y2Ba细胞的代表。gydF4y2Ba28gydF4y2Ba, 3395 - 3405。e3396(2019)。gydF4y2Ba
周,H.等。MERS-CoV刺突糖蛋白n端结构域中和表位的结构定义。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 3068(2019)。gydF4y2Ba
Chi, x等。一种中和人抗体与SARS-CoV-2的Spike蛋白的n端结构域结合。gydF4y2Ba科学gydF4y2Ba369gydF4y2Ba, 650-655(2020)。gydF4y2Ba
Cerutti, G.等人。针对刺突n端结构域的强效SARS-CoV-2中和抗体靶向单个超位点。gydF4y2Ba细胞宿主微生物gydF4y2Ba29gydF4y2Ba, 819-833 e817(2021)。gydF4y2Ba
麦卡勒姆等人。n端结构域抗原图谱揭示了SARS-CoV-2的易感位点。gydF4y2Ba细胞gydF4y2Ba,gydF4y2Bahttps://doi.org/10.1016/j.cell.2021.03.028gydF4y2Ba(2021)。gydF4y2Ba
Suryadevara, N.等。识别SARS-CoV-2刺突蛋白n端结构域的中和性和保护性人单克隆抗体。gydF4y2Ba细胞gydF4y2Ba,gydF4y2Bahttps://doi.org/10.1016/j.cell.2021.03.029gydF4y2Ba(2021)。gydF4y2Ba
王,C.等。人冠状病毒OC43的抗原结构揭示了暴露和闭塞的中和表位。gydF4y2BaCommun Nat。gydF4y2Ba13gydF4y2Ba, 2921(2022)。gydF4y2Ba
班加鲁等人。人乙型冠状病毒刺突蛋白抗体结构图谱。gydF4y2Ba科学。睡觉。gydF4y2Ba8gydF4y2Ba, eabn2911(2022)。gydF4y2Ba
Du, L.等。新型冠状病毒(sars - cov)的刺突蛋白——疫苗和治疗方法开发的靶点。gydF4y2BaNat. Rev.微生物gydF4y2Ba7gydF4y2Ba, 226-236(2009)。gydF4y2Ba
Shi, W.等。疫苗诱导的小鼠抗体WS6通过识别一个螺旋茎脆弱超位点来中和多种乙型冠状病毒。gydF4y2Ba结构gydF4y2Ba,gydF4y2Bahttps://doi.org/10.1016/j.str.2022.06.004gydF4y2Ba(2022)。gydF4y2Ba
Hurlburt, n.k.等人。刺突S2亚基上泛sarbecvirus中和表位的结构定义。gydF4y2BaCommun。医学杂志。gydF4y2Ba5gydF4y2Ba, 342(2022)。gydF4y2Ba
王,C.等。交叉反应性单克隆抗体描绘的冠状病毒突刺蛋白上的一个保守的免疫原性和脆弱位点。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 1715(2021)。gydF4y2Ba
宋,G.等。SARS-CoV-2和地方性冠状病毒感染中血清和记忆b细胞对刺突蛋白的交叉反应。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 2938(2021)。gydF4y2Ba
绍尔,m.m.等人。广泛中和冠状病毒的结构基础。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba28gydF4y2Ba, 478-486(2021)。gydF4y2Ba
平托,D.等。通过茎螺旋特异性人抗体广泛中和贝塔冠状病毒。gydF4y2Ba科学gydF4y2Ba373gydF4y2Ba, 1109-1116(2021)。gydF4y2Ba
利普,K. M.等。靶向S蛋白HR2结构域和HR2上游区域的单克隆抗体可中和体外感染的严重急性呼吸综合征冠状病毒。gydF4y2Baj .性研究。gydF4y2Ba80gydF4y2Ba, 941-950(2006)。gydF4y2Ba
帕勒森,J.等人。合理设计的MERS-CoV预融合刺突抗原的免疫原性和结构。gydF4y2Ba国家科学院学报美国gydF4y2Ba114gydF4y2Ba, e7348-e7357(2017)。gydF4y2Ba
马西辛,S. R.和恩根,J. R.氢交换质谱:它是什么,它能告诉我们什么。gydF4y2Ba肛交。Bioanal。化学。gydF4y2Ba397gydF4y2Ba, 967-972(2010)。gydF4y2Ba
佩尔松,H.等。合成抗体识别抗原不需要CDR-H3多样性。gydF4y2BaJ. Mol.生物学。gydF4y2Ba425gydF4y2Ba, 803-811(2013)。gydF4y2Ba
Tao, Y.等。量身定制的四价抗体在细胞、类器官和小鼠中有效和特异性地激活Wnt/卷曲通路。gydF4y2BaElifegydF4y2Ba8gydF4y2Ba,gydF4y2Bahttps://doi.org/10.7554/eLife.46134gydF4y2Ba(2019)。gydF4y2Ba
盖洛,E.等人。原位抗体噬菌体展示产生整合素alpha11/beta1的最佳抑制剂。gydF4y2Ba马伯gydF4y2Ba12gydF4y2Ba, 1717265(2020)。gydF4y2Ba
刘,S.等。一种合成人抗体通过变构机制拮抗IL-18Rbeta信号。gydF4y2BaJ. Mol.生物学。gydF4y2Ba432gydF4y2Ba, 1169-1182(2020)。gydF4y2Ba
耶格尔,C. L.等。人氨基肽酶N是人冠状病毒229E的受体。gydF4y2Ba自然gydF4y2Ba357gydF4y2Ba, 420-422(1992)。gydF4y2Ba
黄,a.h.m.等。甲型冠状病毒适应和进化中的受体结合环。gydF4y2BaCommun Nat。gydF4y2Ba8gydF4y2Ba, 1735(2017)。gydF4y2Ba
Corti, D. & Lanzavecchia, A.广泛中和抗病毒抗体。gydF4y2Ba免疫。gydF4y2Ba31gydF4y2Ba, 705-742(2013)。gydF4y2Ba
Corti, D., Purcell, L. A., Snell, G.和Veesler, D.用中和单克隆抗体处理COVID-19。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 3086-3108(2021)。gydF4y2Ba
郑,Z.等。SARS-CoV-1 spike S2亚基单克隆抗体与新出现的SARS-CoV-2发生交叉反应。gydF4y2Ba欧元SurveillgydF4y2Ba25gydF4y2Ba,gydF4y2Bahttps://doi.org/10.2807/1560-7917.ES.2020.25.28.2000291gydF4y2Ba(2020)。gydF4y2Ba
Andreano, E.等人。来自COVID-19恢复期患者的超强人源单克隆抗体。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 1821 - 1835。e1816(2021)。gydF4y2Ba
Dejnirattisai, W.等。SARS-CoV-2受体结合结构域抗原解剖。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 2183 - 2200。e2122(2021)。gydF4y2Ba
袁,等。在SARS-CoV-2和SARS-CoV受体结合域中高度保守的神秘表位。gydF4y2Ba科学gydF4y2Ba368gydF4y2Ba, 630-633(2020)。gydF4y2Ba
Zhou, D.等。来自康复患者的抗体中和SARS-CoV-2的结构基础。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba27gydF4y2Ba, 950-958(2020)。gydF4y2Ba
詹,W.等。对SARS-CoV-2抗体的结构研究发现了一种广谱抗体,它通过分解刺突三聚体来中和欧微米变体。gydF4y2Baj .性研究gydF4y2Ba, e0048022,(2022)。gydF4y2Ba
霍,等。通过破坏预融合峰中和SARS-CoV-2。gydF4y2Ba细胞宿主微生物gydF4y2Ba28gydF4y2Ba, 445-454 e446(2020)。gydF4y2Ba
罗贝尔,A. G.等人。抗体介导的SARS-CoV-2刺突糖蛋白的破坏。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 5337(2020)。gydF4y2Ba
吴,N. C.等。SARS-CoV-2和SARS-CoV之间的自然突变决定了交叉反应抗体的中和作用。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba16gydF4y2Ba, e1009089(2020)。gydF4y2Ba
Shi Y.等。基于刺突蛋白的结构和免疫学分析,深入了解甲型冠状病毒疫苗的开发。gydF4y2Baj .性研究。gydF4y2Ba,gydF4y2Bahttps://doi.org/10.1128/JVI.02284-20gydF4y2Ba(2021)。gydF4y2Ba
孙,x等。包括SARS-CoV-2在内的泛冠状病毒反应的人抗体的中和机制。gydF4y2BaNat。MicrobiolgydF4y2Ba7gydF4y2Ba, 1063-1074(2022)。gydF4y2Ba
达康,C.等。广泛中和抗体针对冠状病毒融合肽。gydF4y2Ba科学gydF4y2Ba377gydF4y2Ba, 728-735(2022)。gydF4y2Ba
Low, J. S.等。ace2结合使SARS-CoV-2融合肽能够广泛中和冠状病毒抗体。gydF4y2Ba科学gydF4y2Ba377gydF4y2Ba, 735-742(2022)。gydF4y2Ba
沃德,a.b. &威尔逊,i.a.基于结构的抗原设计和下一代疫苗免疫监测的创新。gydF4y2Ba咕咕叫。当今。Immunol。gydF4y2Ba65gydF4y2Ba, 50-56(2020)。gydF4y2Ba
Birtalan, S.等人。酪氨酸、丝氨酸、甘氨酸和精氨酸对抗体亲和力和特异性的内在贡献。gydF4y2BaJ. Mol.生物学。gydF4y2Ba377gydF4y2Ba, 1518-1528(2008)。gydF4y2Ba
没有表型选择的快速和有效的位点特异性突变。gydF4y2Ba国家科学院学报美国gydF4y2Ba82gydF4y2Ba, 488-492(1985)。gydF4y2Ba
Fellouse, f.a. & Sidhu, s.s. IngydF4y2Ba制作和使用抗体gydF4y2Ba(Howard, g.c. & Kaser, m.r.) (CRC出版社,2006)。gydF4y2Ba
夏,S.等。针对SARS-CoV-2及其变体的泛冠状病毒融合抑制剂的结构和功能基础及其临床前评估。gydF4y2Ba钙信号。其他目标。gydF4y2Ba6克ydF4y2Ba, 288(2021)。gydF4y2Ba
夏,S.等。通过一种高效的泛冠状病毒融合抑制剂抑制SARS-CoV-2(以前是2019-nCoV)感染,该抑制剂针对其具有高介导膜融合能力的刺突蛋白。gydF4y2Ba细胞Res。gydF4y2Ba30.gydF4y2Ba, 343-355(2020)。gydF4y2Ba
孟,B.等。抑制性抗体在鼻病毒和其他肠道病毒中识别治疗脆弱性的独特位点。gydF4y2Ba国家科学院学报美国gydF4y2Ba117gydF4y2Ba, 13499-13508(2020)。gydF4y2Ba
Biasini, M.等人。SWISS-MODEL:利用进化信息对蛋白质三级和四级结构进行建模。gydF4y2Ba核酸测定。gydF4y2Ba42gydF4y2Ba, w252-w258(2014)。gydF4y2Ba
彼得森,E. F.等人。UCSF chimera -用于探索性研究和分析的可视化系统。gydF4y2Baj .第一版。化学。gydF4y2Ba25gydF4y2Ba, 1605-1612(2004)。gydF4y2Ba
Schneidman-Duhovny, D., Inbar, Y., Nussinov, R. & Wolfson, H. J. PatchDock和SymmDock:刚性和对称对接的服务器。gydF4y2Ba核酸测定。gydF4y2Ba33gydF4y2Ba, w363-w367(2005)。gydF4y2Ba
Mashiach, E., Schneidman-Duhovny, D., Andrusier, N., Nussinov, R. & Wolfson, H. J. FireDock:用于分子对接中快速交互优化的web服务器。gydF4y2Ba核酸测定。gydF4y2Ba36gydF4y2Ba, w229-w232(2008)。gydF4y2Ba
Otwinowski, Z. & Minor, W.振荡模式下x射线衍射数据的处理。gydF4y2BaEnzymol方法。gydF4y2Ba276gydF4y2Ba, 307-326(1997)。gydF4y2Ba
夏冬,夏国强2:大分子晶体学数据还原专家系统。gydF4y2Baj:。Crystallogr。gydF4y2Ba43gydF4y2Ba, 186-190(2009)。gydF4y2Ba
用相位器分子置换法求解蛋白质复合物结构。gydF4y2BaActa Crystallogr。D:杂志。Crystallogr。gydF4y2Ba63gydF4y2Ba, 32-41(2007)。gydF4y2Ba
埃姆斯利,P. &考坦,K.库特:分子图形模型构建工具。gydF4y2Ba结晶学学报D:生物学。Crystallogr。gydF4y2Ba60gydF4y2Ba, 2126-2132(2004)。gydF4y2Ba
亚当斯,P. D.等。PHENIX:一个全面的基于python的大分子结构解决方案系统。gydF4y2BaActa Crystallogr。D:杂志。Crystallogr。gydF4y2Ba66gydF4y2Ba, 213-221(2010)。gydF4y2Ba
陈,v.b.等。MolProbity:大分子结晶学的全原子结构验证。gydF4y2Ba结晶学学报D:生物学。Crystallogr。gydF4y2Ba66gydF4y2Ba, 12-21(2010)。gydF4y2Ba
克里斯奈尔,E. &亨利克,K.从晶体状态推断大分子组装。gydF4y2BaJ. Mol.生物学。gydF4y2Ba372gydF4y2Ba, 774-797(2007)。gydF4y2Ba
Zivanov, J.等人。在RELION-3中自动高分辨率冷冻- em结构测定的新工具。gydF4y2BaElifegydF4y2Ba7gydF4y2Ba,gydF4y2Bahttps://doi.org/10.7554/eLife.42166gydF4y2Ba(2018)。gydF4y2Ba
唐,G.等。EMAN2:用于电子显微镜的可扩展图像处理套件。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba157gydF4y2Ba, 38-46(2007)。gydF4y2Ba
郑世强等。MotionCor2:改进冷冻电子显微镜光束诱导运动的各向异性校正。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 331-332(2017)。gydF4y2Ba
Rohou, A. & Grigorieff, N. CTFFIND4:快速准确的电子显微图像离焦估计。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba192gydF4y2Ba, 216-221(2015)。gydF4y2Ba
Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A. cryoSPARC:快速无监督冷冻em结构确定的算法。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 290-296(2017)。gydF4y2Ba
西弗斯,F.等人。使用Clustal Omega快速,可扩展生成高质量的蛋白质多序列比对。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba7gydF4y2Ba, 539(2011)。gydF4y2Ba
克鲁克斯,G. E, Hon, G., Chandonia, J. M. & Brenner, S. E. WebLogo:一个序列logo生成器。gydF4y2Ba基因组Res。gydF4y2Ba14gydF4y2Ba, 1188-1190(2004)。gydF4y2Ba
Berezin, C.等。蛋白质序列中功能和结构上重要残基的鉴定。gydF4y2Ba生物信息学gydF4y2Ba20.gydF4y2Ba, 1322-1324(2004)。gydF4y2Ba
确认gydF4y2Ba
本工作得到科技部资助项目2018ZX10731-101-001-010,国家自然科学基金资助项目32070170,国家自然科学基金资助项目82041036。这项工作也得到了上海市教育委员会(SMEC)对上海科技大学生物大分子和精准医学前沿科学中心的部分资助。作者感谢上海科技大学上海高级免疫化学研究所的发现技术平台、分析化学平台和低温电镜设备,为SPR、HDX-MS和EM实验提供仪器和技术支持。作者还感谢上海国家蛋白质科学中心(NCPSS) BL19U1光束线工作人员在x射线数据收集过程中的帮助。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
b.y., s.m., l.l.和D.S.设计并监督实验和数据分析。J.C.X.表达和纯化所有相关蛋白,进行SPR, HDX-MS,生物信息学和结构研究和分析。J.S.用j.c.x S.M.提供的蛋白质进行噬菌体筛选,并进行亲和成熟实验。Q.S.L.在s.w.w.z.的帮助下进行了伪病毒感染实验,Y.Z.帮助J.C.X.制备了IgG格式的抗体,并参与了HDX-MS实验。Y.W.X.和J.N.分别帮助J.C.X.收集电子显微镜数据和x射线数据。Q.L.Q.准备细胞。b.y.、s.m.、l.l.和D.S.根据其他作者的意见撰写了这篇论文。B.Y.构想并管理了这个项目。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
通信生物学gydF4y2Ba感谢匿名审稿人对本工作的同行评议所作的贡献。主要处理编辑:邱智娟。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
补充信息gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
向,J,苏,J,兰,Q。gydF4y2Baet al。gydF4y2Ba抗原定位揭示了α-HCoV刺突蛋白的易感位点。gydF4y2BaCommun杂志gydF4y2Ba5gydF4y2Ba, 1179(2022)。https://doi.org/10.1038/s42003-022-04160-8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s42003-022-04160-8gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba